作者简介: 董建江, 1998年生, 中国海洋大学物理与光电工程学院硕士研究生 e-mail: abc18366531189@163.com
旨在实现对海洋牧场水下底栖动物的原位识别, 使用随机森林算法实现识别分类检测, 对目标生物进行分类识别分析, 深入挖掘数据, 提高工作效率和决策可靠性。 利用研发的水下高光谱成像分析仪, 在不同的水下环境中通过获取五种海洋牧场常见经济动物(虾夷扇贝、 栉孔扇贝、 脉红螺、 皱纹盘鲍、 仿刺参)的高光谱数据, 归一化处理后运用机器学习中的随机森林(RF)、 基于主成分分析的随机森林(PCA-RF)、 基于递归特征消除的随机森林(RFE-RF)三种随机森林算法对五种底栖动物进行分类识别以及对比分析。 通过RF的变量重要性排序, 筛选出排名较高, 对模型贡献度高的波段所对应的反射谱强度数据, 再将排名靠前的特征波段数据输入分类器中, 通过优化参数, 得到分类准确度。 将数据的分类结果输出混淆矩阵, 可以看到五种样品的识别情况。 脉红螺样品识别精度最低, 为64%; 仿刺参与栉孔扇贝的识别精度最高, 达到了100%; 虾夷扇贝与皱纹盘鲍的识别精度分别为91%与96%。 三种方法最终得到的分类精度分别为: RF 90.13%; PCA-RF 95.20%; RFE-RF 98.74%, 达到了较为理想的分类效果, 体现了随机森林算法运用在水下高光谱数据分类研究的可行性。
This study aims to identify underwater benthic animals in situ, use random forest algorithm to achieve recognition classification detection, classify and identify target organisms for analysis, dig deeper into the data, and improve efficiency and reliability of decision making. The hyperspectral data of five common economic animals (scallop, ctenophore, veined red snail, wrinkled disc abalone, and imitation spiny ginseng) in different underwater environments were acquired, normalized and processed using random forest (Random Forest, RF) in machine learning, random forest based on principal component analysis method (Principal Component Analysis-Random Forest, PCA-RF), and random forest based on recursive feature elimination method (Recursive feature elimination- Random Forest, RFE-RF). Three random forest algorithms were used to classify five benthic species and for comparative analysis. By ranking the importance of the variables of RF, the reflection spectrum intensity data corresponding to the bands with higher ranking and higher contribution to the model were filtered. Then the top-ranked feature band data were input into the classifier, and the classification accuracy was obtained by optimizing the parameters. The classification results of the data were output to the confusion matrix, and the identification of the five samples could be seen. The lowest recognition accuracy of 64% was obtained for the veined red snail sample; the highest recognition accuracy of 100% was obtained for imitation spiny ginseng and ctenophore scallops; the recognition accuracies of 91% and 96% were obtained for the scallop and wrinkled disc abalone, respectively. The final classification accuracies of the three methods were 90.13% for RF, 95.20% for PCA-RF, and 98.74% for RFE-RF, which showed the feasibility of using the random forest algorithm in the classification of underwater hyperspectral data.
近年来近海生态系统受到全球变化和人类活动的多重影响, 出现生境退化、 资源衰退、 生物多样性降低等问题。 海洋牧场建设是实现生境恢复和资源增殖的重要手段, 是渔业产业结构转型升级的重要抓手。 海洋牧场的生物丰度统计等工作, 以往通过人工解决, 效率低、 周期长、 成本大且准确率不高。 目前, 已经有不少海洋牧场企业利用水下摄像系统开始视频数据的收集和利用, 实时信息量巨大, 难以利用人工方法进行生物群落数据的提取, 图像分析技术与机器学习等在海洋牧场生物资源监测研究中显得尤为重要[1]。 在水产养殖中应用人工智能、 机器视觉技术及其他传感器技术, 可以实时的监测生态环境等, 并结合深度学习、 随机森林(random forest, RF)等算法实现机器视觉的识别分类检测, 对海洋生物进行分类识别统一分析, 深入挖掘养殖过程数据, 提高工作效率和决策可靠性。
水下目标探测所使用传统的红-绿-蓝(RGB)相机取得图像的技术越来越成熟。 传统的图像处理方法[2, 3]和基于机器学习、 深度学习的目标检测算法, 如基于区域的快速卷积神经网络(faster R-CNN)[3], You Only Look Once(YOLO)[4]等已广泛应用于水下目标检测。 在理想的水下成像环境中, 检测速度和检测结果均优于传统方法, 各种算法的精度都能达到较为理想的水平。 然而, 传统的RGB图像检测技术存在一系列问题。 当水下成像环境较差且海洋动物具有保护色彩机制时, 很难从复杂的背景中有效地检测和识别实验目标[5, 6]。
高光谱成像技术可以提供比RGB图像更高的光谱分辨率, 可从紫外、 可见光、 近红外到中红外波段, 提供丰富的光谱信息。 高光谱数据一般由数百个相邻的窄光谱波段获取, 可以解决传统RGB图像检测技术所遇到的问题, 也使其具有较好的目标识别能力和相似目标识别能力。 经典的高光谱目标检测算法包括由Reed和Yu开发的异常检测器RXD算法[7]、 核RXD (KRXD)算法[8]、 正交子空间投影(OSP)算法[9]和约束能量最小化(CEM)算法[10]。 Mohite等[11]使用高光谱数据检测葡萄上的农药残留, 比较了XGBoost、 RF、 SVM和人工神经网络(ANN)四种分类器。 此外, 研究了LASSO和Elastic Net特征选择的效用。 结果表明, 当同时使用LASSO和Elastic Net选择的波段时, RF获得了最准确的分类模型。 目前, 文献中关于高光谱水下目标检测与分类的研究较少。
随机森林(RF)算法已成功应用于一系列高维数据分类研究, 其中包括高光谱数据分析[12]。 RF是一个bagging(即bootstrap聚合)集成过程, 其中分类树是从训练数据中获得的随机样本中生长出来的[13]。 RF使用套袋和随机变量选择在集合中构建决策树[14]。 作为集成分类器, RF算法拥有几个优点: (1)该算法结合了特征之间的交互作用; (2)在计算上比装袋或增压更有效; (3)不容易出现过拟合现象; (4)提供了可变强度估计和内部误差估计[13]。
本研究的目的是: (1)利用水下高光谱数据和三种RF算法建立模型, 在水下环境中对五种海洋牧场常见的底栖动物进行分类识别; (2)评估RF、 主成分分析的随机森林(principal component analysis-random forest, PCA-RF)和递归特征消除的随机森林(recursive feature elimination-random forest, RFE-RF)三种算法在水下高光谱数据分类分析中的效用, 选择可能产生最佳分类精度的波段子集。 (3)比较RF、 PCA-RF与RFE-RF算法的分类性能, 测试不同特征选择算法选择的波段组合是否能够提高最终的分类精度。
所使用的水下目标物的高光谱数据由中国科学院海洋研究所研发的水下推扫式高光谱成像仪获得, 采用标准卤素灯(400~1 000 nm)作为水下主动照明光源, 分光模组(Imspector V10, 江苏双利合谱科技有限公司, 中国)入射狭缝为30 μ m, CCD(ICX, SONY, 日本)像素数为1 392× 1 040, 扫描视场角为22° , 通道数分1 440、 720、 360、 176四档, 光谱分辨率和空间分辨率由通道数决定。 整机兼容静态定点扫描和动态巡航扫描, 可坐底或者搭载无人艇或者水下机器人进行工作。
选用5种海洋牧场常见的经济动物(虾夷扇贝、 栉孔扇贝、 脉红螺、 皱纹盘鲍、 仿刺参)作为目标物, 使用研发的水下推扫式高光谱成像仪, 选用静态定点式扫描, 获取以上5种目标物的高光谱数据。
实验数据由收集的7张高光谱图像提取。 经过预处理, 得到360个反射谱波段。 在此提供了5张图片的真实情况如图1所示。 为了获得、 训练和评价分类模型, 70%的光谱数据用于分类算法训练集, 训练集数据来自图1的高光谱图像的反射谱数据; 30%的光谱数据作为测试集用于分类模型评估, 测试集数据来自额外的2张高光谱图像的反射谱数据。
所选择的高光谱图像的目标样品有五种, 分别为虾夷扇贝、 栉孔扇贝、 脉红螺、 皱纹盘鲍、 仿刺参。 在将他们进行分类识别之前, 每个目标都打上相应的数字与颜色标签。 每种样品在其表面随机点与随机区域全覆盖共提取35条光谱, 每张高光谱图像提取175条光谱, 五张高光谱图像总共提取875条光谱构建为训练集。 测试集数据则是用另外两张拍摄位置不同但其他实验条件相同的高光谱图像提取, 总共提取375条光谱。
五种底栖动物样品的数字标签分别为: (1)仿刺参、 (2)虾夷扇贝、 (3)脉红螺、 (4)栉孔扇贝、 (5)皱纹盘鲍。 通过对五种样品进行数字标签标识, 可以在随机森林算法分类过程中用数字标签输出分类结果, 提高目标识别便利性。
本研究的重点是建立对水下目标底栖动物的高光谱数据的随机森林分类模型。 实验采集了363.87~1 047.92 nm光谱范围内的数据。 所收集的光谱曲线横坐标为波长, 纵坐标为反射谱强度, 将收集到的光谱进行归一化处理, 图2为五样品的归一化光谱曲线, 光谱曲线颜色与样品的颜色标签相对应, 分别为: 仿刺参(红色)、 虾夷扇贝(绿色)、 脉红螺(蓝色)、 栉孔扇贝(黄色)、 皱纹盘鲍(青色)。 由于水体对光的吸收作用, 选取了3 63.87与830.00 nm波长区间内的光谱。
1.2.1 随机森林(RF)
RF方法流程图如图3所示。
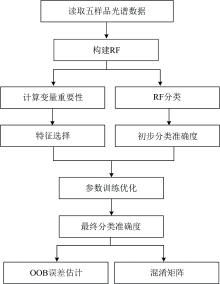 | 图3 基于RF算法的高光谱数据分类方法流程图Fig.3 Flowchart of hyperspectral data classification method based on RF algorithm |
RF算法是一个决策树的集合。 由于其简单、 精度好, 已在多个高光谱分类工作中使用[13]。 决策树是输入数据的一种递归分割方法[15]。 从根节点(树的第一级)到叶节点进行分割, 减少每次分割时的熵。 叶节点是树的最后一层也是熵最小的地方。 其目的是在叶子中只保留同一类的样品。 从根节点到叶节点的路径上有几个分离节点。 这些包括基于可用特征和应用于所选特征的阈值的决策规则。
尽管决策树非常快速和简单, 但它们对噪声非常敏感, 经常过拟合训练样本。 正因为如此, 决策树可以被归类为弱学习者。 为了克服这些缺点, 将决策树集成到一个强学习器中。 在RF算法中, 森林中的树必须是不相关的, 每棵树都是唯一的, 因此应用了随机子空间(feature bagging)和bootstrap aggregating (bagging)技术。
Breiman在2001年提出了Bootstrap aggregating算法, 它包括对训练数据集样本子集的随机选择和替换[15]。 Ho在1995年提出随机子空间由每个节点的所有输入特征中随机选择的特征子集组成, 并从所选择的新特征子集中考虑在下一层产生较小熵的分割节点的特征[16]。
如前所述, 这两种技术在不增加趋势的情况下使模型方差最小化。 因此, 当单个决策树对噪声敏感时, 对于树的集合的平均预测不敏感, 前提是这些树是不相关的。 在完成森林后, 每棵树都为一个类投了一票, 标签由多数投票来定义。 该算法的主要优点是能很好地处理噪声、 可调参数少、 计算代价低。
对于这项工作, RF算法使用了python软件中的scikit-learn包的RandomForestClassifier库实现。 树的数量设置为500, 深度设置为8。 这些值是在一个调优过程之后定义的。 使用RF进行5倍交叉验证模型参数训练, 通过这个过程输出最优参数, 超过最优参数后整体精度也不会再显著提高, 因此设定了这些参数值。
在RF训练过程中, 三分之一不用于种树的样本(“ out of bag” , 或称为OOB)用于计算: (1)OOB误差, 它提供了分类性能的内部度量; (2)变量重要性, 这是基于准确性的平均下降或基尼指数[14]。
RF的一个有用的副产物是变量重要性, 它可用于特征排序。 因此, RF的变量重要性揭示了相关波段对模型分类的贡献程度[15]。 本研究中使用基尼系数重要性或排列重要性作为波段重要性的度量, 以此选择特征波段。 基尼系数重要性是用森林中树木数量归一化的分裂变量的基尼系数杂质减少量的总和计算。
利用OOB观测值计算分类精度平均下降时的变量重要性。 它是通过测量当OOB观测数据与原始观测数据随机排列时预测精度的变化来计算的。 然后对所有树的预测精度差取平均值, 计算排列重要性值[17]。
1.2.2 基于主成分分析的随机森林(PCA-RF)
PCA-RF算法流程如图4所示。
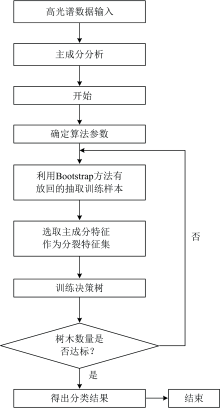 | 图4 基于PCA-RF算法的高光谱数据分类流程图Fig.4 Flow chart of hyperspectral data classification based on PCA-RF algorithm |
主成分分析(PCA)是常用的特征降维方法, 其思想是求解一个正交线性变换, 使原始数据可以用少数几个正交方向线性表示, 同时使得原始数据的信息量损失最小。 然而, PCA变换使原始数据的物理意义发生了改变, 而且, 当波段间的相关性很弱时, PCA方法的效率会大大下降。 本工作使用PCA对高光谱数据进行降维, 提取隐藏在数据中的主成分, 压缩特征空间, 再对主成分数据进行光谱特征提取, 根据主成分数对模型的特征贡献度分析, 算法最终采用6个主成分分量进行特征提取。 然后将PCA降维后的结果输入RF分类器, 得到分类准确度。 利用python软件中的sklearn.decomposition包的PCA库实现数据的降维。
1.2.3 基于递归特征消除的随机森林(RFE-RF)
图5为RFE-RF算法的流程图。
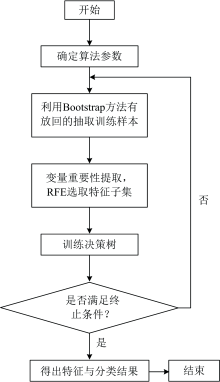 | 图5 基于RFE-RF算法的高光谱数据特征选择分类方法流程图Fig.5 Flowchart of feature selection classification method for hyperspectral data based on RFE-RF algorithm |
Dıáz-Uriarte和Alvarez de Andre’ s[18]在工作中引入了递归特征消除(RFE)算法。 通过迭代建立多个随机森林模型, 并在每次迭代中依次剔除变量重要性排名最低的波段, 得到最优波段子集, 这是由RF置换重要性评估确定的。 通过拟合所有RF模型得到的最佳模型的m个标准误差范围内的最小OOB错误率来定义最佳波段子集。 利用python软件中的sklearn.feature_selection包的RFECV库实现RFE算法。
对于三种算法, 70%的光谱数据作为训练数据集, 30%的光谱数据则是作为独立的测试数据集, 以提供模型精度的独立估计。 分类精度首先使用RF中的OOB误差估计和混淆矩阵来评估, OOB误差估计公式可参考Simone Vincenzi团队的工作[19]。
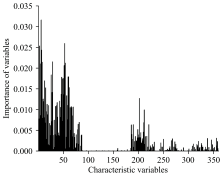
实验数据共360个波段, 从RF算法运算得到的基于变量重要性的波段选择结果来看(如图6), 准确度排名最高的前二十个波段组合为: 367.48、 371.08、 471.22、 365.67、 412.75、 374.70、 372.89、 458.38、 445.57、 451.06、 456.55、 454.72、 378.31、 438.26、 380.12、 407.30、 469.39、 449.23、 443.74和473.06 nm, 该顺序按照变量重要性由高到低排列。
通过RF的变量重要性排序, 筛选出排名较高, 对模型贡献度高的最佳波段数所对应的反射谱强度数据, 再将RF运算结果中排名靠前的最佳特征波段数据输入分类器中, 通过优化参数, 得到分类准确度。 通过对训练集与测试集的划分训练, 以及OOB误差估计(图7), 优化后的n_estimators值为400; max_depth值为8。
 | 图7 五种样品的OOB误差估计 n_estimators为400时, OOB误差为0.004 6Fig.7 OOB error estimation for five samples OOB error is 0.004 6 when n_estimators=400 |
将数据的分类结果输出混淆矩阵(图8), 可以看到五种样品的识别情况。 第三种(脉红螺)样品识别精度最低, 为64%; 第一种(仿刺参)与第四种(栉孔扇贝)的识别精度最高, 为100%; 第二种(虾夷扇贝)与第五种(皱纹盘鲍)的识别精度分别为91%与96%, 总体分类精度较高。 因为仿刺参与栉孔扇贝的反射谱特征与其他四种样品有明显差异, 所以两种样品更容易被识别, 分类精度最高。 实验所提取的脉红螺反射谱光谱曲线趋势与光谱特征与其他样品在某些波段部分相似, 测试集中的75个脉红螺样品有20个被错误识别, 因此识别精度最低。 通过识别结果可知, 脉红螺有36%的几率被识别为皱纹盘鲍。 RF分类精度为90.13%, 在n_estimators为400情况下OOB误差为0.004 6, kappa系数为0.876 7, 模型稳定度优异。
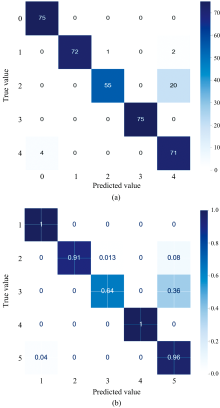 | 图8 (a)根据测试集样品数量输出混淆矩阵; (b)根据样品识别精度输出混淆矩阵Fig.8 (a) Output confusion matrix based on the number of samples in the test set; (b) Output confusion matrix based on sample identification accuracy |
主成分分析可以将数据从N维降低到M维, 此时需要找到M个向量用于投影原始数据, 使投影误差(投影距离)最小。 因此, 可以对原始数据进行主成分分析, 这样就可以使用具有较少维度且不相关的数据来取代原始的高维数据, 然后用变换后的数据进行建模。 对经归一化处理的光谱数据进行主成分分析降维, 得到帕累托图(Pareto chart)。 通过帕累托图可以推断, 当保留6个主成分时, 特征贡献率达到了99.92%, 因此计算中采用前6个主成分。 图9给出了数据的PCA散点图分布。 将最佳解释度选出的主成分数输入PCA* RF分类器中, 得到降维后的五种样品的光谱分类精度。 PCA-RF分类精度为95.20%, Explained variance(解释度)0.999 2, kappa系数0.843 3, 模型稳定度优异。
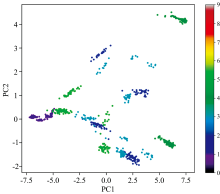 | 图9 五种样品高光谱数据的PCA主成分分布散点图, 主成分数设定为6Fig.9 Scatter plot of PCA principal component distribution for hyperspectral data of five samples with the first 6 principal components |
图10为五种样品的高光谱数据在RFE-RF模型运行过程中的RFECV精度随筛选特征数的变化。 由RFE算法选择了最佳波段子集, 用于五种样品的分类, 如表1, 提供了有关所选波段的细节。 RFE选择的波段范围为400~1 000 nm。
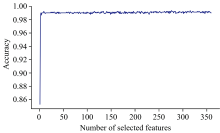 | 图10 五种样品的高光谱数据在RFE-RF模型中RFECV精度随筛选特征数的变化Fig.10 Variation of RFECV accuracy with the number of screening features for RFE-RF model based on hyperspectral data of five samples |
| 表1 五种样品的高光谱数据通过RFE所筛选的特征波段 Table 1 Characteristic bands filtered by RFE for the hyperspectral data of the five samples |
采用RF算法和所有波段进行底栖动物的分类, 优化的n_estimators和max_depth值也用于RFE。 从表1可以看出, 使用RFE算法对五种样品进行分类得到了最佳的总体分类结果。 RFE总共选择了83个波段。 总体而言, RFE显著降低了数据维度。 与使用RF和所有波段相比, RFE-RF提供了更好的分类结果(分类精度为98.74%, kappa系数0.876 6)。
RFE算法已经在一些高光谱研究中使用[20], 实现了显著的维数下降, 同时具有高分类性能。 在本研究中, RFE实现了波段子集的显著减少。 此外, 在本研究的实验模型下, RFE-RF算法的准确度优于RF与PCA-RF算法, 产生了更小的子集和更好的分类精度。
本研究评估了高光谱数据在识别水下底栖动物方面的效用。 更具体地说, 我们评估了RF的三种使用方法, 以获得一个最佳主成分数或最佳波段子集, 可用于区分不同水下样品。 RF包装框架通过识别最优波段子集显著降低了原始数据集的维数, 从而简化了建模过程, 最终提高了分类性能。 本研究的总体结果表明, 使用的三种RF算法令维数显著下降, PCA-RF与RFE-RF都提高了分类精度, 如表2。
| 表2 三种方法分类识别精度对比 Table 2 Comparison of classification recognition accuracies of the three methods |
旨在实现对海洋牧场水下底栖动物的原位识别, 同时评估RF、 PCA-RF、 RFE-RF特征选择算法在高光谱数据分析中的效用。 运用高光谱手段, 收集五种底栖动物样品的高光谱图像并提取样品反射光谱, 数据经过光谱归一化预处理后用三种不同特征选择方式的随机森林算法进行分类, 得到的分类精度分别为: RF 90.13%; PCA-RF 95.20%; RFE-RF 98.74%。 结果表明, 在本研究的水下分类模型中, RFE-RF算法的分类精度优于RF与PCA-RF算法。 此外, 与使用RF的所有波段相比, RFE-RF显著降低了维数, 并提高了分类精度。 RFE-RF模型体现了随机森林运用在水下高光谱数据分类研究的可行性。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|