作者简介: 肖仕杰, 1993年生, 华中农业大学工学院硕士研究生 e-mail: 1175760869@qq.com
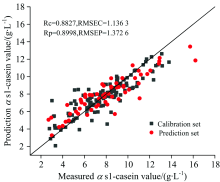
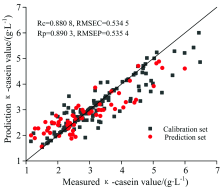
为了找到一种能够对牛乳中的两种主要过敏原(αs1和κ-酪蛋白)含量快速检测的方法, 以河南、 湖北、 宁夏和内蒙古四省区的211份中国荷斯坦牛牛乳样本为研究对象, 建立了基于傅里叶变换中红外光谱技术的牛乳中αs1和κ-酪蛋白含量的无损快速检测模型。 首先对牛乳的原始光谱进行预分析, 发现水对牛乳的光谱吸收具有很强的干扰, 对水的两个主要吸收区域1 597~1 712和3 024~3 680 cm-1进行分析, 发现水的吸收区域1 597~1 712 cm-1和蛋白的部分吸收区域1 558~1 705 cm-1(酰胺Ⅰ)基本重合, 通过对比去除1 597~1 712 cm-1前后的效果, 最终选择925.92~3 005.382 cm-1的光谱区域作为敏感波段用于后续分析。 选取的全光谱经手动降维, 利用MCCV剔除异常样本, 分别采用标准正态变量变换(SNV)、 多元散射校正(MSC)等8种预处理算法和竞争性自适应重加权算法(CARS)、 无信息变量消除法(UVE)等3种特征选择算法联合建立支持向量机回归模型(SVR)。 经检验, 对于αs1-酪蛋白, 一阶导数和CARS算法结合建立的SVR模型效果最优, 训练集相关系数Rc和测试集相关系数 Rp分别为0.882 7和0.899 8, 训练集均方根误差RMSEC和测试集均方根误差RMSEP分别为1.136 3和1.372 6; 对于κ-酪蛋白, 一阶差分和UVE算法结合建立的SVR模型效果最优, 训练集相关系数 Rc和测试集相关系数 Rp分别为0.880 8和0.890 3, 训练集均方根误差RMSEC和测试集均方根误差RMSEP分别为0.534 5和0.535 4。 研究结果表明, 基于傅里叶变换中红外光谱技术建立的SVR模型可以对牛乳中的过敏原αs1和κ-酪蛋白含量进行无损检测, 预测效果良好, 此研究弥补了国内利用光谱技术对牛乳中的酪蛋白进行无损快速检测的空白。
In order to find a rapid detection method for the content of two main allergens (αs1 and κ-casein) in milk, 211 Chinese Holstein milk samples from four provinces of Henan, Hubei, Ningxia and Inner Mongolia were selected as the research objects, and a non-destructive and rapid detection model of αs1 and κ-casein in milk was established based on Fourier transform mid infrared spectroscopy. Firstly, the original spectrum of milk was pre-analyzed, and it was found that strongly influenced the spectral absorption of milk. The two main absorption regions of water (1 597~1 712 and 3 024~3 680 cm-1) were analyzed. It was found that the absorption region of water (1 597~1 712 cm-1) overlapped with that of protein (1 558~1 705 cm-1)(amide Ⅰ). By comparing the effect of removing 1 597~1 712 cm-1, the spectral region of 925.92~3 005.382 cm-1 was selected as the sensitive band for subsequent analysis. The dimension of the selected full spectrum was reduced manually, and MCCV eliminated the abnormal samples. The support vector machine regression model (SVR) was established by using eight preprocessing algorithms, such as Savitzky-Golay convolution smoothing (S-G), standard normal variable (SNV). Meanwhile, three feature selection algorithms were combined, such as competitive adaptive reweighting algorithm (CARS) and information-free variable elimination algorithm (UVE). The results showed that for αs1- casein, the SVR model established by the combination of the first derivative and CARS algorithm was the best, the training set correlation coefficient ( RC) and test set correlation coefficient ( RP) were 0.882 7 and 0.899 8, respectively, and the training set root mean square error (RMSEC) and test set root mean square error (RMSEP) were 1.136 3 and 1.372 6, respectively. For κ-casein, the SVR model established by the combination of second-order difference and UVE algorithm was the best. The training set correlation coefficient ( RC) and test set correlation coefficient ( RP) were 0.914 7 and 0.887 7, respectively, and the training set root mean square error (RMSEC) and test set root mean square error (RMSEP) were 0.473 5 and 0.558 1, respectively. The results showed that the SVR model based on Fourier transform mid-infrared spectroscopy can be used to detect the content of allergens αs1 and κ-casein in milk, and the prediction effect was good. This study can make up for the blank of rapid and non-destructive detection of casein in milk by spectral technology in China.
牛乳中含有丰富的蛋白质, 对人体的生长发育起着重要作用, 尤其对于婴幼儿来说是不可或缺的优质蛋白质来源, 但同时, 牛乳也是一种过敏原。 FAO/WHO已经将牛奶和乳制品确定为引发人类食物过敏现象的8种主要食物之一[1], 相关数据表明牛乳过敏患病率在婴儿中高达2%~7.5%[2], 随着乳制品销量的增加, 牛乳过敏率不断上涨已变为不可忽略的食品安全问题。 对牛乳过敏, 实际上是对牛乳中的蛋白质敏感, 乳蛋白的两个主要类别分别是乳清蛋白和酪蛋白, 其中酪蛋白的含量约占总蛋白质含量的80%, 约65%的牛乳过敏人员对酪蛋白过敏。 其中, α s1和κ -酪蛋白为主要的过敏原[3]。 牛乳过敏目前无法根治, 只能避免饮用牛奶或食用乳制品。 牛乳蛋白过敏病人通常在消化系统和皮肤两个方面有明显的症状表现, 如呕吐、 腹泻、 腹痛和湿疹、 荨麻疹等[4], 因此很多国家都制定了食品过敏原强制标识条例来保障大众健康[3], 我国制定的GB 7718— 2011《预包装食品标签通则》[5]和GB/T 23779— 2009《预包装食品中的致敏原成分》[6]建议商家标明可能的致敏物。
如果能够可靠地检测出牛乳中α s1和κ -酪蛋白的含量, 就能为牛乳敏感人员提供饮用参考指示。 乳成分的主要检测方法有气相色谱法、 色谱-质谱联用法和高效液相色谱法等, 这些方法灵敏度高、 可靠性好, 但成本高、 技术难度大、 分析时间长, 因此找到一种简单高效的替代方法非常重要。 红外光谱法具有快速无损、 简单易行的优点, 相比于近红外光谱, 中红外光谱的波段范围更广、 包含的信息量更丰富, 在国外被广泛应用于牛乳中各营养成分如蛋白成分的检测。 Etzion等[7]表明中红外光谱法可以预测乳蛋白的含量, Bonfatti等[8]基于中红外光谱对牛乳中的酪蛋白等的含量进行了预测, Niero等[9]表明UVE算法可以提高中红外光谱对乳蛋白组分含量的预测精度, McDermott等[10]基于中红外光谱预测了牛乳中的蛋白质和氨基酸含量。 但在国内, 利用光谱技术检测牛乳中蛋白成分的研究鲜有报道。
为此, 本文利用傅里叶变换中红外光谱技术对牛乳中α s1和κ -酪蛋白两种过敏原进行分析, 利用竞争性自适应重加权算法(competitive adaptive reweighed sampling, CARS)、 无信息变量消除法(uninformative variables elimination, UVE)和连续投影算法(successive projections algorithm, SPA)筛选出能代表酪蛋白含量的特征变量, 并利用支持向量机(support vector regression, SVR)模型分别构建了α s1-酪蛋白含量和κ -酪蛋白含量的无损检测模型, 模型的预测精度优于Bonfatti等[8]、 Niero等[9]和McDermott等[10]前人研究结果。
试验材料来源于河南、 湖北、 宁夏和内蒙古四省区的211头中国荷斯坦牛, 一头牛采集一份牛乳, 牛乳采集利用自动挤奶装置完成, 先用消过毒的毛巾擦拭牛乳房, 然后用碘甘油混合溶液再次消毒, 挤出前三把乳汁后进行牛乳采集, 每份牛乳采集40 mL, 分装到直径3.5 cm, 高9 cm的圆柱形全新采样瓶里, 依次编号, 并向每个采样瓶里立即加入溴硝丙二醇防腐剂, 缓慢摇晃使其充分溶解, 运回途中在牛乳样品周围放置冰袋防止变质, 到达实验室后立即放入冰箱保存(4 ℃), 并于第二天进行光谱采集。
MilkoScanTM FT+[傅里叶变换中红外光谱仪(FTIR), 丹麦FOSS公司]; 电热恒温水浴锅; 十万分之一电子天平; Waters e2695液相色谱仪。
α s-酪蛋白(lot C-6780, 纯度≥ 70%)、 κ -酪蛋白(lot C-0406, 纯度≥ 80%)标准品(Sigma 公司); 乙腈(色谱级, 纯度≥ 99.8%)、 盐酸胍、 三氟乙酸(TFA)(上海生工公司); 其他试剂均为国产分析纯。
1.3.1 中红外光谱的采集
利用MilkoScanTM FT+进行光谱采集, 具体采集步骤: 将牛乳分批放入45 ℃电热恒温水浴锅内预热5 min, 预热好的牛乳放在检测架上上下摇晃数次使牛乳混合均匀, 将检测架放在检测履带上, 打开瓶盖, 依次进行检测, 采集完光谱后的牛乳置于-20 ℃冷冻保存。
1.3.2 α s1-酪蛋白和κ -酪蛋白的含量测定
(1)标准样品的处理
先用去离子水将混合标样溶解, 直到浓度约为10 g· L-1左右, 然后往1 600 μ L处理液(6 mol· L-1盐酸胍溶液, 内含 0.1 mmol· L-1 EDTA-Na2, pH 6.0)中滴加400 μ L配好的标样溶液, 于室温下孵育90 min, 上机前用0.22 μ m尼龙滤膜过滤。
(2)牛乳的处理
取80 μ L牛乳于320 μ L处理液中, 室温孵育90 min, 将离心机转速调为14 000 r· min-1, 5 min后取上清液。 上机前用0.22 μ m尼龙滤膜过滤。
(3)RP-HPLC的色谱条件
色谱柱: ZORBAX 300SB-C18; 进样量: 50 μ L; 柱温: 40 ℃; 流速: 1 mL· min-1; 洗脱时间: 42 min; 检测波长: 214 nm。
流动相A: 10%乙腈+90%去离子水+0.1%TFA; B: 90%乙腈+10%去离子水+0.1% TFA。 流动相B梯度(变化率)如下: 从33%到38%洗脱10 min(0.50%B· min-1), 从38%到40%洗脱6 min(约0.33%B· min-1), 保持40%洗脱6 min(0.00%B· min-1), 从40%到40.5%洗脱2 min(0.25%B· min-1), 保持40.5%洗脱2 min(0.00%B· min-1), 从40.5%到48%洗脱14 min(约0.54%B· min-1), 最后立刻以初始梯度平衡色谱柱2 min, 准备下一批牛乳的检测, 平均每批次检测牛乳30份。
同一批次检测结束后用10%甲醇+90%去离子水与100%甲醇清洗色谱柱, 以保证下一批次牛乳的正常检测。
牛乳胶束的散射以及仪器运行过程中产生的随机噪声会对光谱造成干扰, 因此光谱中不仅包含许多有用的化学信息, 还存在大量的背景噪声和无用信息。 为了最大可能的削弱干扰信息, 保留有效信息, 提高模型的稳键性, 正式建模前先对光谱预处理。 分别利用标准正态变量变换(standard normal variate transformation, SNV)、 多元散射校正(multivariate scatter correction, MSC)、 一阶导数、 一阶差分、 归一化(normalize)、 二阶导数和二阶差分7种方法进行预处理。
中红外光谱的波段范围广, 冗余信息繁多, 通过特征提取算法, 能够大大减少光谱维数, 优化算法, 提高模型的识别率。 本文利用CARS、 UVE和SPA算法提取特征变量。
CARS算法[11]基于“ 优胜劣汰” 准则剔除不适应的波长变量, 在有效去除无信息变量的同时压缩共线性变量, 最终选择出针对预测目标最为关键的变量。
UVE算法[12]基于PLS回归系数进行变量选择, 该算法的基本思想是利用回归系数来衡量变量的权重, 消除模型中低贡献率的特征变量。
SPA算法[13]是一种让变量间共线性最小化的算法, 能够减少干扰信息。
通过训练集相关系数(Rc)和训练集均方根误差(RMSEC)以及测试集相关系数(Rp)和测试集均方根误差(RMSEP)对模型的精度和可信度进行评价。 Rc(Rp)高, 则预测结果好, RMSEC(RMSEP)低, 则稳定性好。 各评价指标的相关计算公式如式(1)和式(2)
其中, ypi为训练集或测试集中第i份牛乳的预测值, ymi为训练集或测试集中第i份牛乳的实测值, ymean为训练集或测试集牛乳实测值的平均值, n为训练集或测试集的牛乳总数。
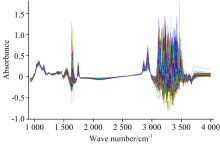
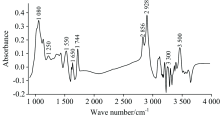
牛乳的原始光谱如图1所示, 水在牛乳中的含量高达80%以上, 1 597~1 712和3 024~3 680 cm-1谱区由于水吸收导致较低的信噪比, 对模型有很强的干扰, 通常在建模前先将这两个区域去除[8, 9, 10]。 鉴于3 024~3 680 cm-1波段的干扰太强, 因此将其舍弃。 图2所示为牛乳的平均光谱。 1 080 cm-1处的谱峰主要与乳糖中的C— O有关, 1 774 cm-1为脂肪的特征谱峰, 2 856与2 928 cm-1处的谱峰分别与脂肪中的甲基和亚甲基有关[14]。 1 250, 1 550和1 650 cm-1为蛋白质的特征谱峰, 酰胺Ⅰ (1 558~1 705 cm-1)、 酰胺Ⅱ (1 480~1 613 cm-1)、 酰胺Ⅲ (1 200~1 280 cm-1)和O=P— O拉伸(1 060~1 100 cm-1)被认为是与蛋白质有关的主要区间[7]。 1 640, 3 300和3 500 cm-1处的谱峰与水的弯曲振动和O— H的拉伸振动有关。
考虑到酰胺Ⅰ 带与水的吸收区域1 597~1 712 cm-1基本重合, 同时Etzion等[7]的研究表明酰胺Ⅰ 带最有可能不受水的影响, 对比了去除1 597~1 712 cm-1前后的效果, 发现保留该部分的效果更好。 在3 680~4 000 cm-1谱区, 没有观察到特征峰, 对比了去除3 680~4 000 cm-1前后的效果, 发现去除该部分的效果更好, 于是选择925.92~3 005.382 cm-1的光谱区域。 此外, 经探索研究发现先对光谱手动降维, 即每隔一个波点(对应波数为3.858 cm-1)取一个透射率, 再进行数据处理, 可以优化模型的最终效果。 因此, 先对光谱手动降维, 使波数范围由925.92~3 005.382 cm-1变为925.92~3 001.524 cm-1, 波点数由540变为270, 最终选择925.92~3 001.524 cm-1的光谱区域用于后续建模。
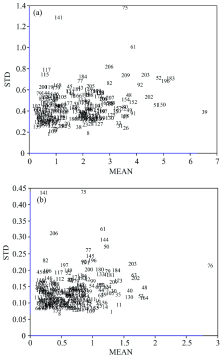
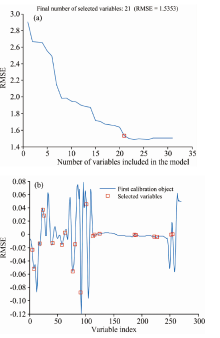
由于牛乳胶状结构不稳定, 容易发生沉淀和析出, 可能出现奇异样本, 本研究利用蒙特卡洛交叉验证法(Monte-Carlocross-validation, MCCV)对α s1和κ -酪蛋白分别进行奇异样本检测与剔除。 MCCV基于 PLS获取最佳主成分数, 利用随机数按4: 1的原则将光谱数据和酪蛋白测定值划分为训练集和测试集, 分别建立PLS回归模型, 设定循环次数为2 500, 计算出各牛乳的预测残差后分别求均值与方差[16], 结果如图3所示, α s1-酪蛋白模型的奇异样本编号为39号、 75号和141号, κ -酪蛋白模型的奇异样本编号为61号、 75号、 76号、 141号和144号。
SPXY(sample set partitioning based on joint X-Y distances)法在划分样本时同时了考虑光谱数据和测定的理化指标, 被划分的样本集更合理[11], 本文利用SPXY将剔除异常后的样本按7: 3划分为训练集和测试集。 其中, α s1-酪蛋白的训练集和测试集样本数量分别为146和62, κ -酪蛋白的训练集和测试集样本数量分别为145和61, 各样本集的数据统计情况如表1所示。
| 表1 利用SPXY算法划分样本集的数据统计 Table 1 Data statistics of partitioning sample sets by SPXY algorithm |
在导数预处理中, 利用Savitzky-Golay求导法进行9点平滑、 3点差分宽度的导数预处理。 使用CARS, UVE和SPA分别对预处理后的光谱数据进行特征提取, 分别找出能够代表α s1-酪蛋白含量与κ -酪蛋白含量的特征变量。 因为α s1和κ -酪蛋白的特征变量选择过程相同, 下文仅以α s1-酪蛋白的特征变量选择为例分别对CARS、 UVE和SPA的变量选择过程进行阐述。
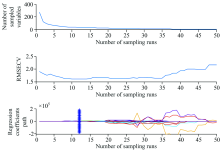
(1)CARS进行变量选择的过程如图4所示, 将CARS的采样次数设为50, 采用5折交叉验证, 重采样率为0.8。 图4(a)表明, 随着取样运行次数的增加, 被选取的特征变量数量在逐步减少。 图4(b)的均方根误差(RMSECV)值先逐渐减小, 表明无用信息被消除, 再逐渐增加, 表明有效信息被消除。 图4(c)竖线处迭代22次, 取得最小RMSECV值。
(2)UVE进行变量选择的过程如图5所示, 将UVE的阈值参数设为0.99, 结合20个主成分数建立PLS模型进行变量选择。 图中左侧曲线表示实变量, 右侧曲线表示添加的随机变量, 两条水平虚线为随机变量的最大阈值线, 两条水平线之间为被剔除的非有用变量, 水平线之外则为建模的特征变量, 共选出108个变量组合。
(3)SPA进行变量选择的过程如图6所示, 根据RMSE的变化来确定被选取的特征变量。 在变量个数的增加过程中, RMSE先迅速下降, 说明光谱中的无效信息被高效剔除, 然后趋于平稳, 说明无效信息基本有效剔除, 选择此过渡点处的变量作为被选取的特征变量组合。
将不同的预处理和特征选择算法结合获得的特征变量组合分别带入SVR模型, α s1-酪蛋白的预测结果如表2所示。 对于CARS算法, 一阶导数预处理的光谱数据带入SVR模型取得了最优效果, 选择的特征变量数量为24, 占建模光谱变量的8.89%; 对于UVE算法, 一阶导数预处理的光谱数据代入SVR模型取得了最优效果, 选择的特征变量数量为108, 占建模光谱变量的40%; 对于SPA算法, 直接将建模光谱带入SVR模型取得了最优效果, 选择的特征变量数量为23, 占建模光谱变量的8.52%。
| 表2 基于3种特征选择算法建立的α s1-酪蛋白SVR预测模型 Table 2 SVR prediction model of α s1-casein based on 3 characteristic variable selection methods |
κ -酪蛋白的预测结果分别如表3所示。 对于CARS算法, 二阶差分预处理的光谱数据带入SVR模型取得了最优效果, 选择的特征变量数量为16, 占建模光谱变量的5.93%; 对于UVE算法, 一阶差分预处理的光谱数据带入SVR模型取得了最优效果, 选择的特征变量数量为55, 占建模光谱变量的20.37%; 对于SPA算法, 一阶导数预处理的光谱数据带入SVR模型取得了最优效果, 选择的特征变量数量为14, 占建模光谱变量的5.19%。
| 表3 基于不同特征选择算法建立的κ -酪蛋白SVR预测模型 Table 3 SVR prediction model of κ -casein based on different characteristic variable selection methods |
对于α s1-酪蛋白预测模型, CARS算法与UVE算法建立的SVR模型训练集Rc和测试集Rp均在0.85以上, SPA算法建立的SVR模型训练集Rc和测试集Rp在0.82和0.85之间, 一阶导数预处理和CARS算法结合建立的SVR模型最优, 训练集Rc和测试集Rp分别为0.882 7和0.899 8, 训练集RMSEC和测试集RMSEP分别为1.136 3和1.372 6。 对于κ -酪蛋白, CARS算法、 UVE算法和SPA算法建立的SVR模型训练集Rc和测试集Rp均在0.85以上, 一阶差分预处理和UVE算法结合建立的SVR模型最优, 训练集Rc和测试集Rp分别为0.880 8和0.890 3, 训练集RMSEC和测试集RMSEP分别为0.534 5和0.535 4。 分别将α s1和κ -酪蛋白含量的最优SVR回归模型用散点图表示, 预测结果如图7和图8所示。
基于傅里叶变换中红外光谱技术, 分别建立了牛乳中α s1和κ -酪蛋白含量的SVR无损快速检测模型。
对于α s1-酪蛋白, 一阶导数结合CARS算法、 一阶导数结合UVE算法和原始光谱结合SPA算法的最优模型提取的特征变量数分别为24, 108和23。 结果表明, α s1-酪蛋白含量的最佳预测模型为一阶导数与CARS算法结合建立的SVR回归模型, 训练集Rc和RMSEC分别为0.882 7和1.136 3, 测试集Rp和RMSEP分别为0.899 8和1.372 6; UVE算法提取的特征变量包含无效信息, 影响了预测精度; CARS算法与SPA算法提取的特征变量数相当, 但SPA算法的精度远低于CARS算法, 表明SPA算法不适合α s1-酪蛋白含量预测模型的建立。
对于κ -酪蛋白, 二阶差分结合CARS算法、 一阶差分结合UVE算法和一阶导数结合SPA算法的最优模型提取的特征变量数分别为16, 55和14; 结果表明, κ -酪蛋白含量的最佳预测模型为一阶差分和UVE算法结合建立的SVR模型, 训练集Rc和RMSEC分别为0.880 8和0.534 5, 测试集Rp和RMSEP分别为0.890 3和0.535 4。 三种算法的预测精度较为接近, UVE算法优于CARS算法与SPA算法, 表明κ -酪蛋白含量最佳预测模型的建立需要提取更多的特征变量。
本研究可为后续利用中红外光谱法对牛乳中其他与过敏有关的蛋白含量进行快速无损检测提供重要参考。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|