作者简介: 章海亮, 1977年生, 华东交通大学电气与自动化工程学院教授 e-mail: hailiang.zhang@163.com
人们对鱼类的品质追求越来越高, 因此对于开发水产养殖中鱼类重要参数脂肪含量的检测显得愈发的重要, 传统的检测方法虽然经过许多研究人员的修改和改进, 但仍然存在费时费力, 需要专业的人员培训存在一些问题。 光谱技术也存在仅使用整条鱼片作为预测样本, 缺乏普遍性, 整个鱼片的成分分布不均匀, 采样时间过长等导致图像质量不高等问题, 该研究通过MCR-ALS算法重建后的数据和图像的增益效果, 评估了采用近红外高光谱成像技术预测并实现鲑鱼片重要参数(脂肪)可视化的可行性。 首先将购买的新鲜三文鱼按照背面和腹部切块分割, 每条三文鱼制作成20个样本, 共100个样本, 其中75个样本用于校正集, 25个样本用于预测集。 用高光谱成像系统采集三文鱼样本的光谱数据, 再通过索氏提取器测定三文鱼脂肪的含量, 并建立其理化值样本, 然后通过MCR-ALS对光谱数据进行重构, 发现重构后的光谱有效信息随着组分推荐评分上升, 通过连续投影算法(SPA)选择特征波长, 并建立最小二乘支持向量机(LS-SVM)模型评估两种预测效果(原始和重建数据)。 MCR-ALS-SPA-LS-SVM的预测精度最高, Rp=0.955 5, RMESP=1.650 5, RPD=3.389 9; 采用MCR-ALS和未处理的模型对鱼片脂肪进行视觉图像预测, 大大减少了噪音的输入, 有效还原了鱼片的轮廓, 并且令鱼的脂肪条纹更加的清晰, 图像质量更优。 进一步分析聚类图像, 通过不同成分的主成分贡献和相同成分的主成分贡献比, 发现类别为20种时, 样品与背景簇存在干扰, 然而采用少量的簇类分析发现, 仅5和10个种类即可完整描绘出整个样品的轮廓, 对于光谱强反应物质存在很好的聚类效果, 具有简化模型的可能。 无论是数据还是图像, 令人满意的预测结果证实了近红外高光谱成像用于鲑鱼脂肪定量和视觉图像预测的可行性, 并且算法的优化大大缩短了检测时间, 为实时在线检测创造了更好的条件。
People's pursuit of fish quality is getting higher and higher, so it is more and more important to develop the detection of the fat content of important parameters of fish in aquaculture. Although many researchers have modified and improved traditional detection methods, they are still time-consuming. It is laborious and requires professional personnel training. There are some problems. The emerging spectral technology also has problems such as low image quality caused by only using the whole fish fillet as a prediction sample, lack of universality, uneven distribution of components of the whole fish fillet, and long sampling time. This study uses the MCR-ALS algorithm. After reconstruction of the data and image gain, the feasibility of predicting and visualizing an important parameter (fat) of salmon fillets using near-infrared hyperspectral imaging was assessed. First, the fresh salmon bought from the market is cut into pieces according to the back and abdomen. Each salmon is made into 20 samples, a total of 100 samples, of which 75 samples are used for the calibration set, and 25 samples are used for the prediction set. Then, the spectral data of salmon fish samples were collected by hyperspectral imaging system, the content of salmon fat was measured by Soxhlet extractor, and the physical and chemical value samples were established. Then the spectral data was reconstructed by MCR-ALS. It was found that the reconstructed spectrally valid information increases with the component recommendation score, and then the characteristic wavelengths are selected by a continuous projection algorithm (SPA), and a least squares support vector machine (LS-SVM) model is established to evaluate the two prediction effects (raw and reconstructed data). MCR-ALS-SPA-LS-SVM has the highest prediction accuracy, Rp=0.955 5, RMESP=1.650 5, RPD=3.389 9. Then, using MCR-ALS and the unprocessed model to perform visual image prediction on fish fillet fat, its effect It greatly reduces the input of noise, effectively restores the outline of the fish fillet, and makes the fat stripes of the fish clearer, and the image quality is better than the latter. Further analysis of the cluster image, through the principal component contribution of different components and the principal component contribution ratio of the same component, it is found that when the category is 20, the sample will interfere with the background cluster, but it is found that only 5 and 10 A single species can completely describe the outline of the entire sample, and it has a good cluster presentation effect for spectrally strong reactive substances. It is possible to simplify the model. Whether it is data or images, the satisfactory prediction results confirm the feasibility of NIR hyperspectral imaging for salmon fat quantification and visual image prediction, and the optimization of the algorithm greatly shortens the detection time, creating better real-time online detection conditions.
鲑鱼是硬骨鱼, 是一种在冷水中高度洄游的鱼类, 主要分布在大西洋、 太平洋和北冰洋交界处海域。 大多数研究表明, 鲑鱼味道鲜美, 富含EPA和DHA等生物活性物质。 三文鱼的不饱和脂肪使人体吸收率高达95%, 而且基本不增加人体体重, 有助于人体降低胆固醇, 预防心脑血管疾病[1, 2]。 传统检测脂肪含量的方法中, 如氯仿-甲醇法、 干柱法、 标准气相色谱法(GS)检测脂肪酸及其他脂肪成分[3], 都具有一定的破坏性。 这些方法虽然经过许多研究人员的修改和改进, 但仍然存在费时费力的问题, 需要专业的人员培训。 因此, 无论是对于水产养殖业者还是购买者, 开发一种更好、 快速、 无损技术完成鲑鱼片或脂肪含量的定量检测具有重要意义。
随着机器视觉和新传感器的出现, 研究人员以很多创意来检测鲑鱼的脂肪, 比如用计算机视觉和立体视觉区分脂肪条纹[4], 用荧光抗体监测脂肪酶及其产物[5], 用可见光和近红外、 拉曼光谱或阻抗特性检测脂肪含量和用超声波快速振荡吸收游离脂肪并测定等[6]。 一些研究人员通过分析可见光/近红光谱确定鲑鱼的储存时间和pH值, 并通过机器学习算法对其新鲜度进行分类, 同时采用短波红外光谱变化解释了冻融循环和冰中冷藏期间血红素蛋白的氧化, 成功建立了筛选多不饱和脂肪酸(PUFA)浓度的模型[7]。 大量研究集中在建立鲑鱼冻融天数、 重复冻融循环次数和细菌生长状态的光谱模型对其新鲜度进行分类, 均取得了较好的效果[8]。 然而, 传统的光谱技术只停留在对数据的解析和分析, 对于更直观的空间图像结构信息缺乏验证。 与Vis-NIR光谱相比, 传统的计算机视觉技术是利用物理传感器获得的图像, 配合适当的计算硬件和软件工具进行分析, 提高视觉质量并实现机器的自我识别[9]。 研究人员利用机器视觉技术的颜色实现鲑鱼伤口和裂缝(包括对张开、 瘀伤和血斑的量化)分类, 并对鲑鱼含水量、 水分活度、 脂肪和质地以及盐度进行了量化的分级处理[10]。 进一步采用二值化图像通过三文鱼鱼体不规则的空隙或不良的花边状外观实现在线质量监测, 并且这种自动图像分析方法可以方便地扩展到其他的形态参数, 如形状、 红白肌区、 肌瘤和肌节区, 构建了一条自动化鲑鱼屠宰线和自动疫苗注射系统[11]。 机器视觉技术侧重于颜色、 形状、 结构等的变化。 然而, 化学成分的改变往往伴随着许多因素的变化, 使影像技术无法准确获取体内代谢物信息。
由于能够同时评估样品的化学成分、 质地和空间结构, 高光谱成像技术(HSI)在肉类和食品行业的发展显著增加[12]。 国内外学者采用高光谱成像技术对鲑鱼探索了细菌生长与脂质氧化之间的相关性, 评估养殖鲑鱼的嫩度信息并建立了可视化图像, 预测鲑鱼的总活细胞数和挥发性碱性氮值等指标[13, 14]。 然而上述工作仍然存在一些问题, 例如仅使用整条鱼片作为预测样本, 缺乏普遍性, 整个鱼片的成分分布不均匀, 采样时间过长等导致的图像质量不高等。 据我们所知, 目前还没有研究优化图像的解构分析并可视化鲑鱼的化学成分。
本研究提出结合MCR-ALS-SPA-LS-SVM算法选择特征波长来开发低成本、 实时的多光谱成像系统。 具体目标是: 优化原始光谱图像数据比较校准模型的预测性能, 以确定准确预测鲑鱼鱼片脂肪含量的最佳模型, 实现鲑鱼脂肪的可视化分析。
三文鱼, 总重372~580 g(平均重512 g), 全长27.5~32 cm(平均长30.5 cm), 购自杭州水产品市场。 敲击鱼头令其死亡, 去除头尾和内脏, 将样品洗涤并干燥。 将每条鱼的背部肌肉均匀地切成左右2个部分作为2个样本。 将每个样品放入塑料密封袋中, 放入装有新鲜碎冰的干净泡沫培养箱(带滴水孔)中, 如图1(a)所示, 然后在0.5~1 h以内快速运送到实验室。
近红外高光谱成像系统结构如图1(b)所示。 波长范围为900~1 700 nm, 共有167个波长。 相机像素为167× 320(光谱维度× 空间维度), 镜头与样品的距离为40 cm。 两盏50 W卤钨灯以45° 角固定在金属支撑架上。 经过多次调整, 移动平台的传输速度设置为1.58 mm· s-1, 以获得不失真的图像。 采集数据前, 在移动平台上放置一块具有反射率的黑色背景板, 关闭所有周围环境光并拧上相机镜头盖采集黑色校准图像, 然后打开卤素灯获得白色校准图像。 最后用吸水纸擦去鱼样品表面的多余水分, 将样品垂直于固定光谱相机移动, 通过线扫描获得三维高光谱图像。
对切割得到的100个小块鱼样品, 采集近红外高光谱图像后, 立即采用标准方法测定脂肪含量; 图1(c)显示了主要仪器(索氏提取器)。 将干燥的鱼肉用研磨机粉碎, 用分析天平称取3~5 g鱼肉粉作为样品, 将白色套筒固定在索氏脱脂仪上, 加入40~50 mL石油醚到玻璃容器中, 白色套筒在打开循环水流的情况下浸入石油醚中。 然后将提取的脂肪放入玻璃容器中, 最后将装有油脂的容器放105 ℃的烘箱中30 min, 使油脂中残留的石油醚完全挥发, 将混合物放入干燥器中冷却30 min, 称重获取的脂肪。
在反射率校正和图像采集后, 从感兴趣区域(ROI)中选择图像, 大小为230× 320像素。 在提取的图像中, 图片的背景与鱼片样品的光谱存在一定的差异[图1(d)], 因此使用简单的阈值分割和掩模操作将背景像素转换为零反射率以去除背景。 具体操作是每张标定图像生成一个灰度图, 灰度图是在400波段(高反射率)的图像中减去30波段(低反射率)生成的, 然后用参数为0.15的阈值对图像进行分割.最后, 将分割区域作为主要区域(ROI), 提取光谱和图像信息。 计算光谱反射曲线的平均值以代表每个样品的光谱。 实验重复上述步骤, 构建了200个样本× 167个波段的光谱矩阵。
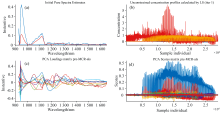
ALS是SVD++算法之一, 以SVD(奇异值分解)为理论基础, 意味着任何矩阵都可以分解为一个正交矩阵和一个对角矩阵以及另一个正交矩阵的乘积。 因此, 可以通过矩阵的低秩重构来完成矩阵A中没有化学值打分的光谱信息的打分预测(由于打分矩阵比较稀疏, 意味着为重构之前评分矩阵中有大量尚未评分的光谱信息), 可以增加光谱信息的有效性。 LS(Iterate once)计算得到的纯谱估计和无约束浓度分布分别如图2(a)和(b)所示。 并将PCA降维转换为SVD分解, 分别得到图2(c)和(d)中的加载矩阵和目标评分矩阵。 对化学成分越来越敏感的组分给予更高的权重, 而对没有反馈的组分给予更少的权重。
原始光谱数据和MCR-ALS重建的数据分别如图3(a, b)所示。 鱼类样品的光谱反射率总体趋势一致(波长范围898.63~1 694.1 nm, 167个波段), 然而反射率值存在一定差异在原始数据中, 这是由于鱼样品的脂肪含量不一致造成的。 腹部脂肪含量高于背部, 中部脂肪含量高于头部和尾部。 在MCR-ALS重构的图中, 可以发现两个主要峰(1 081 nm附近和1 273 nm附近)的位置没有变化, 而在1 643 nm附近增加了一个峰, 增加了原来的有效信息量光谱。
由于原始光谱中有167个波段, 因此提取特征波长以提高SPA算法后续模型计算的效率, 并为实时在线检测做好准备。 原始数据和MCR-ALS重建数据的SPA提取如图4所示, 分别提取了40和25个特征波段。 两组特征谱带分布在各个峰谷处相对均匀。 有研究发现肌肉内羔羊脂肪与950 nm的C— H键的第三次伸展泛音附近的区域有重要的相关性, 奶制脂肪与1 460 nm处的C— H弯曲振动有关, 另外脂肪还包括在(902、 1 052、 1 378~1 386、 1 656和1 695 nm)处C— H 伸缩振动的第一、 第二和第三泛音等[15]。 SPA算法选择的波长不限于这些波长点, 还包括其他有效的波长点, 同时表明特征波长的可靠性和有效性。
 | 图4 提取原始数据和MCR-ALS重建的SPA特征波长Fig.4 Extraction of SPA feature bands reconstructed from original data and MCR-ALS |
在5个完整的鱼片中, 随机选择4个全鱼片作为建模集(共80个样本), 剩余的1个全鱼片作为预测集(共20个样本)。 建模集和预测集的鱼样本脂肪(以湿重计)统计如表1所示。 脂肪化学含量范围3.30%~25.13%, 覆盖范围较大, 说明整条鱼不同采样位置脂肪含量差异较大, 有利于建立稳健的标定模型。
| 表1 校准和预测样本中所有鲑鱼片的脂肪统计 Table 1 Fat statistics for all salmon fillets in calibration and prediction samples |
两个光谱图的全波段模型建立预测结果如表2所示, 其校正集的相关系数均高于0.95, 体现了LS-SVM模型的优越性。 结果表明重建的光谱数据其相关系数优于原始光谱数据, MCR-ALS对纯光谱成分的化学浓度有很好的细化作用。 另外全波段模型的效果略好于特征波段, 分析认为一些隐藏信息没有被挖掘出来, 但相对于计算速度的提高和简化模型, 这个损失完全可以接受。 其原始[图5(a)]和重建[图5(b)]光谱数据的预测值和参考值如图5(a, b)所示, MCR-ALS-SPA-LS-SVM模型的预测相关系数和预测均方根误差达到了令人满意的结果(RP=0.955 5, RMSEP=1.650 5)。
| 表2 不同输入类型下鲑鱼片脂肪含量模型的预测结果 Table 2 Prediction results of models for fat contents in salmon fillet based on different input types, respectively |
在实际数据中, 往往存在许多在空间分布中具有一定形状的成分, 其浓度变化在其形状范围内具有一定的连续性, 这与噪声引起的颗粒状波动不同。 当仅使用非负约束处理此类数据时, 可以发现可视化图像中会出现一些噪声, 或者一个主成分呈现的浓度分布形状也出现在另一个主成分的浓度中, 主成分相互重叠, 影响图像质量, MCR-ALS算法可以很好地解决这些问题。 将原始高光谱图像像素的光谱信息放入模型MCR-ALS-SPA-LS-SVM和SPA-LS-SVM中, 得到以下两个用于预测脂肪含量的可视化图像。 与原始高光谱图像相比, 通过MCR-ALS重建的图像更能反映其脂肪的纹理特征。 未重建图像中的右半边鱼产生了噪声预测信息, 而重建后的图像显示了清晰的脂肪条, 轮廓与原始图像一致。 并且重构后的图像保证了边缘信息, 减少了背景与样本的差异, 更符合客观规律。
该研究对高光谱图像下的化学组成分数据进行了聚类分析, 在前面使用SVD分解10个主成分的情况下, 分别进行了5、 10和20个类别的分析[图7(a, b, c)]。 导入20个类别时, 可以显示更多的结构细节, 但会受到背景的干扰, 出现一些噪声。 但对于5或10类, 背景和样本完全分离, 边缘处理相对较差, 仍能显示鱼片的轮廓和大致的光谱信息。 表明通常不需要对高光谱图像进行高密度表面扫描。 在分析图7每个对应的浓度贡献时发现, 不仅不同主成分对不同簇的贡献差异很大, 而且同种对应的比例也有很大差异, 表明浓度权重较大的主成分信息表示样品的脂肪含量较高, 在空间背景下簇浓度明显极低。 进而可以通过分析聚类的差异来简单区分脂肪含量的高低。 研究表明HSI可以大大缩短检测时间, 为实现在线实时检测创造了良好的条件。
结果表明, 采样近红外高光谱成像技术在950~1 650 nm波长范围内可以准确测定鲑鱼鱼片中的脂肪含量。 MCR-ALS对化学成分浓度的重建能力显示出优异的纯光谱数据。 从模型的比较来看, 全光谱的相关系数(r=0.965 7)和特征波段的一个(r=0.955 5)均优于原始光谱数据。 将高光谱图像带入MCR-ALS-SPA-LS-SVM的最优模型中, 可以很好地实现其可视化。 在脂肪可视化图像中, 脂肪条分布更加清晰准确, 边缘轮廓信息得到修正, 显示出极佳的可视化潜力。 同时也表明MCR-ALS在改善图像空间模型、 减少背景等约束方面具有重要作用。 该研究对拟合的脂肪图进行了进一步的聚类分析, 发现只有5或10个类别可以构成一个好的图像。 在未来的实验或实际生产中, 可以通过扫描现场大大降低光谱密度以提高检测效率, 充分说明了高光谱可视化在鲑鱼脂肪检测中的巨大潜力。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|