作者简介: 赵思梦, 1996年生, 中国农业科学院农产品加工研究所硕士研究生 e-mail: 392246771@qq.com
花生球蛋白、 伴花生球蛋白及亚基含量显著影响蛋白质的凝胶性和溶解性等功能特性, 进而影响其在肉制品、 植物蛋白饮料中的应用效果。 目前常采用提取蛋白质后再用电泳及光密度法测定球蛋白、 伴球蛋白及亚基含量的方法, 操作步骤繁琐, 样品损失量大。 为此收集了178个花生品种, 分别提取蛋白, 采用电泳法测定球蛋白、 伴球蛋白、 23.5和37.5 kDa亚基含量并获得大量数据的基础上, 利用近红外光谱技术进行整粒花生样品的光谱扫描, 将其与传统方法测定的化学值进行拟合, 采用偏最小二乘回归(PLSR)化学计量法构建数学模型。 通过比较单一和复合光谱预处理方式, 对比模型相关系数和误差评估预测模型性能。 确定球蛋白模型最佳预处理方法为2nd-der with Detrend, 校正集相关系数为0.92, 标准差为1.41; 伴球蛋白模型最佳预处理方法为Detrend with 1st-der, 校正集相关系数为0.85, 标准差为1.46; 23.5 kDa亚基含量模型最佳预处理方法为Normalization with 2nd-der, 校正集相关系数为0.91, 标准差为0.53; 37.5 kDa模型最佳预处理方法为Detrend with Baseline, 校正集相关系数为0.91, 标准差为0.89。 外部验证结果表明, 球蛋白预测均方根误差(square errors of prediction, SEP)为1.25, 伴球蛋白SEP为0.73, 23.5 kDa模型SEP为0.47, 37.5 kDa模型SEP为0.75。 本研究基于近红外光谱技术实现了对整粒花生进行球蛋白、 伴球蛋白、 23.5 kDa和37.5 kDa亚基含量的同步、 快速和无损检测, 为育种专家加工专用品种选育和蛋白加工企业原料选用提供了根据。
The contents of arachin, conarachin and subunits significantly affect the gel properties and solubility of peanut proteins, and then affect its application in meat products and beverage. In this study, we collected 178 peanut varieties, measured arachin, conarachin, 23.5 and 37.5 kDa subunits contents by chemical methods. On the basis of peanut sample spectrum scan by near-infrared spectrum technology, we used Partial Least Squares Regression (PLSR) stoichiometry to build a mathematical model with the chemical data. By comparing single and composite spectral pretreatments, model correlation coefficient and errors to value the performance of the models. The best pretreatment method for arachin model was determined as 2nd-der with Detrend, the correlation coefficient of correction ( Rc) set was 0.92, and the standard error of calibration (SEC) was 1.41; the best pretreatment method of conarachin model was detrended with 1st-der, the Rc and SEC were 0.85 and 1.46; the best pretreatment method for the 23.5 kDa subunit model was Normalization with 2nd-der, the Rc and SEC were 0.91 and 0.53; Detrend with Baseline was the best pretreatment method for the 37.5 kDa model, the Rc and SEC was 0.91 and 0.53. External validation results showed the Square Errors of Prediction (SEP) of arachin and conarachin were 1.25 and 0.73, respectively. The SEP of 23.5 kDa model and 37.5 kDa model were 0.47 and 0.75 separately. In this study, the contents of arachin, conarachin, 23.5 and 37.5kDa subunits in the whole peanut were detected simultaneously, rapidly and non-destructively based on NIRS. It’s important for the breeding specialist to select special varieties and raw materials for the protein processing industry.
花生是世界上油脂和蛋白的重要来源, 其抗营养因子含量少[1], 综合利用价值高。 我国2019年花生年产量1737万吨(农业农村部统计数据), 产量居全球首位。 花生中蛋白质含量达25%~36%[2], 花生蛋白中水溶性蛋白约为10%, 盐溶性蛋白占90%, 主要由花生球蛋白、 伴花生球蛋白Ⅰ 和伴花生球蛋白Ⅱ 组成[3], 三者的构成和含量影响蛋白质的性能特性。 花生球蛋白包含4个亚基, 分子量分别为40.5, 37.5, 35.5和23.5 kDa, 其中23.5 kDa亚基含量在18.70%~26.50%, 37.5 kDa含量10.50%~17.90%, 相对较高[3, 4]。 王丽[5]等研究表明, 亚基组成影响蛋白质功能特性; 刘岩[6]等研究表明花生球蛋白比伴花生球蛋白具有更高的热稳定性、 更复杂的空间构象和更差的变性协同性, 伴花生球蛋白具有良好的溶解性和表面疏水性, 其乳化活性、 气泡能力和热凝胶特性皆优于花生球蛋白; 杨晓泉[7]等研究表明, 亚基的构成、 二硫键的稳定性和亲水疏水性等与蛋白的加工性质有密切联系。
传统检测蛋白质组分和亚基含量的方法为聚丙烯酰胺凝胶电泳法(SDS-PAGE), 需要对样品进行脱脂、 提取蛋白质进行SDS-PAGE及光密度分析, 该法分析速度慢, 操作步骤繁多, 样品损耗量大。 近红外光谱分析通过反映分子中含氢元素的化学基团C— H, O— H和N— H分子振动的倍频和合频吸收信息[8], 可以实现快速无损检测, 已经广泛应用于花生中水分、 脂肪、 氨基酸、 蛋白质等的检测。 但检测花生蛋白组分的报道较少, 花生蛋白亚基含量的近红外检测模型鲜有报道。
对整粒花生样品作近红外光谱扫描, 将其与化学值拟合, 结合偏最小二乘回归法进行数学模型的构建。 通过比较单一和复合光谱预处理方法, 对比模型校正集和验证集相关系数及误差, 确定了花生球蛋白、 伴花生球蛋白、 23.5 kDa、 37.5 kDa亚基含量预测模型的最佳预处理方法, 建立了近红外光谱检测方法并进行了外部验证。 为育种专家加工专用品种选育和蛋白加工企业原料选用提供了参考。
采用自然风干花生种子, 去除杂质和破损粒, 在常温下放置24 h, 使样品环境与仪器操作环境保持一致。 选取来自山东、 广东等11个省份的178份花生样品(来源: 花生产业技术体系)进行分析。 先后采集近红外反射光谱和测定蛋白质组分及亚基含量。
采用基于MicroNIR光谱仪开发的便携式花生品质速测仪(型号: peanut 1.0; 厂家: VIAVI Solutions Inc., USA), 该装置为一种高通量花生无损检测装置(图1), 组成部件包括: 箱体、 箱盖、 显示器和光谱仪。 近红外光谱仪采用漫反射光谱, 光谱范围为900~1 700 nm, 光源采取10 W的卤素灯, 性能参数见表1。
 | 图1 本试验中使用的便携式花生品质速测仪和检测配件Fig.1 Portable peanut quality spectrometer and test accessories used in the experiment |
| 表1 速测仪性能参数 Table 1 Spectrometer performance specifications |
便携式花生品质速测仪开机后常温下预热30 min, 将花生样品装入样品杯中, 轻摇晃保证花生种子均匀分布, 每个样品扫描5次, 每扫描一次将样品杯旋转一定的角度, 重复装样扫描三次取平均值。
1.3.1 花生球蛋白与伴花生球蛋白的制备
首先将花生脱壳, 进行晾晒, 置于清水中浸泡脱去红衣, 再用烘箱烘干至水分含量7%左右。 采用正己烷对粉碎机粉碎过后的样品粉末进行脱脂, 常温提取5次, 使其脂肪含量少于1%。 脱脂后在通风橱风干粉碎, 得到的脱脂花生粉4 ℃冷藏备用[9]。
花生球蛋白和伴球蛋白的制备采用Chiou[10]等报道的提取方法, 脱脂后花生粉加硫酸铵至饱和, 离心后复溶沉淀, 透析后冷冻干燥得到花生球蛋白和伴花生球蛋白。
1.3.2 SDS-PAGE电泳及亚基含量分析
根据Laemmli[11]报道的方法进行电泳。 电泳结束后, 将凝胶在染色液中染色1 h, 置于摇床上脱色采集图像。 凝胶染色液采用考马斯亮蓝溶液, 脱色采用高甲醇的醋酸溶液[12]。 23.5和37.5 kDa蛋白质亚基相对含量采用光密度分析软件计算。
模型的构建分析采用The Unscrambler X 10.3软件(CAMO公司, 挪威)。
1.4.1 光谱的预处理
由于近红外光谱的测量容易受到试验环境、 仪器设备、 光散射效应等因素的影响, 因此需要对原始的光谱进行不同的预处理消除噪声, 来提高模型的分析准确性。
常用的光谱预处理方法主要有平滑(Normalization)、 基准化(Baseline)、 标准正态变量变换(SNV)、 去趋势(Detrend)、 多元散射校正(MSC)、 一阶导数(1st Derivative, 1st-der)和二阶导数(2nd Derivative, 2nd-der)等[13], 选取以上七种单一预处理方法中较优的两种按不同顺序组合进行复合光谱预处理, 最终选出最佳光谱预处理方法。
1.4.2 模型建立与评价
对预处理后的光谱和化学值进行偏最小二乘回归法分析(partial least squares regression, PLSR)[14]。 PLSR是一种经典的线性建模方法, 它将光谱数据压缩成为潜在变量的正交结构, 描述光谱信息与参考含量值之间的最大协方差。 与传统的多元线性回归相比, 具有综合筛选光谱数据、 充分提取样品光谱的有效信息、 考虑内在联系等优点, 所构建的模型能更加准确的识别信息[12]。
为使数据拟合反复进行内部交叉验证剔除异常值, 采用外部验证评价模型的稳健性。 通过比较模型的决定系数(R)、 标准差(SEC)和测试集标准差(SEP)来筛选最佳模型, 相关系数高且标准差低的模型稳健性好。
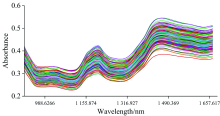
178份花生样品经主成分分析(PCA)剔除异常值后剩余169份, 其原始光谱如图2所示。 花生标准样品集的近红外图谱趋势大致相同, 但不同样品的吸收峰强度不同, 表现出不同的反射率, 说明各样品成分含量不同。 结果表示本试验的花生近红外光谱数据符合建立近红外定量分析模型的要求。
建模所用的169种花生样品的蛋白组分和亚基含量的化学值如表2所示, 收集的花生样品涵盖较广。 样品的花生球蛋白总量变幅为44.30%~71.99%, 伴花生球蛋白变幅为28.01%~55.70%, 花生球蛋白中 23.5 kDa亚基变化范围为5.07%~28.42%, 37.5 kDa花生蛋白质亚基变化范围为7.50%~21.60%。
| 表2 花生样品蛋白组分和亚基含量化学值 Table 2 Chemical values of protein components and subunits in peanut samples |
将样品按照3:1的比例分为校正集和验证集, 用于建模的样品127个, 验证集43个。 数据分析表明预测集化学值含量位于校正集范围内, 可用外部验证进行模型的校正。
2.3.1 光谱预处理方法的确定
为了从光谱中提取与化学组成相关的信息, 消除干扰因素, 采用合适的光谱预处理方法建立稳定可靠的模型至关重要, 研究了多种光谱预处理方法提高信噪比的效果, 包括单一和组合预处理方法, 对相关系数和标准差进行对比确定最佳预处理方法(表3和表4), 花生球蛋白模型的最佳预处理方法为2nd-der with Detrend, 伴花生球蛋白模型的最佳预处理方法为Detrend with 1st-der, 23.5 kDa模型的最佳预处理方法为Normalization with 2nd-der, 37.5 kDa模型的最佳预处理方法为Detrend with Baseline。
| 表3 不同光谱预处理对蛋白组分模型的影响 Table 3 Results of Arachin model with different spectral pretreatment methods |
| 表4 不同光谱预处理对亚基模型的影响 Table 4 Results of 23.5 kDa model with different spectral pretreatment methods |
2.3.2 模型的构建及验证
采用2.3.1节中筛选出的最佳预处理方法对花生球蛋白、 伴花生球蛋白、 23.5 kDa和37.5 kDa亚基含量进行模型构建, 分为校正模型和内部验证模型。
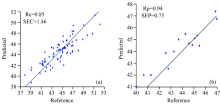
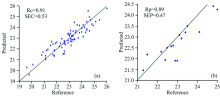
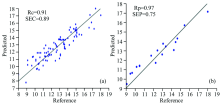
为验证蛋白组分和亚基模型的精确性, 经43个样品对所建模型进行外部验证, 结果见表5。 花生球蛋白模型校正集相关系数为0.92, 误差为1.41%[图3(a)]; 伴花生球蛋白含量模型的校正集相关系数为0.85, 误差为1.46%[图4(a)]; 23.5 kDa亚基含量校正集相关系数为0.91, 误差为0.53%[图5(a)]; 37.5 kDa亚基含量校正集相关系数为0.91, 误差为0.89 %[图6(a)]。 经外部验证, 花生球蛋白的预测值与化学值的相关系数达0.82[图3(b)], 伴花生球蛋白预测值与化学值的相关系数达到0.94[图4(b)], 23.5 kDa亚基预测含量与化学值相关系数达到0.89[图5(b)], 37.5 kDa亚基预测含量与化学值相关系数达到0.97[图6(b)]。
| 表5 模型构建与验证 Table 5 Model construction and validation |
目前NIR技术应用于检测花生蛋白组分的报道较少, 王丽分析141个样品, 建立花生球蛋白模型的校正集相关系数为0.80; 伴花生球蛋白模型校正集相关系数为0.78; 本研究中花生球蛋白相关系数0.92和伴花生球蛋白相关系数0.85均优于已报道模型。 尚未见关于花生蛋白23.5 kDa和37.5 kDa亚基含量的近红外模型。
建立了运用便携式花生品质速测和近红外光谱技术对花生中蛋白质组分和亚基含量进行快速无损检测的方法。 通过
采集的178份数据, 对花生蛋白质组分和含量进行建模分析, 得出花生球蛋白含量最佳模型预处理方法为2nd-der with Detrend, 模型校正集相关系数为0.92, 外部验证SEP为1.07; 伴花生球蛋白含量模型最佳预处理方法为Detrendwith 1st-der, 模型校正集相关系数为0.85, 外部验证SEP为0.73; 23.5 kDa含量最佳预处理方法为Normalization with 2nd-der, 模型校正集相关系数为0.91, 外部验证SEP为0.47; 37.5 kDa含量最佳预处理方法为Detrend with Baseline, 模型校正集相关系数为0.91, 外部验证SEP为0.75。 与目前报道比较, 本方法能较准确的预测花生蛋白质组分和亚基含量。
实现了对整粒花生进行花生球蛋白、 伴花生球蛋白、 23.7 kDa和37.5 kDa亚基含量的同步、 快速、 无损检测, 为育种专家加工专用品种选育和蛋白加工企业原料选择提供了依据。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|