作者简介: 吴纾蕾,女, 2000年生,中国科学技术大学与中国科学院苏州生物医学工程技术研究所联合培养硕士研究生 e-mail: shuleiwu@mail.ustc.edu.cn
目前临床致病菌鉴定方法时效性较差, 期间可能延误最佳治疗时机以及增加细菌耐药性。 因此开发一种快速、 准确、 无培养的致病菌鉴定技术具有较高的临床应用价值。 拉曼光谱能直接作为快速识别菌种的分子指纹, 计算机辅助分类使其在致病菌鉴定领域崭露头角。 然而, 现有研究中基于机器学习和卷积神经网络的分类方法具有泛化性差、 特征挖掘能力不足等问题, 使得致病菌分类精确度难以提高。 因此, 本研究创新性地提出了基于扩散核注意力的Raman Transformer(RaTR)深度学习网络。 RaTR通过使用基于径向基核函数的核注意力计算方式提升网络挖掘拉曼光谱特征的能力、 提高模型分类精度, 并引入扩散过程以提高模型泛化能力。 此外, 使用离散小波变换处理输入数据, 缓解参数数量庞大和小样本问题。 基于开源致病菌拉曼光谱Bacteria_ID数据集和ATCC数据集进行效果验证, 对比实验结果表明, RaTR在上述两个数据集的分类准确率分别达85.83%、 84.73%, 验证了RaTR的分类准确性以及良好的泛化性。 最后, 对模型进行光谱关键特征可视化, 验证了其挖掘特征的有效性。
Current methods for identifying pathogenic bacteria are time-consuming, leading to delays in optimal treatment and promoting antibiotic resistance. Therefore, developing a rapid, accurate, culture-free technique for this scenario has high clinical value. Raman spectroscopy can serve as a molecular fingerprint for rapid bacterial species identification, and computer-assisted classification is the current research hot spot. However, the classification methods based on machine learning and CNN in related works have poor generalization and insufficient feature mining ability, which leads to low classification accuracy. This study innovatively proposes a deep learning network named Raman Transformer (RaTR). RaTR can improve feature miningcapability and classification accuracy using kernel attention computation based on radial basis kernel function, and its model generalization is enhanced by introducing the diffusion process. Moreover, the discrete wavelet transform is proposed to address the excessive parameters and few-shot issues. Experimental validation on the Bacteria_ID and ATCC datasets shows that RaTR achieves classification accuracies of 85.83% and 84.73% respectively, demonstrating its accuracy and strong generalization. Visualizing key spectral features further confirms the effectiveness of feature extraction by the model. Finally, visualizing the spectral key features further confirms the effectiveness of RaTR feature extraction.
2019年因细菌感染而死亡的人数约为1 370万人, 细菌感染已成为全球第二大死因[1]。 目前临床常用的致病菌鉴定方法是光学显微镜观察法和核酸分析法。 光学显微镜法通过观察病菌形态进行鉴定, 具有设备便宜、 操作简便等优势, 但其仅能提供形态学信息, 难以区分形态相似的病菌。 核酸分析法(如 PCR)通过检测病菌的基因组或特定序列进行鉴定, 具备高灵敏度和特异性, 但制样流程复杂, 通常需等待样品积累成批检测, 后续上机检测耗时1~2 h, 难以满足快速检测需求, 可能延误最佳治疗时机并增加细菌耐药风险。 因此, 致病菌鉴定急需技术创新与突破。
拉曼光谱技术基于分子的拉曼散射效应来生成拉曼光谱信号, 能直接作为识别菌种的分子指纹。 与上述方法相比, 拉曼光谱方法在几秒内即可获取一个样品光谱[2], 能够快速鉴定病菌的化学成分, 并以此区分形态相似的病菌。 拉曼光谱是非典型一维谱峰信号, 谱峰位置和强度大小是鉴定样品化学成分的关键[3]。 但目前常用技术生成的拉曼光谱信噪比低、 特征高度混杂, 人工难以分辨, 而高通量单细胞检测耗时且设备昂贵[3], 因此, 利用人工智能进行拉曼光谱数据驱动型分类已成为致病菌鉴定的研究重点[4]。
最初, 机器学习结合主成分分析(PCA)等降维进行分类是热门方案[3]。 如 Nakar等[5]利用核PCA和决策树分类七种食源性病菌, 准确率达87.1%~95.8%。 然而, 随着临床上需识别的致病菌种类增加, 由于训练时的小样本以及维度爆炸问题, 这类方法难以在精度上满足临床需求[3]。 此外, PCA更适合特征理想情况下不相关的表格数据源, 而在拉曼光谱中, 不同拉曼位移的拉曼峰相关, 这导致了模型的泛化性较差[6]。
目前, 一维卷积神经网络(CNN)广泛用于致病菌拉曼光谱分类[7]。 Ye等[8]训练的CNN模型能够高效识别通过拉曼光谱成像的病毒。 Ho等[9]应用CNN识别30种常见细菌病原体, 分类精度达82.2%。 但是, 上述方法使用的一维卷积和池化操作导致了平移不变性[6], 这将拉曼光谱视为横坐标无关的一维信号, 会丢弃对分类至关重要的谱峰组合全局特征。
Transformer通过自注意力机制, 从模型底层捕捉长序列中不同位置的依赖关系, 相较CNN架构更适合于挖掘序列特征, 提高模型建模的准确性[10]。 此外, 其多头注意力机制可使模型能从不同表示空间中获取信息, 进一步提高了模型的泛化能力[10]。 Chang等[11]结合拉曼光谱和Transformer编码器, 成功区分黑色素瘤和正常组织, 分类准确率为99.69%。
综上, 为了充分挖掘拉曼光谱信号包含的分子指纹特征以及提高模型的泛化能力, 本研究设计了基于扩散核注意力的改进Transformer模型— — Raman Transformer(RaTR), 并使用离散小波变换进行数据降维, 实现致病菌拉曼光谱数据的准确分类。 本文提出的方法优势如下: (1)将基于径向基函数核的核注意力Transformer架构创新性地用于致病菌拉曼光谱分类, 提高网络挖掘对应特征的能力, 提升分类精度。 (2)为了更好地捕捉输入数据中的相关信息, 调整注意力权重, 引入扩散过程以提高模型的鲁棒性和泛化能力。 (3)使用离散小波变换处理输入数据, 解决参数数量庞大和缓解小样本问题。
致病菌拉曼光谱的数据长度约为1 000, 直接用未处理数据输入模型会导致维度爆炸和小样本问题[6]。 Ibtehaz等[6]将拉曼光谱数据通过滑动窗口切割成小块, 输入基于Dense的网络模型以此实现稀疏连接, 解决上述问题。 但输入数据的碎片化将导致模型损失全局性特征。 而常用的PCA降维方法将导致原始样本特征重构, 损失可解释性。 因此, 需寻求更为适合拉曼光谱数据的预处理方法。
离散小波变换(DWT)对于解有限、 非周期的信号具有优势, 可以将复杂的信息片段分解成不同位置和尺度的时频分量[12]。 原始信号通过一系列滤波器组, 分级输出细节系数和近似系数, 提高频率分辨率, 解构原始信号。
将DWT用于降维的优势有: (1)DWT能够在不同尺度上对信号进行可逆特征分解, 实现降维同时可以保留原始光谱的特征, 增强模型的可解释性。 (2)DWT可分析拉曼位移对应的拉曼峰变化, 优化拉曼峰特征提取。 (3)拉曼光谱信号信噪比较低, 使用DWT可通过舍去集中主要噪声能量的细节系数, 实现去噪。
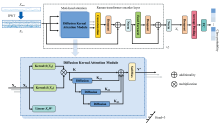
RaTR整体架构如图1, 该网络以Transformer的编码器网络为基本骨架, 并针对拉曼光谱数据的特殊性提出以下两方面的改进: (1)为了进一步解决参数数量庞大和缓解小样本问题, 使用基于径向基函数 (RBF) 的核注意力机制替代原来的线性注意力计算模块; (2)为了更好地捕捉拉曼光谱数据中的特征相关信息, 调整注意力权重, 引入扩散过程以提高模型的泛化能力。
在拉曼光谱中, 不同位置特征峰的组合代表了样品的分子指纹。 Transformer架构从网络的最底层开始关注输入数据的全局相关性, 其编码器本质上是一个特征变换函数, 允许模型根据输入序列中的不同部分来赋予不同的注意权重, 具有擅长提取长序列数据相关性的优势。 因此, 本研究采用该编码器网络作为致病菌拉曼光谱数据分类模型的骨架。
1.2.1 扩散核注意力
Transformer中的自注意力采用计算特征之间的线性依赖关系作为特征权重的度量。 本研究将线性注意力计算替换为RBF的核注意力计算, 结构如图1所示。 每个扩散核注意力模块的输入为Xh∈
其中
式(1)中, Dhead为D/Head。 WQ, Wk, Wv均为要优化的矩阵, 每个单头线性自注意力计算都有3× D× Dhead个参数需要优化, 因此计算效率低, 且增加了优化所需的训练样本数量。
核函数通过将数据隐式映射到高维空间, 然后计算该空间中数据对之间的内积来实现特征权重的度量。 本研究通过使用RBF来代替点积计算, 即使用核注意力机制替换线性注意力机制QKT, 计算表达式如式(2):
< ϕ (xi), ϕ (xj)> 表示输入数据Xh矩阵中的第i行第j列做RBF运算
式(3)中的β 为需要模型优化的核参数, 可以在模型训练期间自适应学习。 因此, 计算每个单头自注意力时所需优化的参数量由3× D× Dhead减少到
1.2.2 扩散过程
本研究引入自注意力扩散过程, 促使不同位置甚至更远位置之间的特征信息更好地交互, 有助于捕捉长距离依赖关系, 从而更全面地对拉曼光谱数据中的特征信息进行建模。 扩散过程可以帮助模型更好地适应不同的输入数据分布情况和任务要求, 从而提高模型的鲁棒性和泛化性[13]。 核注意力矩阵K经过s步的随机游走之后表示为Kds, Ps为状态转移矩阵
不同阶的扩散核注意力通过加权组合, 表示为式(5)
其中权重α j, j∈ [0, 1, …, s]为模型训练自适应学习的权重, 可以更好地适应不同的分类任务。
本文的模型在目前最大开源致病菌拉曼光谱数据集(Bacteria-ID)上进行训练和验证[9]。 该数据集确保了大多数单个光谱是在单个细胞上获取的。 其包含30种细菌, 覆盖了2016年— 2017年斯坦福医院治疗的所有细菌感染病例的94%, 并且大多数分离株来自于重症监护室中的感染病例。 数据集有信噪比较高的60 000例训练数据、 以及信噪比较低的3 000例微调数据和3 000例测试数据。
为了进一步验证模型的泛化性和鲁棒性, 我们使用包含9种致病菌共11 141个拉曼光谱数据的ATCC数据集作为外部验证集[14]。 该数据集也确保了大多数单个光谱是在单个细胞上获取的。 两个数据集中包含的细菌种类及拉曼光谱数据如图2所示, 菌种名后的数字表示不同的品系(strain), 黑色实线为均值。
为了减少对信号进行分析和重构时的相位失真, 本研究使用了有良好对称性的小波基sym8将拉曼光谱数据进行DWT分解。 经预试验, 采用以下最适合RaTR的预处理方法: 首先通过DWT进行4级解构原始数据, 然后舍弃level 1与level 2中的细节系数作为去噪, 最后将余下level 4的近似系数以及细节系数与level 3的细节系数长度对齐, 一并作为模型输入。
RaTR训练时使用交叉熵损失函数, Adam、 SGD分别作为模型预训练、 微调的优化器。 使用Bacteria-ID的训练集进行预训练, 使用十折交叉验证进行模型筛选, 排除偶然因素以及验证模型的泛化性。 取预训练中的最优模型, 使用微调集进行适应性微调, 在微调过程中使用6折交叉验证, 用于测试模型选择。 最后选择微调时表现最好的模型在测试集上进行测试, 评估RaTR的分类准确性, 在试验中得到了85.83%的分类准确率。
使用ResNet、 VGG16、 Transformer以及SVM进行对比实验。 Ho等[9]使用基于一维CNN的ResNet对Bacteria_ID数据集进行分类, 分类准确率为82.2%[11]。 实验结果如表1所示, 采用DWT处理数据可提升分类精度。 RaTR具有最高的分类准确率, 与Ho等[9]提出的一维ResNet模型相比, RaTR的准确率提高了3.63个百分点。
| 表1 不同模型对Bacteria_id数据集的分类准确率(%) Table 1 Classification accuracy of different models on the Bacteria_id dataset (Unit: %) |
RaTR模型测试的混淆矩阵以及每种病菌的灵敏度、 特异性柱状图如图3所示, 大多数菌种的分类结果集中于对角线, 表明模型在样本分类上的准确性较高, 且多数菌种的灵敏度和特异性接近1, 表明模型在识别目标类别及排除非目标类别方面均具有较强的能力。 然而, RaTR在识别同为肠球菌的E.faecalis 1和E.faecalis 2、 同属于肠杆菌科的E.cloacae、 S. marcescens和P.mirabilis的灵敏度相对较低, 可能与这些菌种相似性有关。 通常, E. faecalis 1和E. faecalis 2使用青霉素治疗, E. cloacae、 S. marcescens和P. mirabilis使用美罗培南, 因此模型对这些病菌的低灵敏度在实际应用中的影响较小。 综上, RaTR对致病菌拉曼光谱多分类具有更好的适用性, 实验结果同时表明采用DWT作为拉曼光谱数据预处理方法具有良好的效果。
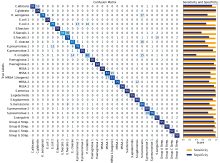 | 图3 分类结果混淆矩阵以及每种病菌的灵敏度、 特异性柱状图Fig.3 Confusion matrix for classification results and Histogram of sensitivity, specificity for each pathogen |
使用ATCC数据集验证模型泛化性能。 将ATCC数据集划分为微调集∶ 测试集=1∶ 1, 对上述预训练模型进行微调, 并采用五折交叉验证方法选择分类精度最高的微调模型进行测试, 结果如表2所示, 可见RaTR相对一维CNN架构模型的泛化性更优。
| 表2 不同模型对ATCC数据集的分类准确率(%) Table 2 Classification accuracy of different models on the ATCC dataset (Unit: %) |
本研究使用Chefer等[15]提出的理论与方法进行光谱关键特征的可视化, 并从模型可解释性方面进一步验证RaTR模型抽取特征的有效性。 该研究使用深度泰勒展开来推理Transformer架构中的输入对整个模型预测结果的相关性得分, 通过合并相关性和梯度信息, 迭代消除负特征值, 即可得到类相关的自注意力贡献得分归因图。
本研究将单个拉曼光谱输入训练好的RaTR模型中, 使用上述可解释性方法, 计算输入特征的归因图, 并使用小波逆变换将其对应到原始拉曼位移上, 如图4所示。 致病菌拉曼光谱各位置拉曼峰所代表的生物组分如图5所示。 通过该实验可知, 在表示RNA、 膜磷脂、 特定氨基酸等细菌种类标识物的拉曼位移处的拉曼峰对模型分类任务贡献最大, 可进一步证明模型抽取特征的有效性与正确性。
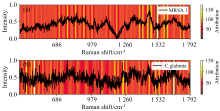 | 图4 MRSA1(a)和C.glabrata(b)的特征相关性归因图Fig.4 Characteristic correlation attribution plots for MRSA1 (a) and C.glabrata (b) |
本研究创新性地提出了基于扩散核注意力的RaTR模型来进行致病菌拉曼光谱分类, 该模型使用DWT进行数据预处理, 使用基于扩散核注意力机制提取特征并进行分类, 并显著提高了致病菌的分类准确率。 RaTR在Bacteria_id数据集和ATCC数据集上的分类准确度分别达到了85.83%和84.73%, 均高于已发表研究工作中提出的模型。 此外, 本研究对模型学习到的特征进行可视化分析, 验证了RaTR模型抽取特征的有效性。 综上所述, RaTR可有效提高致病菌拉曼光谱的分类精度, 为拉曼光谱和深度学习技术在未来临床AI辅助诊断和病理研究中的应用提供了新的研究证据。 该实验模型可扩展至其他类型疾病的多分类检测任务, 并在未来进一步推广到实际的体外诊断应用场景当中。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|