作者简介: 陈 蓓,女, 1982年生,陕西科技大学电气与控制工程学院副教授 e-mail: chenbei@sust.edu.cn
近红外光谱(NIRS)技术因其快速、 无损和高效的特点, 广泛应用于石油、 纺织、 食品、 制药等领域。 然而传统的分析方法在处理变量多、 冗余大的光谱数据时, 往往存在特征提取困难和建模精度不高等问题。 因此提出一种适用于近红外光谱且无需变量筛选的一维波长注意力卷积神经网络(WA-1DCNN)定量建模方法, 该建模方法结构简单、 通用性强、 准确率高。 该研究引入波长注意力机制, 通过赋予不同波长数据不同的权重, 增强模型对重要波长特征的捕捉能力, 从而提高定量分析的准确性和鲁棒性。 为了验证所提出方法的可行性, 采用了公开的4种近红外光谱数据集, 将所提出的算法与加入波长筛选偏最小二乘法(PLS)、 支持向量回归(SVR)、 极限学习机(ELM)三种传统建模方法和一维卷积神经网络(1DCNN)建模方法进行了对比, 并通过模型性能指标均方根误差(RMSE)和决定系数( R2)对模型性能评估。 结果表明没有使用波长筛选算法的WA-1DCNN建模方法性能指标均优于加入波长筛选算法的传统建模方法和1DCNN建模方法。 其中在655药片数据集中测试集决定系数为0.956 3, 相比于1DCNN和加入波长筛选的PLS、 SVR、 ELM提升了4.34%、 12.56%、 18.42%、 11.59%; 在310药片数据集中测试集决定系数为0.957 4, 相比于1DCNN和加入波长筛选的PLS、 SVR、 ELM、 1DCNN提升了2.72%、 8.28%、 7.27%、 1.17%; 在玉米水分和蛋白质数据集中测试集决定系数分别为0.980 3和0.968 5, 相比于1DCNN和加入波长筛选的PLS、 SVR、 ELM提升了6.24%、 1.48%、 1.75%、 6.08%和5.81%、 1.85%、 1.58%、 2.96%; 在小麦蛋白质数据集中测试集决定系数为0.960 0, 相比于DCNN和加入波长筛选的PLS、 SVR、 ELM提升了8.67%、 5.79%、 7.94%、 0.56%。 为了验证WA-1DCNN模型结构的最佳性, 在4种近红外光谱数据集上进行了改变WA-1DCNN模型结构的消融实验。 研究结果表明: 基于波长注意力卷积神经网络是一种结构简单、 通用性强、 准确率高的光谱定量分析方法, 该方法对于近红外光谱定量分析具有促进作用。
Near-infrared spectroscopy(NIRS) technology is widely used in petroleum, textiles, food, pharmaceuticals, etc., due to its fast, non-destructive, and efficient characteristics. However, there are problems with traditional analysis methods,such as difficulty in feature extraction, low modeling accuracy when dealing with spectral data with many variables, and high redundancy. Therefore, this paper proposes a quantitative modeling method of one-dimensional wavelength attention convolutional neural network (WA-1DCNN) suitable for near-infrared spectroscopy without variable screening. The modeling method has a simple structure, strong versatility, and high accuracy.This study introduces the wavelength attention mechanism, which enhances the model's ability to capture important wavelength features by giving different weights to different wavelength data, thereby improving the accuracy and robustness of quantitative analysis. Four publicly available near-infrared spectral datasets were used in this paper to verify the feasibility of the proposed method. The proposed algorithm was compared with three traditional modeling methods that added wavelength screening, namely partial least squares (PLS), support vector regression (SVR), extreme learning machine (ELM), and one-dimensional convolutional neural network (1DCNN)modeling method. The model performance indicators root evaluated the model performance mean square error (RMSE) and coefficient of determination ( R2). The results show that the performance indicators of the WA-1DCNN modeling method without the wavelength screening algorithm are better than those of the traditional modeling method and the 1DCNN modeling method with the wavelength screening algorithm. The R2 of the test set in the 655 tablets dataset is 0.956 3, which is 4.34%, 12.56%, 18.42%, and 11.59% higher than that of 1DCNN and PLS, SVR, and ELM with wavelength screening; the R2 of the test set in the 310 tablets dataset is 0.957 4, which is 2.72%, 8.28%, 7.27%, and 1.17% higher than that of 1DCNN and PLS, SVR, ELM, and 1DCNN with wavelength screening; The R2 of the test set were 0.980 3 and 0.968 5, respectively, which were 6.24%, 1.48%, 1.75%, 6.08% and 5.81%, 1.85%, 1.58%, 2.96% higher than those of 1DCNN and PLS, SVR, and ELM with wavelength screening; in the wheat protein dataset, the R2 of the test set was 0.960 0, which was 8.67%, 5.79%, 7.94%, and 0.56% higher than those of 1DCNN and PLS, SVR, and ELM with wavelength screening. To verify the optimality of the WA-1DCNN model structure in this paper, ablation experiments were conducted on four near-infrared spectral datasets to change the WA-1DCNN model structure. The results show that the wavelength-attention convolutional neural network is a spectral quantitative analysis method with strong versatility, high generalization ability and simple structure, which can promote the quantitative analysis of near-infrared spectra.
近红外光谱技术作为一种绿色分析技术, 有分析速度快、 操作简单、 不需要样本预处理、 可实现无损、 在线分析等优点[1]。 由于近红外光谱存在吸收强度弱、 光谱重叠严重等缺点, 要想进行准确的定量分析, 必须借助化学计量学方法和人工智能建立高质量的模型[2]。 近红外光谱传统建模方法主要有: 多元线性回归(multiple linear regression, MLR)[3]、 主成分分析(principal component analysis, PCA)[4]、 偏最小二乘回归(partial least squares, PLS)[5]、 人工神经网络(artificial neural network, ANN)[6]、 支持向量回归(support vector regression, SVR)[7]等, 这些建模方法已经取得了一定的成功, 但由于样本量不足引起的模型预测精度不高、 光谱分析步骤复杂、 多重共线性等难点问题依然存在。
卷积神经网络(convolution neural network, CNN)首先在图像识别领域取得了成功, 并逐渐在近红外光谱建模得到了一些成绩。 于水等[8]基于Inception网络思想提出一种并联加宽自适应调参的卷积神经网络定量分析模型, 该模型由16个一维卷积层、 2个平均池化层、 1个展平层、 4个全连接层和1个参数调节器组成, 在谷物、 柴油、 啤酒、 牛奶数据集上测试决定系数分别为0.887、 0.878、 0.981、 0.920, 但该模型卷积层数多, 结构复杂。 杨友等[9]将一维光谱数据转化为二维矩阵, 利用卷积神经网络提取特征, 对小麦蛋白质进行最小最大凹罚回归(minimax concave penalty, MCP)测试均方根误差和决定系数分别为0.465 8和0.928 4, 该方法计算量大, 且模型效果不好。 张明赞等[10]结合波长筛选与注意力机制设计了一维卷积神经网络(one dimensional convolution neural network, 1DCNN)用来预测红茶中的各种儿茶素的含量。 该模型有波长选择器、 特征提取器、 注意力机制和含量预测器。 结果表明, 决定系数都在0.96以上, 模型性能较好, 但该模型不仅使用了波长筛选算法, 还加入空间和通道注意力机制, 使得模型参数增多, 训练模型难度增大, 容易过拟合。
针对上述问题, 提出一种适用于近红外光谱且无需变量筛选的一维波长注意力卷积神经网络(wavelength attention-one dimensional convolution neural network, WA-1DCNN)建模方法。 为了评估模型的通用性和泛化能力, 用4种近红外光谱数据集对WA-1DCNN建模方法进行测试, 并与三种传统建模方法PLS、 SVR、 极限学习机(extreme learning machine, ELM)和1DCNN建模方法的预测精度进行比较。 结果表明, WA-1DCNN模型有自主提取特征的能力, 不仅简化了光谱建模中的波长筛选, 而且模型性能要优于传统建模方法。
数据集1: 来源于(https://eigenvector.com/resources/data-sets/), 包含两个仪器(Foss/NIRSystems Multitab Spectrometers), 记录的655个药片活性药物成分(active pharmaceutical ingredient, API)。 采用仪器1(Foss)的光谱数据, 波长范围为600~1 898 nm, 间隔2 nm, 每条光谱有650个波长。
数据集2: 采用公开数据集310个API, 该数据集来源于(http://www.models.kvl.dk/tablets), 波长范围为780~2 526 nm, 每条光谱有404个波长。
数据集3: 来源于(https://eigenvector.com/resources/data-sets/), 其中包括有3个不同的近红外光谱仪器(m5、 mp5和mp6), 测量的80个玉米样品光谱数据以及每个样品的水分、 油脂、 蛋白质和淀粉含量值。 采用m5仪器的光谱数据, 波长范围为1 100~2 498 nm, 间隔2 nm, 每条光谱有700个波长。
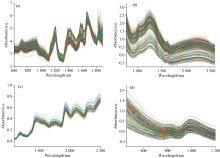
数据集4: 来源于公开小麦数据集(https://www.cNIRS.org), 使用A、 B、 C三个不同厂商9台仪器测得248个小麦蛋白质含量。 采用A厂商的光谱数据, 波长范围为730~1 100 nm, 间隔0.5 nm, 每条光谱有741个波长。 4个数据集的近红外光谱分别如图1(a— d)所示。
1.2.1 波长注意力机制
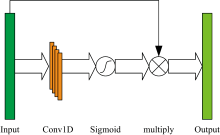
由于光谱数据变量多、 冗余大的特点, 模型在训练中无法准确地筛选出关键的波长。 波长注意力机制旨在告诉模型在何处需要注意什么, 通过增强对关键波长的关注, 能够更有效地提取与标签相关的特征光谱, 提高模型的预测准确性。 在面对复杂和高噪声的数据时, 波长注意力机制能够自适应地调整对各波长的关注程度, 减少噪声的影响, 增强模型的鲁棒性。 其原理是将输入特征层通过一维卷积神经网络提取特征, 由激活函数Sigmoid获得输入特征层中每个特征波长的权值, 将这个权值乘以输入特征层。 如图2所示, 本研究使用卷积核数量为16, 卷积核大小为3, 步长为1的卷积层。
1.2.2 WA-1DCNN网络
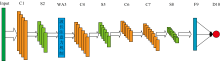
CNN是一种深度学习结构, 它由多个卷积层和池化层组成, 用于进行特征提取和数据压缩, 然后全连接层和输出层对结果进行预测并输出[11]。 传统的1DCNN在处理一维光谱数据时, 对所有的波长关注程度是均等的, 这样可能会导致关键的波长信息会被淹没, 影响模型的准确率。 本工作针对NIRS定量分析问题建立了WA-1DCNN模型, 在1DCNN中加入波长注意力机制, 使得模型更加关注对建模中至关重要的波长区域, 可以更好的让1DCNN提取特征, 提高1DCNN定量分析的精度。 模型结构图如图3所示, 模型的参数设定见表1所示, 包含4个卷积层、 3个最大池化层、 1个全连接层、 1个展平层和一个波长注意力层。
| 表1 WA-1DCNN模型参数设置 Table 1 WA-1DCNN model parameter settings |
在卷积层中, 将卷积核大小设置为3, 是为了确保卷积核能够捕捉相邻波长之间的局部特征。 将步长设置为1, 可以保证卷积核在每个波长位置都进行卷积运算, 提取到所有可能的局部特征。 使用Relu激活函数, 能够有效地传播梯度到较早的层次, 使得在深层网络中梯度能够更快传播和更新参数, 加速模型收敛速度。 如果层是卷积层, 则一维卷积层运算公式如式(1)所示。
式(1)中: k为卷积核, j为核数, M为输入通道数,
采用最大池化(Max-pooling)方法来处理每个特征图, 筛选出最重要的特征数据。 特征图经过最后一层池化后, 由展平层将其转换为一维向量, 并传递到全连接层, 全连接层输出结果。 如果最终池化层为l+1, 并且其输出提供给全连接层, 则全连接的输出为如式(2)所示。
式(2)中: w表示权重, b表示偏差。
在网络模型训练中, 对权重、 偏置等参数进行初始化。 输入通过卷积层、 池化层和全连接层前向传播得到输出值, 然后计算输出值与期望值之间的误差。 将误差通过反向传播的方式返回, 更新权重和偏差, 直到满足可接受的误差条件, 训练完成。 使用预测值与真实值之间差值的平方的平均值作为损失函数, 也就是均方误差(mean squared error, MSE), 如式(3)所示。 使用Adam(adaptive moments estimation)优化器、 学习率为0.000 5对模型进行优化, batch_size=10, epochs=200, 训练完成后, 对最小损失函数值的那次迭代模型权重进行保存。
式(3)中: m为样本数, yi为真实值, ypi为预测值。
训练均方根误差(root mean squared error of calibration, RMSEC)/测试均方根误差(root mean squared error of prediction, RMSEP)是模型的训练集/测试集预测结果与真实结果的误差, 数值越小, 模型预测的准确性越高, 如式(4)所示。 决定系数(R2)是衡量模型对因变量变异性的解释程度, 表示预测值与真实值之间的方差占总方差的比例, 数值越接近于1, 说明模型拟合效果越好, 如式(5)所示。
式(4)和式(5)中: m为校正集样本个数或验证集样本个数,
运行环境: Intel Core i9-10900KCPU, 运行内存64GB, 显卡NVIDIA GeForce GTX3090, 采用Python3.8编程语言, TensorFlow2.11.0和Sklearn1.0.2框架。 此外还使用了Numpy、 Pandas等数据库。
为了验证本文提出的WA-1DCNN模型预测效果, 对4种数据集分别建立WA-1DCNN、 1DCNN、 PLS、 SVR、 ELM 5种定量模型, 选用相同的预处理方法, 最后对五种模型的预测效果进行对比、 测试与分析。 同时为了验证WA-1DCNN模型通用性和泛化能力, 在进行不同数据集实验时, 保持WA-1DCNN模型结构不变。
采用经典的Kennard-Stone(KS)算法, 将数据集按照4∶ 1的比例划分为训练集和测试集, 数据集划分结果如表2所示。
| 表2 数据集划分统计表 Table 2 Dataset partitioning statistics table |
对4种数据集利用PCA、 竞争性自适应重加权(competitive adaptive reweighted sampling, CARS)、 无信息变量消除(uninformative variable elimination, UVE)、 最小角回归(least angle regression, LARS)四种不同波长筛选算法, 分别建立PLS、 SVR、 ELM模型。 以小麦中检测蛋白质为例, 建模结果如表3所示。
| 表3 小麦蛋白质在不同波长筛选和不同模型下的性能指标 Table 3 Performance indicators of wheat protein under different wavelength screening and different models |
其中CARS-PLS、 SVR、 UVE-ELM模型性能最好, 测试均方根误差和测试决定系数分别为0.438 1、 0.902 1, 0.469 5、 0.880 6, 0.298 5、 0.954 4。
对其他数据集分别如2.1所示进行4种不同的波长筛选算法和3种传统建模方法一一组合, 挑选出每个数据集对应的不同光谱筛选与传统建模方法的最佳组合方式, 将其与不使用波长筛选的WA-1DCNN和1DCNN进行对比实验。 结果如表4所示, WA-1DCNN的4种数据集模型性能均最优, 表明该模型的通用性强、 准确率高。
| 表4 不同数据集不同模型的性能指标 Table 4 Performance metrics of different datasets under different models |
WA-1DCNN对比1DCNN发现, WA-1DCNN测试集R2分别上升了0.036 6、 0.027 2、 0.062 4、 0.058 1、 0.086 7, 在RMSEP方面, 也表现出了最佳效果。 说明加入波长注意力机制能够显著提升模型的预测精度和拟合能力。 在1DCNN中加入波长注意力机制, 通过赋予不同波长数据的不同权重, 增强1DCNN对重要波长特征的捕捉能力, 从而使得模型准确率更高、 拟合能力更好, 因此WA-1DCNN模型有自主提取特征的能力。
以655药片中检测API为例, 测试均方根误差和测试决定系数分别为0.008 9、 0.956 3, 0.017 5、 0.830 7, 0.020 3、 0.772 1, 0.015 8、 0.840 4, WA-1DCNN模型的测试集R2比PCA-PLS、 SVR、 UVE-ELM、 1DCNN分别提高了12.56%、 18.42%、 11.59%、 4.34%, WA-1DCNN不仅建模性能最好, 而且训练时间小于加入CARS、 UVE的传统模型, 没有使用波长筛选算法的WA-1DCNN模型性能优于使用了波长筛选的传统模型。 为了进一步验证和对比建模效果, 将测试集数据输入不同建模方法得到的散点图如图4所示。 散点图中的黑线表示y=x的直线, x轴代表真实值, y轴代表预测值。 当点落在直线上表示真实值等于预测值, 样本点越接近于直线说明预测结果越准确。
由图4可以看出, 其中图4(a)WA-1DCNN模型的样本点更接近于直线, 因此WA-1DCNN不但简化了光谱建模步骤, 而且模型性能要优于使用了波长筛选的传统模型。 WA-1DCNN模型在655药片数据集的表现进一步验证了其预测能力要比其他建模方法效果好。 与传统建模方法相比, WA-1DCNN不依赖于复杂的波长筛选步骤, 而是通过训练过程自主提取重要特征, 对噪声较大的数据集能保持较高的准确性, 说明其对复杂光谱数据的鲁棒性表现优越。
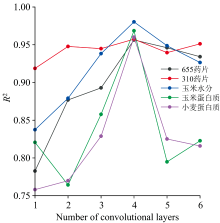
为了验证WA-1DCNN结构的最佳性, 基于原始结构, 改变卷积层数。 分别去掉卷积层C4C6C7、 去掉卷积层C6C7、 去掉卷积层C6、 保持结构不变、 在卷积层C6之后加入一层卷积(filters=64, kernel_size=3, strides=1, activation=‘ relu’ )和在卷积层C6之后加入两层卷积(filters=64, kernel_size=3, strides=1, activation=‘ relu’ )对4种数据集进行验证实验。 将测试集的R2绘制点线图, 如图5所示。
可以看出, 当卷积层数为1时, 模型的R2值最低, 说明卷积层的加入有助于提升模型的拟合能力。 当卷积层数少于4层时, 模型出现欠拟合的现象; 而当卷积层数超过4层时, 模型的R2值开始下降, 复杂度增加, 过拟合的风险加大。 当卷积层数为4时, 模型的R2值达到最大, 由此可见, 采用4层卷积的WA-1DCNN模型结构是更加合理的选择。
提出一种适用于近红外光谱且无需变量筛选的一维波长注意力机制卷积神经网络(WA-1DCNN)定量建模方法, 将该方法利用4种公开数据集进行对比实验。 实验结果表明, WA-1DCNN在测试集取得了最小的RMSE, 最大的R2。 波长注意力机制能更好的让1DCNN提取关键信息, 这种结合方法不仅可以提高模型的泛化能力, 还可以使模型的精度提高。 研究表明: 基于波长注意力卷积神经网络是一种结构简单、 通用性强、 准确率高的光谱定量分析方法。 在后续的研究中可以将该建模方法推广到更多物品的近红外光谱定量分析中。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|