作者简介: 陈灼廷, 1999年生,华中农业大学工学院硕士研究生 e-mail: 1027566415@qq.com
种鸭蛋的孵化是鸭蛋和鸭肉生产的重要保障, 无精蛋不能孵化出雏鸭, 且在孵化箱内容易变质影响受精蛋的孵化。 为了解决人工照蛋剔除无精蛋的劳动强度大、 资源浪费等问题, 以入孵前种鸭蛋为研究对象, 提出了一种基于可见-近红外光谱与深度学习的种鸭蛋孵前受精信息无损检测方法。 使用可见-近红外光纤光谱仪对321枚樱桃谷种鸭蛋(受精蛋144枚, 无精蛋177枚)采集光谱数据, 将光谱数据按3∶1的比例划分出训练集和测试集, 采用在原光谱数据中添加噪声与随机偏移、 随机选取并计算平均光谱两种方法将训练集进行扩充。 设计了一个端到端深度学习模型: 自动编码1维卷积神经网络CAE-1DCNN, 使用卷积、 池化层代替自动编码器中的全连接层, 得到改进的卷积自动编码器CAE, 采用联合优化策略训练CAE-1DCNN模型, 使其具备自动编码器在数据的压缩-重构过程中提取有用特征的能力, 并且能够有针对性地提取适用于分类任务的特征。 采用了竞争性自适应重加权采样算法(CARS)、 连续投影算法(SPA)、 无信息变量消除算法(UVE)三种常用特征波长选取算法和K-最近邻(KNN)、 朴素贝叶斯(NB)、 随机森林(RF)三种机器学习分类模型进行组合, 与本文提出的模型进行对比; 采用t分布随机邻域嵌入算法(t-SNE)将特征提取效果进行可视化。 最后采用梯度加权类激活图(Grad-CAM)将本文提出的模型对光谱数据的关注区域进行了可视化, 探讨了光谱信息的生物可解释性。 研究结果表明, 所提出的CAE-1DCNN模型能较好地提取光谱数据中的有效信息, 判别准确率为95.06%, 可见-近红外光谱技术与深度学习相结合可以实现种鸭蛋孵前受精信息无损检测, 使用联合优化策略训练的卷积自动编码器有较好的特征提取能力。 端到端的CAE-1DCNN模型便于集成, 为开发无损检测设备提供技术支持。
The hatching of duck eggs is an important guarantee for producing duck eggs and duck meat. Eggs without sperm cannot hatch ducklings, and they are prone to spoilage in the incubator, which affects the hatching of fertilized eggs. To solve the problems of high labor intensity and resource waste caused by manually removing -sperm-free eggs through egg photography, this paper takes pre-hatching duck eggs as the research object. It proposes a non-destructive detection method for pre-hatching fertilization information of duck eggs based on visible near-infrared spectroscopy and deep learning. This article uses a visible near-infrared fiber optic spectrometer to collect spectral data from 321 Cherry Valley duck eggs (144 fertilized eggs and 177 azoospermia eggs). The spectral data is divided into training and testing sets in a 3∶1 ratio, and the training set is expanded by adding noise and random offset to the original spectral data, randomly selecting and calculating the average spectrum. This article designs an end-to-end deep learning model: the Autoencoder 1DCNN, which uses convolutional and pooling layers instead of the fully connected layers in the autoencoder to obtain an improved convolutional autoencoder CAE. The CAE-1DCNN model is trained using a joint optimization strategy to enable the autoencoder to extract useful features during the data compression reconstruction process and selectively extract features suitable for classification tasks. This article uses three commonly used feature wavelength selection algorithms, namely Competitive Adaptive Reweighted Sampling (CARS), Continuous Projection (SPA), and Uninformative Variable Elimination (UVE), as well as three machine learning classification models, K-Nearest Neighbor (KNN), Naive Bayes (NB), and Random Forest (RF), to combine and compare with the proposed model. The t-distribution Random Neighborhood Embedding (t-SNE) algorithm is used to visualize the feature extraction effect. Finally, this article used a weighted Class Activation Graph (Grad CAM) to visualize the focus areas of spectral data designed in this paper. It explored the biological interpretability of spectral information. The research results indicate that the CAE-1DCNN model proposed in this paper can effectively extract information from spectral data with a discrimination accuracy of 95.06%. The combination of visible near-infrared spectroscopy technology and deep learning can achieve non-destructive detection of pre-incubation fertilization information in duck eggs. The convolutional autoencoder trained using a joint optimization strategy has good feature extraction ability. The end-to-end CAE-1DCNN model facilitates integration and provides technical support for the development of non-destructive testing equipment.
种鸭蛋的孵化是鸭蛋和鸭肉生产的重要保障, 而在进入孵化的鸭蛋中, 混入了部分不能发育为雏鸭的无精蛋[1]。 无精蛋无法孵化出雏鸭, 并且无精蛋在温湿度较高的孵化箱中极易变质与滋生细菌, 进而影响受精蛋的孵化, 应尽早剔除无精蛋。 在鸭蛋孵化行业内, 判断鸭蛋是否受精的方法仍是对孵化7天以后的鸭蛋进行照蛋, 再由人工挑选出无精蛋。 照蛋法劳动强度大, 效率低, 并且此时的无精蛋已不能作为食用蛋, 导致资源浪费。 因此, 若能在鸭蛋进入孵化前实现无精蛋的快速、 无损检测, 将会为鸭蛋孵化行业带来巨大的经济效益。 在禽蛋检测领域, 国内外学者使用机器视觉、 热成像、 核磁共振成像、 高光谱以及可见-近红外光谱等无损检测技术进行了相关研究[2, 3, 4, 5, 6, 7, 8]。 深度学习方法的自动编码器(autoencoder, AE)是一种较新颖的无监督特征提取算法[9, 10, 11]。 然而, 如何让自动编码器更好地提取特征以及如何更好地利用提取到的特征用于后续任务是自动编码器在应用中需要解决的问题, 一个可行的思路是采用联合优化策略[12]。 联合优化策略将多个任务对应的深度学习模型整合到一个模型, 通过同时考虑多个模块的损失函数或约束条件, 实现整体模型的能力在多个任务上的平衡与提升。
为实现孵前种鸭蛋受精信息无损检测, 解决上述问题, 主要研究工作如下:
(1)提出了一个改进的1维卷积神经网络CAE-1DCNN, 该分类模型包含特征提取卷积自编码器CAE模块、 特征增强残差连接模块和输出模块。 使用卷积、 池化层代替传统自动编码器中的全连接层得到改进的卷积自动编码器CAE, 构成了CAE-1DCNN模型。 使用联合优化训练策略对模型进行训练, 使得模型既具备自动提取光谱数据有用特征能力, 同时也使得提取出的特征适合用于光谱分类任务;
(2)本文提出的CAE-1DCNN模型可以实现输入原始光谱数据, 直接输出预测结果即“ 端到端” 分类, 简化了分析流程, 实验结果验证了CAE-1DCNN模型的可行性;
(3)通过可视化算法, 可视化了CAE-1DCNN模型对光谱数据的关注程度, 分析了表征孵前种鸭蛋受精信息与光谱信息之间的内在联系。
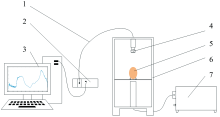
试验样品为321枚产自中国河南省商丘市的樱桃谷种鸭蛋, 种鸭蛋大小接近, 蛋壳颜色均为白色。 在采集光谱数据前, 用浓度为75%的酒精溶液擦拭种鸭蛋表面脏污, 随后大头朝上静置, 自然风干。 种鸭蛋可见近红外透射光谱采集系统如图1所示, 该系统由: Maya2000Pro光纤光谱仪、 光源、 暗箱、 光纤及84UV准直透镜组成。 Maya2000Pro光谱仪采集的波长范围是198.52~1 113.94 nm, 分辨率为0.5 nm。 光源功率为150 W的卤素光源, 卤素光源的有效发光波长范围约为380~1 150 nm, 色温为6 500 K。
进行光谱数据采集前, 将暗箱关闭并将此时光谱仪采集到的数据保存为暗光谱。 然后将光源打开预热30 min, 并保存为参考光谱。 将种鸭蛋竖直放置, 大头朝上, 光谱仪积分时间设置为50 ms, 采集3次取平均值, 保存种鸭蛋透射光谱中的吸光度曲线。
种鸭蛋在孵化箱孵化7天后取出, 进行照蛋和破壳观察, 若能在蛋内发现血丝, 则该枚鸭蛋为受精蛋。 统计后得到144个受精蛋, 177个无精蛋。
由于本研究所用光纤光谱仪的采集波长范围与卤素光源的发光波长范围不一致, 波长范围在198.52~380 nm内的数据存在大量噪声, 且理论上不存在有用信息, 故对原始光谱数据按波段截取, 只保留波长范围为400~1 113.94 nm的光谱数据。
由于光源功率波动、 光谱仪元件的温度偏移、 电流噪声和环境温度变化, 可能导致采集到的光谱数据出现基线漂移, 并引入噪声。 另外同类别的不同种鸭蛋样本所体现的光谱特征具有一定相似性, 因此采用向截取后的光谱数据添加噪声与漂移以及计算样本平均值两种方法模拟上述情况, 用于光谱数据扩充。
添加高斯噪声与基线漂移的计算如式(1)所示
式(1)中, XO为生成的新光谱数据样本; XI为截取后的光谱数据集中的任意一条光谱数据; N为高斯噪声数据, 其形状与光谱数据样本一致并且N的每一个特征维的噪声数值ni服从方差为0.01的高斯分布; D为随机基线漂移量, 其形状与光谱数据样本一致并且D的每一个特征维的基线漂移数值均为d, d是一个随机数, 其取值范围是[-0.47, 0.47], 取值范围与截取后的光谱数据集的平均极差0.94相等。
计算样本平均值扩充数据的方法步骤如下:
(1)从原光谱数据集中随机选取属于同一类别的两条光谱数据A和B;
(2)计算光谱数据A, B每一特征维的平均值, 得到新的平均光谱C;
(3)重复步骤(1)和步骤(2), 直到新生成的光谱数据样本量满足需求。
为了尽量避免样本分布不均, 使用SPXY(sample set partitioning based on joint X-Y distances)算法按3∶ 1的比例划分出训练集(受精蛋105个, 无精蛋135个)和测试集(受精蛋39个, 无精蛋42个)。 再使用前述扩充方法将训练集按原比例扩充至3倍。
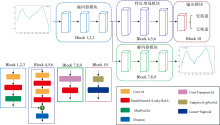
本文提出的CAE-1DCNN模型由以下部分串联组合而成: 特征提取卷积自编码器CAE、 特征增强残差连接模块和输出模块。
1.4.1 特征提取卷积自编码器CAE
针对光谱数据特征维度较高的特点, 设计了一个用于实现特征提取的1维卷积自编码器模块, 该模块包括编码器和解码器。 编码器包括多个1维卷积层、 最大值池化层和1维批量标准化层, 在充分考虑可见近红外光谱数据之间相关性的前提下, 实现光谱数据的特征提取, 降低特征维度。 输入数据经过编码器的特征提取与降维后, 特征维度变为25, 通道维度变为1。 解码器包括多个1维转置卷积层和1维批量标准化层, 将经过编码器压缩的光谱特征逐层还原至与原数据相同的形状。 本文设计的自编码器模块使用泄漏线性整流函数(leaky rectified linear unit, LeakyReLU)作为非线性激活函数, 负斜率设置为0.01, 使用均方误差(mean squared error, MSE)损失衡量解码器的输出与编码器的输入的差异。
1.4.2 特征增强残差连接模块
为了避免神经网络层数增加导致梯度消失, 将多个残差连接模块串联作为自编码器中编码器的后续结构, 并实现特征增强。 残差连接模块内部包括1维卷积层、 1维批量标准化层和随机失活层, 使用LeakyReLU函数作为非线性激活函数。 随机失活层以25%的概率随机使神经元失活, 抑制过拟合。
1.4.3 输出模块
将前续模块的输出结果拉伸为1维张量, 使用全连接层将学习到的特征表示映射到样本标记空间, 使用Sigmoid激活函数用于对样本类别进行概率预测, 二元交叉熵(binary cross-entropy, BCE)损失衡量网络预测分类结果与真实标签的差异。
网络整体结构如图4所示。
1.4.4 联合优化策略的训练流程
CAE-1DCNN模型中包含了两个子任务: 一是由编码器与解码器模块构成的CAE对输入数据的压缩与重构, 二是由编码器、 特征增强残差连接模块以及输出模块构成的1DCNN对输入数据的分类。 具体的训练步骤如下:
(1) 向CAE-1DCNN模型输入样本进行前向计算, 在解码器与输出模块分别得到重构数据与预测分类, 计算输入数据与重构数据的均方误差损失和正确分类标签与预测分裂结果的二元交叉熵损失;
(2) 冻结CAE-1DCNN模型的特征增强残差连接模块和输出模块, 使用均方误差损失更新解码器与编码器的网络权重, 使编码器提取并压缩输入数据的有用信息, 同时使解码器通过提取到的特征还原出输入数据;
(3) 冻结CAE-1DCNN模型的解码器模块, 使用二元交叉熵损失更新输出模块、 特征增强残差连接模块和编码器的权重, 使编码器倾向于提取并压缩对分类任务更有用的信息, 同时使输出模块、 特征增强残差连接模块获得特征增强以及输出正确分类预测结果的能力;
(4) 重复步骤(1), (2), (3)直到训练轮次达到预设值。
CAE-1DCNN模型在训练时设置其最大训练轮次为200, 批量大小为20, 学习率为0.001, 使用Adam优化器更新模型权重。
选用了以下3种光谱特征波长选取算法, 竞争性自适应重加权采样算法(competitive adaptive reweighed sampling, CARS)、 连续投影算法(successive projections algorithm, SPA)、 无信息变量消除算法(uninformative variable elimination, UVE), 作为与使用联合优化训练的CAE进行特征提取的对比算法。 3种光谱特征波长选取算法使用Matlab 2017A进行编写。 选用了以下3种机器学习算法: K-最近邻(K-nearest neighbor, KNN)、 朴素贝叶斯(Naive Bayes, NB)、 随机森林(random forest, RF)与3种光谱特征波长选取算法进行组合, 作为与本文提出的CAE-1DCNN模型对比的分类算法。 3种机器学习算法使用Python 3.9进行编写。 为验证本文提出的CAE模块用于特征提取的有效性, 设计了一个与CAE-1DCNN结构相同, 但是去除CAE中解码器模块且不使用联合优化策略进行训练的1DCNN模型作为对比模型。
孵前种鸭蛋受精信息检测是二分类问题, 以受精蛋为正样本, 无精蛋为负样本。 受精蛋中被正确识别的样本为真阳性(true positive, TP), 被错判为无精蛋的样本为假阴性(false negative, FN), 同理, 无精蛋中被正确识别的样本为真阴性(true negative, TN), 被错判为受精蛋的样本为假阳性(false positive, FP)。 因此使用准确率(Accuracy)、 精确度(Precision)、 召回率(Recall)和F1分数(F1-score)评价分类模型的性能。
采用t分布随机邻域嵌入(t-distributed stochastic neighbor embedding, t-SNE)算法将光谱特征波长选取算法与CAE的自动特征提取方法的效果进行可视化。
采用梯度加权类激活图(graeient-weighted class activation mapping, Grad-CAM)可视化本文提出的CAE-1DCNN模型对于受精蛋和无精蛋光谱数据的关注程度, 探究CAE-1DCNN模型的可解释性。
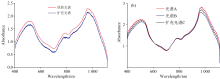
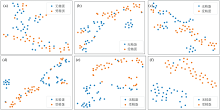
在各模型进行训练之前, 使用t-SNE算法对光谱数据扩充前后的训练集进行了可视化, 验证数据扩充前后数据集的分布情况是否发生改变, 可视化结果如图5所示。
 | 图5 数据扩充前后训练集分布可视化 (a): 扩充前训练集; (b): 扩充后训练集Fig.5 Visualization of training set distribution before and after data augmentation (a): Training set before augmentation; (b): Training set after augmentation |
从图5可以看到, 使用光谱数据扩充方法并没有明显地改变训练集的分布情况, 且从图5(b)可以看到扩充后的训练集在样本空间中分布更加均匀。
本文提出的CAE-1DCNN模型与用于对比的1DCNN模型均使用Python 3.9和Pytorch 1.11搭建, 并在Windows 10操作系统上使用Inter (R)Core (TM) i5-7300HQ CPU和NVIDIA GeForce GTX 1050 GPU进行训练和测试。 数据扩充前后各模型在测试集上的判别结果分别如表1和表2所示, 判别结果的数值保留2位小数。 可以发现进行数据扩充后, KNN与RF两个模型的4个指标有不同程度地提高。 这可能是由于数据扩充后使得数据集中的数据分布更加均匀, 从而使模型更好地学习到了对分类有用的特征。 NB分类模型在数据扩充前后的判别效果均比较差, 可能是光谱数据的特征维之间是存在相关性的。 同时3种特征波长选取算法的使用并没有明显地提升3种机器学习模型的判别效果。 1DCNN和CAE-1DCNN两个深度学习模型则更能适应数据集的扩充, 4个指标均随数据量的增加而获得提高。 CAE-1DCNN模型由于采用了CAE模块与联合优化策略, 扩充前的数据集的判别准确率比1DCNN模型高2.46%, 扩充后的数据集判别准确率比1DCNN模型高3.7%。 本文提出的CAE-1DCNN模型在数据扩充前的准确率高于其余作为对比的模型, 在数据扩充后以准确率95.06%、 精确度100%、 召回率 90.48%、 F1分数95%达到了最好的判别效果。 以上结果表明, 本文提出的CAE-1DCNN模型在实现孵前种鸭蛋受精信息检测具有较好的应用潜力。
| 表1 数据扩充前各模型的测试集判别结果 Table 1 Discriminative results of test set for each model before data expansion |
| 表2 数据扩充后各模型的测试集判别结果 Table 2 Discriminative results of test set for each model after data expansion |
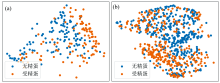
为了进一步探究不同特征选择方法以及本文设计的提取模块、 特征增强模块的效果, 选用了以下数据集: 测试集、 CARS算法选取特征波长后的测试集、 SPA算法选取特征波长后的测试集、 UVE算法选取特征波长后的测试集、 经过CAE-1DCNN模型中CAE编码器提取特征后的测试集、 经过CAE-1DCNN模型中特征增强模块处理后测试集, 采用t-SNE算法进行可视化。 可视化结果如图6所示。
从图6(a)可以看到两类样本混杂程度较高, 聚类效果差, 分类难度较高; 而深度学习模型的模型结构更复杂, 并且其分类能力在一定程度上随数据集的扩大而提高, 所以对未进行降维的数据也能在高维特征空间中实现较好的分类效果, 验证了2.1节中的结果。 从图6(b)、 (c)和(d)可以看到使用CARS、 SPA、 UVE算法进行特征提取之后, 两类样本出现了一定程度的按类别聚集现象, 但仍有样本交错分布, 这可能是2.1节的表2中3种机器学习算法的分类效果有小幅度提升的原因。 从图6(e)可以看到, CAE模块具有更好地特征提取与降维能力, 两类样本各自聚类现象更加明显, 混杂程度进一步降低, 这表明使用联合优化策略使得CAE模块不仅通过编码器-解码器结构实现了数据的压缩-重构过程, 还在一定程度上实现了对分类任务进行有针对性地压缩数据、 提取数据特征的功能, 验证了联合优化策略的可行性与应用潜力。 另外从图6(f)可以看到, 经过CAE模块以及特征增强模块后, 除少量样本外两类样本的分布出现了明显的边界, 证明了特征增强模块在分类任务中的重要作用。
根据2.1节与2.2节的结论, 为了从数据层面探索孵前种鸭蛋光谱数据与孵前种鸭蛋受精信息之间的联系, 计算了受精蛋与无精蛋的平均光谱, 使用Grad-CAM算法计算并可视化已经完成训练的CAE-1DCNN模型对以上两个平均光谱的关注情况, 可视化结果如图7所示。
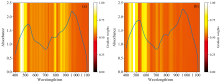 | 图7 模型关注区域热力图 (a): 受精蛋光谱的热力图; (b): 无精蛋光谱的热力图Fig.7 Heat maps of the focus area of model (a): Heat map showing the fertilized egg spectrum; (b): Heat map showing the unfertilized egg spectrum |
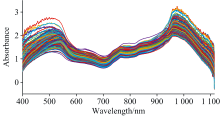
图7中颜色越明亮的区域表明模型对该区域的数据有更高的关注度, 从图7可以看到CAE-1DCNN模型对受精蛋和无精蛋在450~525 nm处的数据均有最高的关注度, 结合图2可以发现在该波长范围内, 样本的数值分布范围也较大。 同时受精蛋和无精蛋的吸光度曲线在525 nm附近存在吸收峰, 这主要与蛋黄色素有关[13]。 因此可以推测受精蛋和无精蛋的蛋黄颜色可能存在一定差异。 另外CAE-1DCNN模型仅对受精蛋在800~900 nm范围内的光谱数有一定关注度, 而吸光度曲线在836 nm附近存在吸收峰, 该处的吸收峰主要与C— H键的拉伸振动有关[14], 由此可以推测受精蛋中有机物如维生素E、 磷脂、 胆固醇等的含量与无精蛋可能存在一定差异。
使用可见-近红外光谱技术实现种鸭蛋孵前受精信息无损检测对鸭蛋孵化行业具有重要意义。 现有研究多采用传统的化学计量法对孵化期种鸭蛋进行检测, 流程复杂, 对检测人员专业水平要求较高。 本文提出了基于联合优化策略的端到端分类模型CAE-1DCNN, 由联合优化策略训练的CAE自动压缩和提取光谱数据中最能体现受精蛋与无精蛋差异的有效信息, 特征增强模块进一步对特征进行增强, 提升了模型的分类能力。 本文提出的CAE-1DCNN模型相对于常用的光谱分析方法, 其优势在于能够减少分析流程。 并且, 模型的检测性能可以随着样本数量的增加而提升。 通过可视化算法探究了特征波长选取算法在分类任务上的缺陷以及鸭蛋孵前受精信息对应的光谱差异。 实验结果表明, CAE-1DCNN模型的分类准确率95.06%、 精确度100%、 召回率 90.48%、 F1分数95%, 可以实现种鸭蛋孵前受精信息无损检测, 端到端的模型便于集成, 为开发无损检测设备提供技术支持。 后续可增加不同品种的种鸭蛋作为训练样本, 增加模型在实际应用中的普适性。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|