作者简介: 赵玉霞, 1999年生,青海师范大学化学化工学院硕士研究生 e-mail: 2234261649@qq.com
“互助”青稞酒作为保护地理标志产品, 对其准确评价分类具有重要意义。 紫外光谱(UV)和近红外光谱(NIR)技术具备快速、 准确、 无损检测、 无需样品预处理等优势, 在食品等领域已广泛应用。 本研究采用UV、 NIR及紫外-近红外中级数据融合光谱(UV-NIR)结合反向传播神经网络(BPNN)法建立了快速、 无损、 高效的“互助”青稞酒判别分类模型。 由于光谱特征峰叠加干扰, 未经优化的光谱受到噪声和基线漂移等影响, 采用标准正态变量变换(SNV)、 Savitzky-Golay平滑(SG)、 一阶导数(1D)和二阶导数(2D)4种预处理方法对光谱进行去噪处理。 相对单一光谱, 融合光谱能够互补多元化学信息, 提高分类模型性能, 通过竞争自适应重加权采样(CARS)、 连续投影算法(SPA)、 主成分分析(PCA)、 变量投影重要性分析(VIP)和变量组合集群分析(VCPA)5种变量筛选方法选择特征变量, 达到优化模型性能及融合两种光谱有效信息。 选择最佳方法建立单一光谱和融合光谱的BPNN模型。 结果表明, UV光谱经SNV预处理以SPA选择30个特征变量建立的分类模型识别效果最好, 分类准确率为100%, MSE值、
Chinese Huzhu Qingke Liquor is a protected geographical indication product, and it is of great significance for its accurate evaluation and classification. Due to the advantages of ultraviolet (UV) and near-infrared (NIR) spectroscopy, such as fast, accurate, non-destructive detection and no sample pretreatment, are widely used in food and other fields. In this study, a fast, nondestructive, and efficient discriminative classification model for Huzhu Qingke Liquor was established based on UV, NIR, and UV-NIR intermediate data fusion spectroscopy (UV-NIR) combined with theback-propagation neural network (BPNN) method. Since the unoptimized spectra are affected by noise and baseline drift due to the superimposed interference of spectral eigenpeaks, the spectra are denoised using four preprocessing methods, namely, standard normal variable transform (SNV), Savitzky-Golay smoothing (SG), first-order derivative (1D) and second-order derivative (2D). Further, relative to a single spectrum, the fused spectrum can complement the diversified spectroscopic information and improve the performance of the classification model, so the feature variables are selected by five variable screening methods, namely, competitive adaptive reweighted sampling (CARS), successive projection algorithm (SPA), principal component analysis (PCA), variable projection importance analysis (VIP), and variable combinatorial clustering analysis (VCPA) to achieve the optimization of model performance and the purpose of fusing the effective information of two spectra. Finally, the best method for establishing the BPNN model for single and fused spectra was selected. The results show that the classification model established by selecting 30 feature variables by SPA after SNV preprocessing for UV spectra has the best recognition effect, with a classification accuracy of 100%. The MSE value,
白酒是中国传统的本土蒸馏饮品, 通常由多种谷物制作而成, 包括高粱、 大米、 小麦和玉米等。 作为中国饮食的重要组成部分, 白酒有悠久历史, 与中国文化有密切关系[1], 在我国社会及食品行业发展中具有十分重要的作用[2]。 白酒酿造过程中, 发酵生香、 蒸馏增香, 可见不仅发酵过程, 蒸馏过程对原酒的品质影响也很大[3, 4]。 “ 互助” 青稞酒是中国原产地保护地理标志产品, 其原料为高原青稞, 其独特的酿造技术、 工艺等, 是青海、 西藏人民喜爱的白酒饮品之一。 青稞酒挥发性组分及原酒的光谱学特性受产酒地区、 生产厂家及酒品种的影响而存在差异[5], 为进一步提高品牌的国内和国际竞争力, 发展地方经济支柱产业, 对“ 互助” 青稞酒进行快速质量评价及掺假识别等研究是行业需要解决的科学问题, 也是产业发展需要解决的技术问题。
长期以来白酒的鉴别方法有两种, (一)专业品酒师用自己的语言描述白酒的各种口味属性来评定原酒的质量等级, 此评定过程受主观因素的影响, 很难得到准确的结果[6]。 (二)传统的分析检测方法, 多种色谱方法应用于白酒物质的鉴定, 如气相色谱、 气相色谱-质谱、 气相色谱-嗅觉测定等[7, 8, 9], 而这些方法成本高、 费时、 费力, 不能满足原酒快速分析的需要。 近年来, 光谱技术结合化学计量学对白酒基酒进行定量、 定性分析研究成为一种发展趋势, 何苗[10]通过紫外光谱、 近红外光谱和三维荧光光谱结合线性判别分析(linear discriminant analysis, LDA)、 支持向量机(support vector machine, SVM)和反向传播神经网络(back propagation neural network, BPNN)等化学计量学方法对不同品牌白酒及同一品牌不同系列的白酒等进行区分鉴别; 周瑞[11]等通过傅里叶变换中红外光谱结合蚱蜢算法优化支持向量机和误差反向传播人工神经网络建模, 实现无损、 快速判别不同类型浓香型白酒; Ding[12]等通过三维荧光光谱结合PCA-SVM方法对209个中国清香型白酒进行了质量控制和等级鉴定的研究。 上述研究, 均通过光谱技术结合化学计量学方法为白酒领域分析提供了可靠的鉴别方案。 由于紫外光谱(ultraviolet, UV)、 近红外光谱(near infrared spectroscopy, NIR)有分析步骤简便、 分析时间短、 对白酒样品醛、 酸、 酯等微量有机物具有选择吸收特性等优势, 近年来很多人采用UV、 NIR光谱对白酒进行了定性研究。 张正勇等[13]通过紫外可见光谱结合化学计量学构建了古井贡酒与紫外可见最大吸收峰强度关系, 从而达到白酒年份酒的快速鉴定。 刘明坤等[14]采用近红外光谱分别建立兰陵浓香型白酒全发酵过程入池、 出池酒醅整体模型, 使得分析工作效率显著提高。 可见UV、 NIR光谱能够满足白酒的鉴别研究, 目前光谱结合化学计量学分析中, UV和NIR光谱联用技术结合化学计量学的报道很少见。
在光谱数据建模前, 为了提高模型稳健性和运算效率, 一般对光谱进行预处理和波长特征变量的筛选[15], 而在实际运算中, 并非所有的模型优化方法均能提高模型预测性能, 因此建模时, 应选取合适的模型优化方法, 提高模型分类预测能力, 减小判别分类误差[16]。 由于BPNN具有处理非线性问题和高维数据的优势, 可以通过训练来自适应调整网络参数和权值, 具有良好的泛化能力, 在模式识别、 数据分类等领域具有广泛应用。 本研究提出一种基于UV、 NIR和紫外-近红外中级数据融合光谱(UV-NIR)结合化学计量学算法的“ 互助” 牌青稞酒快速鉴别方法。 通过收集“ 互助” 牌青稞酒、 非互助青稞酒和其他品牌青稞酒的UV、 NIR光谱数据, 经适当的方法预处理后进行变量筛选, 将两种单一光谱的特征变量矩阵串联在一起组成中级数据融合光谱矩阵, 采用筛选的特征变量建立BPNN模型, 最终探究预处理方法、 变量筛选方法与光谱类型的最佳分类识别组合方案, 为“ 互助” 牌青稞酒品质的快速分析提供一定的参考依据。
白酒样品: 共113个, “ 互助” 牌青稞酒共43种, 购于青海青稞酒股份有限公司专营店; 其他品牌青稞酒53种、 非青稞原料白酒17种, 均购于西宁市各大商场。
甲醇: 色谱纯, 赛默飞世尔科技(中国)有限公司。
无水乙醇: 分析纯, 中国医药集团有限公司。
Lambda系列紫外/可见分光光度计: 美国PerkinElmer公司。
Antaris Ⅱ 近红外光谱仪: Thermo Fisher Scientific公司。
1.3.1 光谱数据采集
取适量样品于1 cm石英比色皿中, 以无水乙醇为参比, 用紫外光谱仪在200~400 nm波长范围内以1 nm间隔扫描光谱, 每个样品扫描3次, 取平均光谱作为样品紫外吸收光谱, 每个样品光谱含201个波长变量; 采用近红外光谱仪透射模式在835~2 630 nm波长范围内扫描样品, 扫描32次, 分辨率为0.38 nm, 每个样品重复测量3次, 取平均光谱即为样品NIR光谱, 每个样品含2 206个波长。 应用采集的UV、 NIR光谱, 分别考察单一光谱、 UV-NIR建立BP模型的样品判别效果。
1.3.2 光谱数据预处理
原始UV、 NIR光谱由于基线漂移[17]、 光散射[18]等产生噪声影响, 在建立模型之前对样品光谱进行预处理消除无关信息的干扰并提取有效信息, 对后续建模的精度具有重大影响。 本研究考察标准正态变量变换(standard normal variate transform, SNV)、 Savitzky-Golay平滑(SG)、 一阶导数(first derivative, 1D)和二阶导数(second derivative, 2D)4种预处理方法对模型的适配性。
1.3.3 数据集的划分
通过Kennard-stone(K-S)[19]方法将白酒样品以7∶ 3比例划分为训练集和测试集, 训练集含79个样本, 测试集含34个样本, 参与识别“ 互助” 牌青稞酒模型的建立。
1.3.4 特征变量筛选
当建模变量数过高时, 不仅耗费大量的建模时间, 更严重的情况可能还会造成维数灾难而难以得到模型计算结果[18], 通过重要特征变量的筛选不仅达到特征提取, 降低数据建模维数, 也可避免光谱中不相关的强吸收峰对分类模型稳健性的影响。 本研究主要采用文献中应用相对比较广泛的几种方法。 在上述预处理方法的基础上进一步筛选特征光谱变量, 主要考察竞争自适应重加权采样(competitive adaptive reweighted sampling, CARS)、 连续投影算法(successive projections algorithm, SPA)、 主成分分析(principal component analysis, PCA)、 变量投影重要性分析(variable importance of projection, VIP)和变量组合集群分析(variable combination population analysis, VCPA)五种变量筛选方法。
CARS[20]算法基于“ 适者生存” 原则, 采用蒙特卡洛随机取样方式, 从校正集中选出一部分样本建模, 通过动态调整光谱特征的权重, 并在稳定时停止采样, 以保留重要特征, 降低数据量和复杂度。 CARS算法的关键为竞争自适应重加权机制, 使用衰减指数法(exponentially decreasing function, EDF)选择波长, 然后使用自适应性权重法优化波长变量, 挑选交叉验证均方根误差最小的变量子集。 本研究UV、 NIR光谱采样次数分别为3、 5次时, 交叉验证均方根误差最小, 选择该采样次数作为CARS算法筛选变量时的参数。
SPA算法是一种前向变量循环选择方法, 目的是找到对样本分类有贡献的关键波长, 而不是使用所有可用的波长[21], 该算法通过找到不相关的波长集合来选取最重要波长, 并逐步缩小这个集合的大小, 实现数据的压缩和特征提取。 通过SPA算法可以选择关键波长的集合, 以避免光谱噪声和不相关波长对数据分析的干扰[22], SPA选择变量的原则是, 新选择的变量是所有剩余变量中前一个选择变量的正交子空间上投影值最大的一个, 以最小化冗余, 提高模型的准确性。
PCA[23]方法在数据降维转换过程中, 得到得分矩阵T和载荷矩阵P, 其中载荷即为该变量表达信息的权重值, 权重绝对值越大, 代表该变量在光谱信息中越重要。 通过设定载荷绝对值阈值的方法筛选得到重要的特征变量。 考察原始UV、 NIR光谱不同载荷绝对值阈值选取重要性变量。
VIP算法[24]计算每个波长的变量投影重要性系数, 并分析VIP系数对目标值的解释能力这两方面的影响作用。 其中变量对目标值的解释能力以通过计算所得VIP系数来表示, 若VIP系数对目标值的解释能力很强, 其VIP值会很大, 该变量会被作为重要变量保留下来。 VIP分析通过计算每个波长的VIP系数, 对各波长按其VIP系数值降序排序, 再按一定规则进行逐步波长筛选。 所有波长的VIP值平方的平均值等于1, 因此有学者提出“ VIP 是否大于1” 作为波长重要性评价标准, 筛选得到特征波长[25, 26]。
VCPA[27]采用二元矩阵抽样(binary matrix sampling, BMS), 该抽样方法考虑了变量间可能存在的交互效应, 通过变量间可能存在的交互影响使用指数递减函数(exponential decreasing function, EDF)强制减少变量, 挖掘出贡献较大的变量, 应用模型总体得到的具有较低交叉验证均方根误差的变量子集建模。 VCPA算法采用了简单而有效的“ 适者生存” 原则来实现选择最佳变量子集。
1.3.5 模型评价
采用BPNN模型建立识别“ 互助” 牌青稞酒、 其他品牌青稞酒和非青稞酒的分类数学模型, 如图1所示BPNN模型方法流程图。 BPNN模型运用分类准确率、 决定系数(determination 邹coefficient of prediction,
2.1.1 紫外、 近红外光谱预处理
对白酒UV、 NIR光谱进行分别进行SNV、 SG、 1D和2D预处理, 图2和图3分别为113个白酒样本UV、 NIR预处理光谱。
 | 图2 不同方法预处理后的UV光谱图 (a): SG; (b): SNV; (c): 2D; (d): 1DFig.2 UV specura after pretreatment with different methods (a): SG; (b): SNV; (c): 2D; (d): 1D |
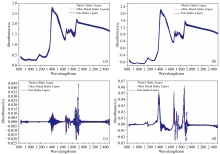 | 图3 不同方法预处理后的NIR光谱图 (a): SG; (b): SNV; (c): 2D; (d): 1DFig.3 NIR spectra after pretreatment with different methods (a): SG; (b): SNV; (c): 2D; (d): 1D |
图2(a— d)所示, 在UV光谱区域, 200~220、 260~280 nm出现明显的吸收峰, 分别为羧基和糠醛吸收峰[28], UV光谱通过预处理后在一定程度上消除毛刺峰现象。
图3(a— d)中, 白酒NIR光谱高度重叠, 难以区分目标组分的光谱信息, 因此必须借助化学计量学手段进行判别分析。 白酒中含有大量的水和醇, 以及微量的风味物质酯、 酸、 醛, 在2 350、 2 306和2 270 nm附近为CH3、 CH2、 CH的合频吸收, 水的合频吸收峰约2 270 nm; 约2 069 nm是— OH的合频吸收区域; 1 936 nm附近为RCOOH、 RCOOR的特征吸收区域; 1 460 nm附近为ROH、 H2O、 CH3、 CH2、 CH的一倍频吸收区域。
2.1.2 主成分分析
将UV、 NIR经SG 预处理全光谱进行主成分分析, UV光谱前3个主成分累积方差贡献率达到90.86%, NIR光谱前3个主成分累积方差贡献率达到99.11%, 说明前3个主成分能代表样本的大部分信息, 可以构建三维PCA散点图对三类样本间的差异进行初步分析, 结果如图4(a, b)所示。 由图4(a), “ 互助” 牌青稞酒和非互助牌青稞酒的主成分散点呈聚类趋势, 分类较好, 但非青稞酒没有明显的聚类现象; 由图4(b), 三类样本的主成分散点分布互相交叉, 相同类别之间没有明显的聚类现象。 分析认为三类样本在主成分方向具有一些相似性, 导致它们在这个方向上不能完全分离。 UV和NIR全光谱进行PCA处理后的前3个主成分不能对目标样本进行正确分类, 因此需要采用模式识别方法进一步分析。
 | 图4 光谱的前3个主成分得分图 (a): UV光谱; (b): NIR光谱Fig.4 Plot of the first 3 principal component scores of the spectra (a): UV spectra; (b): NIR spectra |
2.1.3 特征变量选择
图5(a)和(b)分别为UV、 NIR经过SG预处理光谱后用SPA方法进行变量筛选时选择的变量数与建立BPNN模型分类准确率的关系, 图中分类准确率越高、 MSE值越小, 说明模型效果越优。 由图5(a), 选择5~50个变量时, 其分类准确率均为100%, 因此重点关注MSE值变化。 当SPA方法选择15个特征变量建模时, 其MSE值最小为0.005 6, 因此UV原始和其他预处理光谱采用SPA变量筛选方法建立BPNN模型时均选择15个特征变量。 由图5(b), 相比于其他特征变量数建模, 选择325、 350和375个变量时, 分类准确率最高均为91.18%, 而选择325个特征变量建模时, 其MSE值最小为0.0775, 因此NIR原始和其他预处理光谱采用SPA变量筛选方法建立BPNN模型时均选择325个变量数。
图6(a)和(b)分别为UV和NIR经过SG预处理光谱采用PCA载荷绝对值阈值筛选特征变量时, 不同阈值与分类准确率与MSE之间的关系。 通过选取两种光谱不同载荷绝对值阈值下的变量建立BPNN模型, 通过图6(a), UV的SG预处理光谱选择阈值大于0.025和大于0.05时分类准确率均能达到100%, 其中阈值大于0.05时MSE更小为0.035 9, 因此UV原始和其他预处理光谱均选择PCA载荷绝对值阈值大于0.05的变量建立BPNN模型。 由图6(b), NIR光谱的SG预处理光谱选择PCA载荷绝对值阈值大于0.04和0.1时, 分类准确率均为70.59%, 其中阈值大于0.1时MSE值较小为0.139 1, 因此NIR原始和预处理光谱均选择PCA载荷绝对值阈值大于0.1的变量建立BPNN模型。
 | 图6 (a)UV光谱PCA阈值与模型指标的关系图; (b)NIR光谱PCA阈值与模型指标的关系图Fig.6 (a) Qlot of UV spectral PCA thresholds versus model metrics; (b) Plot of NIR spectral PCA thresholds versus model metrics |
本研究UV、 NIR光谱CARS方法采样次数分别为3、 5次时, 交叉验证均方根误差最小, 选择该采样次数作为CARS算法筛选变量时的参数。 VCPA筛选特征变量时依据经验选取500和50分别作为BMS和EDF运行参数。
图7(a)是对预处理后的光谱, 经不同的方法筛选特征变量后, 用BPNN建模所得的预测效果。 由图7(a)观察, 通过对原始光谱和预处理后的光谱用CARS筛选变量并建模, UV-SG与UV-NIR-SNV的BP模型
对于UV、 NIR以及UV-NIR三类光谱数据, 其预处理方法与变量筛选方法的最佳组合列于表1, CARS、 SPA、 PCA、 VIP筛选变量建模通过SG和SNV预处理光谱即可达到建立优良预测性能的BPNN模型要求, VCPA变量筛选方法与1D、 2D预处理方法结合建模能够满足准确分类预测的效果。
| 表1 光谱预处理方法与变量筛选方法建立 BPNN模型的最佳组合 Table 1 Optimal combination of spectral preprocessing methods and variable screening methods for BPNN modeling |
按表1所述最佳组合方法分别对UV、 NIR和UV-NIR三类光谱数据进行处理, 并建立BPNN模型, 所得的特征变量数、 模型预测能力指标分别见表2、 表3、 表4。
| 表2 UV光谱最佳模型优化组合评价指标对比表 Table 2 Comparison of evaluation indexes of the optimal model optimization combination for UV spectroscopy |
| 表3 NIR光谱最佳模型优化组合评价指标对比表 Table 3 Comparison of evaluation indexes of the best model optimization combination for NIR spectroscopy |
| 表4 UV-NIR融合光谱最佳模型优化组合评价指标对比表 Table 4 Comparison of evaluation indexes of the optimal model optimization combination of UV-NIR fusion spectra |
由表2, UV光谱每种最佳模型优化组合方法的分类准确率均为100%, 即
由表3, NIR光谱中SG-PCA、 1D-PCA和2D-VCPA组合方法建模, 分类准确率和
由于UV、 NIR光谱共有2276个波长变量, 光谱进行低级数据融合难以运行BP模型, 只能采取利用得到的中级数据融合光谱建立BPNN模型。 由表4, 上述模型优化组合方法得到的UV-NIR融合光谱均能达到良好的分类效果, 其中SNV-CARS、 SG-PCA、 SNV-PCA、 2D-VCPA组合方法的BPNN模型分类准确率和
由表2、 表3、 表4分析可知, UV-NIR建立模型各评价指标整体效果优于使用UV、 NIR单一光谱建立的模型效果。 图8(a, b)为UV-NIR-SG-PCA模型优化方法后建立BP模型的分类效果, 其模型预测和识别能力均能达到100%。
BPNN模型相比于PCA分析, 在分类鉴别目标对象时更有优势。 BPNN模型性能随选取方法的变化而互有差异, 且并非所有方法都能使模型得到优化。 对于鉴别“ 互助” 牌青稞酒的预测模型, UV光谱进行SNV-SPA模型优化组合方法, BPNN模型各指标得到不同程度的改善; 对于NIR光谱, 当选取SG-PCA模型优化组合方法后, BPNN模型分类鉴别效果最优; 选用SG-PCA模型优化组合方法得到的UV-NIR建模, BPNN模型预测能力最强; 通过模型优化组合方法得到的UV-NIR与单一光谱建立的BPNN模型预测分类效果相比, UV-NIR建立模型的各指标值比UV、 NIR单一光谱建立模型各指标值均有一定程度的提高。 进一步证明光谱融合技术能更多地体现不同类型白酒的差异, 对青稞酒的快速识别与分类更加有效。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|