作者简介: 王 琰, 1982年生, 中国食品药品检定研究院研究员 e-mail: wy@nifdc.org.cn
β-内酰胺类抗生素是临床中一类重要的抗感染治疗药物, 青霉素G酰基转移酶(PGA)是新型酶法工艺生产β-内酰胺类抗生素的关键技术, 不同菌属来源的PGA具有不同的蛋白序列、 热稳定性及立体选择性, 这些性质会产生不同催化反应活性, 对抗生素合成和生产至关重要。 红外光谱可对蛋白类物质进行结构表征, 基于蛋白质组学的质谱法可从肽段水平对不同蛋白类物质进行鉴定, 由于其复杂精密性使得操作更为简便的红外光谱法为PGA蛋白快速鉴定提供了一种有力分析工具。 本研究采用超滤法和干燥制膜方法对PGA样本进行前处理, 达到纯化PGA原液、 去除基质干扰目的, 同时又能克服溶液浓度低、 信号弱的难题增强红外信号响应。 建立高通量红外光谱法对11批不同菌属来源的PGA进行分析, 结果显示不同菌属PGA红外光谱特征区(1 700~1 500 cm-1)存在相同蛋白经典酰胺红外吸收峰, 同时在指纹区1 200~750 cm-1范围内呈现多个差异化红外吸收峰。 选择指纹区差异化吸收峰谱带(830~795、 1 027~1 020、 1 085~1 080 cm-1)、 采用加权K最邻近(KNN)分类算法优化建立PGA溯源模型, 结合蛋白质组学质谱法结果对不同PGA进行菌属来源分类分析。 结果显示11批PGA分为两类, 第I类结果包括PGA 1、 PGA 3、 PGA 7、 PGA 8, 为大肠杆菌来源, 第Ⅱ类结果包括PGA 2、 PGA 4~6和PGA 9~11, 为无色杆菌来源。 PGA溯源模型分类结果与蛋白质组学分析结果一致, 验证了溯源分类模型的适用性和可行性; 采用该溯源分类模型对3批未知菌属PGA蛋白进行考察, 验证模型的准确性; 最后通过对不同天时间的考察, 进一步验证模型的耐用性。 研究结果表明, 加权KNN高通量红外光谱法可实现对不同菌属来源的PGA快速溯源分类, 方法简便、 准确耐用, 为β-内酰胺类半合成抗生素酶法工艺中催化酶的结构表征和快速分析提供了一种新的检测工具。
β-lactam antibiotics (BAs) are an important class of anti-infective drugs in clinical practice. Penicillin G acylase (PGA) is a key technology used in the new enzymatic process to produce of BAs. PGAs derived from different bacterial origins have different protein sequence structures, thermal stability, and stereo-selectivity, which cause different catalytic activity and are crucial for antibiotic synthesis and production. Infrared spectroscopy (IR) can be used to characterize the structure of high molecular weight proteins. Proteomics-based mass spectrometry can identify different protein substances at the peptide level, but its complexity makes it harder to operate. The simple IR method provides a powerful analytical tool for rapidly characterizing PGAs. This article explored the selection of ultrafiltration and drying membrane preparation methods for the pre-treatment of PGA samples. This way could purify PGA raw solutions and remove matrix interference, while it could also overcome the problem of low PGA solution concentration to enhance IR signal response. Besides, a high-through put IR method was optimized and established to analyze 11 batches of PGAs from different sources. All IR spectra of PGAs showed classical IR absorption peaks of amide groups at the characteristic region (1 700~1 500 cm-1). There were still differential IR absorption peaks within the 1 200~750 cm-1 fingerprint region. A traceability model was established by selecting differentiated absorption peak spectral bands at fingerprint regions (830~795, 1 027~1 020, 1 085~1 080 cm-1). Based on the analysis of proteomics mass spectrometry, a weighted k-nearest neighbor (KNN) algorithm was employed to analyze different PGAs. It showed that 11 batches of PGAs were divided into two classes: those including PGA 1, PGA 3, PGA 7, and PGA 8 belonged to class Ⅰ and were identified as the proteins fermented from E. coli, while the rest of those—PGA 2, PGA 4~6, and PGA 9~11 belonged to class Ⅱ and were produced from Achromobacter sp. CCM 4824. This result verified the applicability and feasibility of the established traceability model, which was consistent with the proteomics result. Then, three batches of unknown PGAs were collected for determination by the traceability model to externally validate the accuracy. Finally, the robustness of the established model was further validated by examining 11 batches of PGAs on different days. The results demonstrate that the high-throughput IR method based on the weighted KNN could rapidly trace PGAs from different bacterial origins. This method is simple, accurate, and durable. It provides a new detection tool for the structural characterization and protein classification of the catalytic enzymes used in producing BAs by enzymatic process.
β-内酰胺类抗生素在临床抗感染治疗中占据重要地位, 这类抗生素目前的工艺主要为新型酶法工艺, 将含有β-内酰胺环的商品化中间体(如6-氨基青霉烷酸或7-氨基头孢烷酸等)与含有不同酰基供体的侧链化合物在青霉素G酰基转移酶(penicillin G acylase, PGA, EC 3.5.1.11)的催化作用下快速制得抗生素, 具有绿色环保、 反应步骤少等优势[1, 2, 3, 4]。 PGA是酶法工艺的关键质量属性, 对生物催化半合成β-内酰胺类抗生素的质量至关重要。 PGA来源于自然界各种微生物, 特别以由革兰氏阴、 阳性菌发酵而得PGA应用最为广泛[5, 6, 7, 8, 9]。 由于不同来源的PGA具有不同立体结构、 不同的对酸和热的稳定性, 导致酶催化活性亦存在差异, 会影响酶法工艺的催化生产效率。 目前, 酶法工艺正大规模用于抗生素生产, 亟需对所应用的商业化PGA催化酶菌属类型进行溯源研究, 对于了解PGA应用发展和抗生素酶法工艺监管均具有重要意义。
基于蛋白质组学理念的高分辨质谱法对蛋白类生物大分子物质的定性和定量具有天然优势, 通过对蛋白样本进行变性、 还原、 烷基化固定和酶切等一系列前处理, 将完整蛋白转化为“ 碎片化” 的各种特征短肽, 经质谱测定得到不同二级肽段碎片和“ 自上而下” 蛋白序列数据库比对分析, 可对未知蛋白进行准确鉴定[10, 11, 12]。 这种方法虽然具有高选择性和可直接获取蛋白氨基酸序列等丰富信息, 但是存在仪器精密昂贵、 操作复杂、 数据分析耗时等弊端, 在推广和应用于质量和工艺评价时受到挑战。 红外光谱技术是一种快速、 无损的分析方法, 在蛋白质结构表征方面具有独特优势, 近年来被越来越多地应用于蛋白质定性检测[13, 14, 15, 16], 尚未有应用于PGA评价研究的报道。 本研究将上述两种方法有机结合, 针对一定数量范围的PGA蛋白样本利用高通量傅里叶变换红外光谱(high-throughput fourier transform infrared spectroscopy, HT-FTIR)进行快速测定, 经蛋白质组学质谱法深度关联分析, 采用优化的加权K最邻近(k-nearest neighbor, KNN)分类算法构建菌属分类模型。 经验证的快速分析模型可用于PGA未知菌属蛋白样本的快速溯源, 有效降低了检测和数据分析的复杂性, 方法的准确性和耐用性好, 使得PGA蛋白质常规菌属定性分析更为简便、 省力。
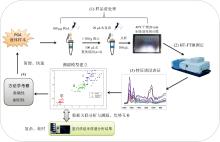
HT-FTIR快速分析方法的研究步骤如图1所示: (1)样本前处理。 采用超滤法对PGA原液进行纯化, 去除基质干扰。 以96孔硅板为载体对PGA纯化液采取干燥制膜方式进行样本制备; (2)优化并建立HT-FTIR检测方法对不同的PGA进行测定, 对特征峰表征和比较; (3)提取指纹区差异化谱段进行预处理, 选用加权KNN算法对关键差异谱段数据进行分析, 将分类分析结果与蛋白质组学质谱法结果进行关联性分析对比, 建立PGA溯源模型; (4)方法学考察: 将收集到的其他3批未知菌属PGA样本同时采用建立的高通量红外光谱分析模型与蛋白质组学质谱法进行定性分析, 对模型进行外部验证考察准确性; 对不同PGA于不同时间(天)进行测定分析, 验证模型的耐用性。
模型建立采用的11批PGA液体样本(建模样本)均由企业提供: PGA 1和PGA 2来自企业1, PGA 3~PGA 6来自企业2, PGA 7~PGA 11分别来自企业3~企业7; 模型验证采用的3批未知PGA液体样本(PGA 12~PGA 14, 验证样本)分别由企业8~企业10提供; 实验中所用水均为实验室自制超纯水。 Tensor 27型红外透射光谱仪(配高通量扫描HTS-XT型高通量透射采样扩展附件、 DTGS检测器和单面抛光96孔硅板, 美国Bruker公司), Ultimate 3000 RSLCnano和Fusion Lumos型纳升液相超高分辨质谱联用仪(美国Thermo公司), DKN412C型干热灭菌箱(日本Yamato公司), Eraeus Fresco17型离心机(美国Thermo公司), 截留分子量为10 kDa的500 μ L超滤管(美国Pall公司), 微量移液器(德国Eppendorf公司)。 使用OPUS 7.0工作站采集光谱图, 用OriginPro 9.0进行红外图谱绘制, 用Matlab2019b进行建模。
由于PGA液体样本为发酵原液, 存在大量发酵过程中产生的复杂基质残留, 研究采用超滤法对样本进行前处理, 目的是分离-纯化-浓缩样本, 排除复杂基质对测定的干扰, 同时可获取增强型红外特征峰。 方法如下: 吸取相当于100 μ g蛋白量的PGA原液置超滤管上层套管中, 于室温下13 000 g离心力将上层溶液完全离心过滤至下层, 使大分子PGA蛋白被保留于上层, 而其他小分子的复杂基质被离心至下层液。 再将上层套管中加入100 μ L水进行置换清洗, 按上述条件离心至完全, 重复操作2次。 最后吸取20 μ L水加入上层套管中复溶PGA。 吸取该溶液10 μ L于96孔硅板, 11批PGA液体样品分别各制样2份(n=11× 2)。 将硅板置于40 ℃烘箱干燥20 min, 制成固态样本膜, 待测。
将载有样本膜的96孔硅板置于HTS-XT型读卡槽中进行红外光谱扫描, 扫描范围为4 000~650 cm-1, 分辨率为4 cm-1, 扫描间隔为1 cm-1, 每孔依次、 连续进行累积光谱扫描, 累积光谱扫描次数为64次, 测定指标为吸收光谱(A), 以硅板本底作为空白进行背景扣除。
采用蛋白质组学质谱方法对PGA样本进行蛋白鉴定。 采用胰蛋白酶对PGA样本于37 ℃下进行膜上过夜酶切, 采用纳升液相Orbitrap质谱联用仪测定分析酶解后的溶液。 色谱柱为Acclaim PepMap RSLC C18分析柱(75 μ m× 250 mm, 2 μ m), 质谱仪采集为数据依赖型(DDA)检测模式, 工作站为Xcalibur(版本4.1.50), 利用Proteome Discoverer软件将检测结果与UniProt公共蛋白质数据库中PGA菌属进行检索并鉴定蛋白样本, 具体分析测定方法见文献[17]。
特征波段选择在本研究的方法建立中非常重要, 是提高模型质量的关键。 本研究利用红外光谱波长归属明确、 吸光度与量值符合比尔郎伯定律的特点, 在光谱预处理(PGA样本光谱图依次进行基线校正、 矢量归一化和一阶导数处理, 平滑处理点数为13)的基础上, 用无信息变量删除(uninformative variable elimination, UVE)法[18], 通过消除无信息变量有效去除干扰, 通过选择有信息的变量及其重要性排序, 筛选与PGA蛋白组最相关的特征谱段, 提高不同谱段区域的红外特征吸收光谱对不同批次菌属来源的PGA的分辨能力, 增强模型的稳健性和可解释性。
与化学物质相比, 生物样本具有较大变异性, PGA酶为多种蛋白的混合物, 同时菌属为基于生物学的分类而非化学结构, 这些对PGA酶的溯源准确性都是较大的挑战。 加权KNN法重点考虑最邻近数据点的距离对于判断结果的影响, 通过权重计算结果最大值的类别为测试样本的类别, 能大大增加判别准确率[19]。 主成分分析是将原有特征变量数据降维成几个新特征变量的低维数据, 用少数新特征变量进行数据建模, 便于分析和数据可视化[20]。 本研究将筛选的PGA特征谱段, 通过主成分分析提取特征变量, 与蛋白质组学分类结果相关联, 用加权KNN法建立PGA溯源模型, 并对模型进行验证。
由于PGA原液浓度较低(< 10 mg· mL-1), 为增加红外信号响应, 建立以96孔硅板为载体、 采用干燥制膜的方式将液态样本转化为固态斑点进行红外测定。 该法既可克服红外信号响应弱的难题又能够实现高通量样本扫描。 通过对干燥制膜条件考察发现, 40 ℃条件下干燥20 min可使PGA溶液较好干燥并形成稳定固态膜; HTS-XT型采样扩展附件配备的96孔硅板每孔点样体积约1~20 μ L, 对不同点样体积进行考察结果, 发现10 μ L上样体积可获得大小稳定、 均一的斑点膜。 采用上述方法制膜, 部分PGA样本原液制得的斑点颜色明显不同于其他正常斑点, 且形成的深色、 浑浊的斑点不易被清洗而在硅板上产生较强印迹, 会影响硅板的正常使用和重复利用。 鉴于PGA蛋白亚基分子量大于20 kDa, 实验又选择截留分子量为10 kDa的超滤膜对PGA原液进行纯化前处理, 如图1中(1)所示, 所得溶液澄清且制膜后的斑点呈透明状, 实验后斑点较易清洗不留印迹, 硅板可重复正常使用不受污染。 本研究建立的样品前处理方法包括超滤纯化和干燥制膜两部分, 具体方法见“ 1.2” 项所述。
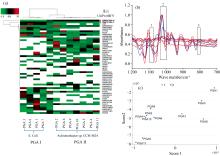
采用所建立的方法对11批PGA原液进行样品前处理, 再通过优化的高通量红外光谱法[16]进行红外光谱测定获得PGA红外特征光谱。 图2(a)结果显示, PGA在3 500~750 cm-1范围内出现不同形式的红外吸收峰, 而其特征吸收峰主要出现在1 800~750 cm-1范围。 图2(b)所示, PGA除呈现蛋白经典红外吸收峰— — 酰胺Ⅰ 带(~1 650 cm-1)和酰胺Ⅱ 带(~1 545 cm-1)外, 还在1 450和1 405 cm-1附近呈现两个弱的吸收峰。 研究中发现, 不同菌属来源PGA间的光谱差异主要体现在指纹区1 200~750 cm-1范围内, 呈现形式各异的红外特征峰: PGA 1、 PGA 2、 PGA 3、 PGA 7、 PGA 8、 PGA 9该6批PGA在1 100~1 000 cm-1范围内具有强吸收峰, 主要与蛋白质氨基酸侧链上的硫醚C— S— C、 羧基和羟基的C— O的伸缩振动(ν )有关; PGA 1、 PGA 2、 PGA 6、 PGA 8该4批PGA在~925和860 cm-1波数处具有明显吸收峰; 特别是, PGA 1、 PGA 3、 PGA 7、 PGA 8该4批PGA在~815 cm-1波数处具有不同程度的红外吸收峰, 而其他PGA此处未见此吸收峰。 在1 000~750 cm-1范围内红外吸收峰主要与蛋白质氨基酸侧链上取代苯基的C— H面外弯曲振动(γ )有关, 上述PGA红外光谱特征峰结构归属见表1。
| 表1 PGA红外光谱振动峰归属 Table 1 Assignment of vibration peaks in PGA infrared spectrum |
分析结果表明, 通过高通量红外测定法可获得有效PGA特征信息, 包括PGA特征区(1 700~1 500 cm-1)酰胺带典型吸收峰和指纹区(1 200~750 cm-1)差异吸收峰。 指纹区光谱差异可用于分析不同PGA间的差异和来源, 揭示不同PGA发酵工艺的差异。
本研究采用蛋白质组学质谱法对11批PGA进行变性、 还原、 烷基化、 过夜酶切前处理和高分辨质谱测定分析[17], 将质谱检测结果与UniProt数据谱库比对, 获得菌属分类结果如图3(a)所示, 检索到53个可信的蛋白, 这些蛋白有无色杆菌(Achromobacter sp. CCM 4824)、 大肠杆菌(E. coli)和木糖氧化产碱菌(Alcaligenes xylosoxydans)等来源, 还有较多的同为E. coli来源的不同基因工程化蛋白。 通过进一步对特征肽(数量≥ 2)、 蛋白覆盖度(> 60%)、 蛋白长度(> 90 kDa)及非冗余数据筛选, 最终, PGA 1、 PGA 3、 PGA 7和PGA 8该4个蛋白鉴定为大肠杆菌来源(UniProt编号为P06875), 定义为第Ⅰ 类PGA, 其特征肽段平均约为3条; 而PGA 2、 PGA 4、 PGA 5、 PGA 6、 PGA 9、 PGA 10和PGA 11该7个蛋白鉴定为无色杆菌来源(UniProt编号为Q3ZEF0), 定义为第Ⅱ 类PGA, 其特征肽段平均约为23条。
采用蛋白质组学分类与红外光谱特征区、 指纹区相关联, 用UVE法筛选与PGA菌属相关的特征谱区: 原始波长3 525个维度, 经过UVE波长筛选后, 得到56个关键数据维度, 主要分布在830~795、 1 027~1 020和1 085~1 080 cm-1三个谱段[图3(b)]; 同时PGA蛋白红外特征峰的表征结果也能对该三段区域做出合理解释。 本研究将其作为特征谱段用于菌属溯源和分类模型的建立。
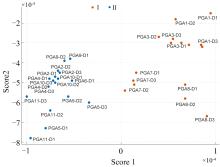
采用主成分分析提取特征变量, 保留2个主成分, 解释方差分别为89.2%、 6.4%, 足以代表95.6%的特征信息。 采用KNN算法建立分类模型, 结果表明加权KNN法的预测准确度为100%, 较经典KNN法(准确度90.2%)更有效。 其中, 加权KNN法中光谱距离计算方法为欧氏距离, 采用距离权重法确定类别, 权重为距离平方的倒数, 利用交叉检验确定最优的k值。 用加权KNN法建立特征变量的分类模型, 最优k值为10。 用25%留出验证法验证模型, 结果如图3(c)所示, 11批不同PGA中, PGA 1、 PGA 3、 PGA 7和PGA 8被判别为Ⅰ 类(即来源于大肠杆菌), PGA 2、 PGA 4、 PGA 5、 PGA 6、 PGA 9、 PGA 10和PGA 11被判别为Ⅱ 类(无色杆菌), 与蛋白质组学质谱分析法结果一致, 分类准确率100%, ROC曲线AUC值为1.0。
2.5.1 方法准确性
采用建立的PGA溯源模型对来自另3个企业的3批未知PGA样本(验证样本, 编号分别为PGA 12~14)进行外部验证, 考察方法准确性。 图4(a)显示, PGA 12 与PGA 14归为第Ⅱ 类, 提示两者来源于无色杆菌; 而PGA 13归为第Ⅰ 类, 提示PGA 13蛋白为大肠杆菌来源, PGA 12~14测定的光谱结果分别见图4(b)。
蛋白质组学质谱的分析结果显示, PGA 12和PGA 14与无色杆菌蛋白(UniProt蛋白数据库编号Q3ZEF0)序列高度匹配, 而PGA 13与大肠杆菌(UniProt蛋白数据库编号P06875)序列高度匹配。 对比分析结果显示, 所建溯源分析方法对PGA蛋白的鉴别结果与蛋白质组学质谱方法分析结果相一致, 准确度100%。
2.5.2 方法耐用性
采用建立的PGA溯源模型于不同时间(D1、 D2和D3)分别对11批PGA进行测定和考察。 图5结果显示, 连续三天测得的PGA分析结果平行性好且与建模结果一致, 11批PGA菌属分类结果分别与大肠杆菌和无色杆菌鉴别结果准确对应。 分析结果表明该法具有良好的精密度, 方法可重复性高, 耐用性好。 由于PGA菌属本身的复杂性和变异性, 例如环境的改变会影响大肠杆菌和无色杆菌的代谢物, 进而对PGA产生影响; 而模型是基于现有样本的统计学模型, 在模型的应用中可能会涉及模型未涵盖的其他PGA样本, 超出模型的预测能力。 当出现这种情况的时候及时向模型中补充新样本光谱更新模型, 将扩大模型的适用范围, 逐渐提高模型的耐用性。
开发了高通量红外光谱PGA蛋白酶菌属溯源分析方法, 对PGA特征谱进行了结构解析, 建立了基于PCA特征变量的加权KNN判别模型, 对不同菌属来源的PGA蛋白酶准确溯源。 研究结果表明, 现阶段应用于抗生素酶法工艺规模化生产的PGA催化酶主要为大肠杆菌和无色杆菌来源。 本法具有微量无损、 简单快速、 准确耐用、 可实现自动化的特点, 为PGA蛋白酶类抗生素酶法工艺质量控制、 保持工艺稳定性提供快速有效的分析手段, 同时也为生物技术药物的连续制造提供科学有效的过程分析技术(PAT)工具。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|