作者简介: 倪钦如, 女, 2000年生, 云南师范大学物理与电子信息学院硕士研究生 e-mail: 1810453425@qq.com
肺癌严重威胁人类健康, 近年我国发病率逐渐增加。 影像学和病理组织学检查是肺癌的主要筛查方式。 影像学检查作为初筛方法, 应用广泛, 但存在不确定性。 病理组织学检查结果准确, 是肺癌诊断的“金标准”, 而组织样本的获取对人体创伤较大。 有必要开发一种可靠且创伤小的肺癌诊断方法。 血清样本的获取比病理组织样本的获取便捷, 且创伤小。 拉曼光谱具有测试简单、 快速及灵敏度高等优点, 可获取血清的生化信息。 该研究使用拉曼光谱技术测试155例健康受试者和92例肺癌患者的血清样本。 在1 800~800 cm-1的波段范围内对健康受试者和肺癌患者血清的拉曼光谱进行曲线分峰拟合, 发现肺癌患者的拉曼光谱在1 005和1 091 cm-1附近的子峰面积百分比较健康受试者分别增加3.36%和5.32%。 在964、 1 522和1 586 cm-1附近的子峰面积百分比, 肺癌患者比健康受试者降低了2.3%、 2.82%和5.6%。 曲线分峰拟合结果初步表明, 肺癌患者的血清中类胡萝卜素、 氨基酸、 核糖、 核酸等生化物质含量发生了变化。 为了深入探索肺癌患者血清的拉曼光谱特征, 利用机器学习挖掘血清的拉曼光谱数据中隐含的信息。 首先使用主成分分析(PCA)提取光谱的特征变量, 并将获得的特征变量分别应用于支持向量机(SVM)、 随机森林(RF)、 k邻近(kNN)、 逻辑回归分类(LRC)、 决策树(DT)和贝叶斯(Bayes)算法建立分类模型, 再使用留一交叉验证法评估模型的预测性能。 结果显示: SVM模型对肺癌患者血清的拉曼光谱分类效果最好, 准确率、 灵敏度、 特异性和F1分数分别达到98%、 94.44%、 100%和97.14%。 SVM模型的9折交叉验证ROC曲线下面积的平均值为0.94, 说明SVM算法的预测性能较好。 研究表明, 血清拉曼光谱结合机器学习算法可对肺癌进行有效诊断, 该技术创伤小且准确率高, 是一种潜在的肺癌诊断技术。
Lung cancer is a serious threat to human health. In recent years, the incidence of lung cancer has been increasing in China. Imaging examination and histopathological examination are the main screening methods for lung cancer. Imaging examinations are widely used as a preliminary screening method, but they have some uncertainties. The result of the histopathological examination is accurate, so the histopathological examination is the “gold standard” of a lung cancer diagnosis. However, the acquisition of tissue samples can cause traumatic lung injury. Therefore, developing a reliable and minimally invasive method for lung cancer diagnosis is necessary. Acquiring serum samples is more convenient and less invasive than pathological tissue samples. Raman spectroscopy has the advantages of a simple operation, rapid sensitivity, and the ability to provide biochemical information on serum samples. This study obtained Raman spectra of the serum in 155 healthy subjects and 92 lung cancer patients. Curve fitting was applied to the Raman spectra data, and characteristic differences between healthy subjects and lung cancer patients were found in the range of 1 800~800 cm-1. The curve fitting results showed that compared with healthy subjects, the area percentages of sub-peaks around 1 005 and 1 091 cm-1 of lung cancer patients increased by 3.36% and 5.32%. On the contrary, the area percentage of sub-peaks around 964, 1 522 and 1 586 cm-1 of lung cancer patients decreased by 2.3%, 2.82%, and 5.6%. The preliminary results of curve fitting showed that the biochemical substances of carotenoids, amino acids, ribose, and nucleic acids in the serum of lung cancer patients were altered. To investigate the Raman spectral characteristics of serum in healthy subjects and lung cancer patients, machine learning methods were used to obtain the hidden information of the Raman spectral data. First, principal component analysis (PCA) was used to extract the characteristic variables of the spectra. The characteristic variables were applied to support vector machine (SVM), random forest (RF), k-nearest neighbors (kNN), logistic regression classification (LRC), Decision Tree (DT), and Bayesian algorithm, respectively, to build classification models. The model’s predictive performance was evaluated by the leave-one cross-validation method. The results showed that the SVM model best classifies serum Raman spectra. The accuracy, sensitivity, specificity, and F1 are 98%, 94.44%, 100% and 97.14%, respectively. The average of values of the 9-fold cross-verification ROC area under the curve for the SVM model was 0.94, which indicated that the SVM model had a good predictive performance. The result showed that serum Raman spectroscopy combined with machine learning methods can effectively diagnose lung cancer. This technique is minimally invasive and highly accurate; it is a potential diagnostic technology for lung cancer.
肺癌是呼吸上皮细胞的恶性肿瘤。 世界卫生组织公布的统计数据显示, 2020年中国新发癌症病4 568 754例, 其中肺癌新发病例占总数的17.9%[1]。 目前, 肺癌的常规诊断方法包括影像学检查(X射线胸片、 电子计算机断层扫描CT、 核磁共振成像MRI等)和病理组织学检查。 影像学检查便捷, 但其属于间接性检测, 不能反映病理组织的生化变化, 存在一定的不确定性。 病理组织学检查结果准确, 但需要进行手术或穿刺获得病变组织, 对患者肺部损伤较大。 有必要建立一种灵敏度高和创伤小的方法来诊断癌症。
拉曼光谱是一种测试简单、 分析速度快、 灵敏度高的光学检测技术。 通过光和物质的相互作用, 获取物质分子键振动、 转动等信息, 常用于大分子结构的研究[2, 3]。 近年来拉曼光谱已被广泛应用于研究人体血清。 血清中含有许多与疾病相关的生物标志物, 血清中生化物质的变化与疾病的发生密切相关[4, 5]。 当疾病发生时, 血清中氨基酸、 核酸、 类胡萝卜素、 蛋白质等相关物质的生化特性会发生改变。 血清样本的获取只需取受试者静脉血5 mL, 再高速离心即可。 该方法相比于病理组织样本的获取, 具有方便、 创伤小等优点。 本研究选用拉曼光谱对肺癌患者的血清进行研究。
随着机器学习的快速发展, 数据挖掘技术在不断更新。 这些数据挖掘技术也被应用于分析拉曼光谱数据, 在疾病诊断方面取得一定成果。 例如Li等[6]将血清的拉曼光谱与线性判别分析(LDA)等结合实现乳腺癌的辅助诊断, 分类模型的准确率高达100%。 本工作前期基于血清的光谱信息结合偏最小二乘判别分析(PLS-DA)对肝癌患者进行诊断, 灵敏度和特异性分别为92.85%和95.23%, 诊断效果较好[7]。 在此基础上进一步探索, 将机器学习算法用于分析血清的拉曼光谱, 用支持向量机(support vector machine, SVM)、 随机森林(random forest, RF)、 逻辑回归分类(logistic regression classification, LRC)、 k近邻(K-nearest neighbor, KNN)、 决策树(decision tree, DT)和贝叶斯(Bayes)算法建立分类模型。 综合比较分类模型在测试集上的评价指标, 探索血清的拉曼光谱结合机器学习算法在肺癌诊断中的适用性。
实验所用的血清样本均来自云南省肿瘤医院。 采集空腹受试者的静脉血5 mL, 在4 ℃下以3 000 r· min-1离心20 min, 取上清液(血清)作为实验样本进行拉曼光谱采集。 其中肺癌患者样本92例, 健康受试者样本155例。 样本的采集获得受试者同意, 且通过伦理委员会审查。 肺癌患者样本均得到术后病理组织学检查证实。
使用英国ANDOR公司生产的SR-500型号的激光显微共聚焦拉曼光谱仪。 仪器分辨率为1 cm-1, 焦长为500 nm, 光栅为1 200 l· mm-1。 在532 nm的激发光源波长, 12 mW的激光发射功率和50倍的物镜下进行光谱采集, 每条拉曼光谱的扫描时间为10 s。 测试时, 选取50 μ m× 50 μ m的区域进行扫描(区域分割为5行, 每行5点, 每个点采集1次), 取25次采集的光谱平均值作为该血清样本的拉曼光谱。
为了减少实验过程中荧光背景、 激光功率波动、 光谱强度、 基线漂移、 光谱噪声等因素对后续数据分析的影响, 将采集到的所有光谱进行常规数据预处理, 包括宇宙射线去除、 背景扣除(Labspec5软件自动扣除背景)、 基线校正(迭代自适应加权惩罚最小二乘法)、 平滑处理(Savizky-Golay平滑法)、 纵坐标归一化(将光谱数据通过等比缩放至0~1区间)和数据中心化等。
拉曼光谱在Python3.10.10中进行数据处理, 并建立分类模型。 选取支持向量机(SVM)、 k近邻(KNN)、 随机森林(RF)、 逻辑回归分类(LRC)、 决策树(DT)和贝叶斯(Bayes)算法建立分类模型。 本实验中, 拉曼光谱包含1 039个数据点, 每个数据点都包含一个特征信息, 导致数据维度较高, 同时也造成了信息的冗余。 直接使用原始数据进行训练会导致模型的判别精度下降。 主成分分析(PCA)是特征数据提取中最常用的方法, 可将高维数据降为低维表示, 以去除冗余信息, 提高模型的判别性能。 本研究中首先采用PCA对拉曼光谱数据进行降维, 结果显示主成分1和主成分2的累积贡献率达到95.07%。 将主成分1和主成分2提取的光谱信息作为模型的输入进行数据分类。
肺癌患者统计为阳性, 健康受试者统计为阴性。 所有测试集样本可以分为真阳性、 假阳性、 真阴性和假阴性。 真阳性指被模型正确判别为患病的患者, 用TP表示。 假阳性指被模型错误判别为健康的患者, 用FP表示。 真阴性指被模型正确判别为健康的健康受试者, 用TN表示。 假阴性指被模型错误判别为患病的健康受试者, 用FN表示。 分类模型的性能指标通过灵敏度、 特异性、 准确率、 精确率、 召回率和F1分数进行评估, 计算如式(1)— 式(6)。 F1可以提高精确率和召回率的同时缩小两者之间的差异, F1的值越大说明模型的质量越高。
拉曼光谱采集范围为1 800~800 cm-1, 该波段属于拉曼光谱的“ 指纹区” , 血清中蛋白质、 脂质、 类胡萝卜素、 糖类等有机化合物的拉曼特征带主要出现在该波段。 与健康人群的血清相比, 肺癌患者血清中类胡萝卜素、 蛋白质、 核酸和脂质等成分的含量和构象会发生细微的变化[8]。 健康受试者和肺癌患者在该范围内的平均光谱和误差带见图1。 对健康受试者和肺癌患者血清拉曼光谱进行相关分析, 相关系数为0.966, 整体较为相似。 在970~800 cm-1范围内出现多个弱吸收的特征峰, 不易辨认。 在1 800~970 cm-1范围内, 两组样本光谱的特征峰呈现出一定的差异。
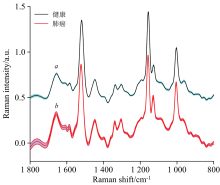 | 图1 健康受试者(a)和肺癌患者(b)的归一化平均光谱(曲线)和标准误差(阴影带)Fig.1 Normalized mean spectra (curves) and standard deviations (shaded bands) for healthy subjects (a) and lung cancer patients (b) |
由于原始光谱中吸收带的相互叠加, 一些弱吸收峰被掩盖, 不能真实反映样本中分子结构或含量的变化。 为了全面分析健康者和肺癌患者血清中脂类、 糖类、 蛋白质等物质的差异, 本研究选用相同的拟合函数(Gaussian函数), 其表达式如式(2)
式(7)中, ν 为频率; ν 0、 γ G以及A分别为初始频率、 半峰宽和波峰面积。 对肺癌患者和健康受试者血清的拉曼光谱进行全波段曲线分峰拟合, 将重叠的峰剥离出来, 并采用叠加子峰面积在全谱中的百分比定量说明特征带的变化。 健康受试者和肺癌患者血清光谱的曲线分峰拟合结果见图2(a, b)和表1(Area%表示子峰面积所占叠加带总面积的比例)。 曲线分峰拟合结果和原始光谱的相关系数为0.998, 较好地重现了光谱的叠加过程。 肺癌患者在1 005和1 091 cm-1附近子峰面积百分比较健康受试者显著增强, 峰面积百分比分别增加了3.36%和5.32%。 在964、 1 158、 1 522和1 586 cm-1附近, 肺癌患者的吸收子峰面积百分比较健康受试者降低, 分别降低了2.3%、 1.2%、 2.82%和5.6%, 可能因为肿瘤细胞的增值需要大量的营养物质, 导致蛋白质的分解, 与其他学者研究结果一致[9]。 对比健康受试者和肺癌患者血清的拉曼光谱拟合结果, 在1 129、 1 307、 1 339、 1 448、 1 553和1 659 cm-1附近叠加带上, 肺癌患者拟合子峰的面积百分比较健康受试者均有变化, 表明肺癌患者血清中生化物质的结构或含量发生了改变。
 | 图2 健康者(a)和肺癌患者(b)的拉曼光谱在1 800~800 cm-1范围内的曲线分峰拟合分析Fig.2 Curve fitting analysis of Raman spectra in the range of 1 800~800 cm-1 in healthy subjects (a) and lung cancer patients (b) |
| 表1 健康受试者和肺癌患者血清拉曼光谱叠加带的子峰位置和面积百分比 Table 1 Sub-peaks location and area percentage of serum Raman spectral over lappedband in healthy subjects and lung cancer patients |
将数据集划分为两个互斥的集合(训练集S和测试集T)。 用S训练分类模型, 用T评估模型分类效果。 当机器学习的数据被分割时, 70%~80%数据应用于训练, 20%~30%数据应用于测试[23]。 Kennard-Stone(K-S算法)是把所有的样本都看作训练集候选样本, 依次从中挑选样本进训练集的样本划分算法[24]。 本研究依据K-S算法将数据集按照4∶ 1的比例划分为训练集和测试集, 测试集中肺癌患者17例, 健康受试者35例。 首先采用PCA降低光谱数据的维度, 提取显著特征变量。 PC1和PC2累积贡献率已达95.07%, 说明前两个主成分已经包含了光谱的重要信息。 将提取的特征变量输入算法中建立分类模型。 最后采用“ 留一交叉验证法” (leave-one-out cross-validation, LOOCV)评估模型的预测性能。
支持向量机( SVM)是有监督的分类器, 可用于二分类和多分类, 在解决非线性和高维模式识别中具有特有的优势。 SVM模型中的核函数可以将低维的特征向量映射到高维的特征空间中, 使得原本线性不可分的数据在高维度的空间中线性可分[25]。 分别选取Liner、 RBF和Polynomial作潍SVM模型的核函数, 对比发现RBF的分类效果最好, 本研究中SVM模型使用参数为γ 的高斯径相基函数(RBF)作为核函数, 其表达式为式(8)
最优判别函数为式(9)
式(9)中, sgn为阶跃函数, x为输入的光谱特征向量, xi为输出的第i个支持向量。 经过多次实验表明, 当C∈ (22, 23), γ ∈ (2-2, 1)时模型的训练精度较高, 在此范围内确定超参数。 当误差惩罚因子C=7, 核函数参数γ =0.6, 模型分类性能最好, 测试集的准确率达到98%, 灵敏度和特异性分别为94.44%和100%, F1分数为97.14%, 说明模型的性能较优。 图3是SVM模型在测试集中的分类结果。 决策边界(通过等高线来显示)是一个超平面, 将基础向量空间划分为两个集合。 决策边界一侧的所有样本归属于一个类, 另一侧的所有样本归属为另一个类。 等高线可以帮助理解分类器的性能和边界的形状, 以及评估分类器的准确性和泛化能力。 深色虚线是肺癌患者和健康者之间的决策边界, 浅色虚线是不同肺癌患者之间或不同健康者之间的决策边界。 深色虚线反映出测试集中有1例健康受试者被误判为患病。
 | 图3 SVM在测试集中的分类结果 H: 健康受试者; F: 肺癌患者; × : 分类错误的样本Fig.3 The SVM algorithm of classification results in test set H: Healthy subjects; F: Lung cancer patients; × : The misclassified sample |
受试者工作曲线(receiver operating characteristic curve, ROC)反映敏感性和特异性之间的关系, 它的横轴代表假阳性率, 纵轴代表真阳性率。 曲线下的面积被称为AUC(aera under curve), 反映模型的预测性能。 AUC取值在0和1之间, 越接近1, 说明模型分类性能越好。 本研究中, 用9折交叉验证法绘制SVM模型的ROC曲线。 9次验证中, AUC值最低0.84, 最高0.99。 为了客观评价SVM分类器的预测性能, 取9次验证结果的平均值作为该模型的AUC值, 其值为0.94, 说明SVM分类器的预测性能较好, 可用于肺癌的诊断。 图4中10条ROC曲线分别对应9折交叉验证的结果和验证结果的平均值。
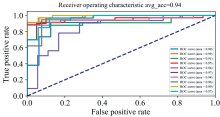 | 图4 SVM的9折交叉验证结果的ROC曲线 area=0.94代表9折交叉验证结果的平均值Fig.4 ROC curve of the SVM algorithm with 9-fold cross-validation results area=0.94 represents the average of the 9-fold cross-validation results |
k近邻(KNN)算法的分类工作机制是通过使用某种距离度量(例如欧氏距离、 余弦距离、 球面距离等)寻找训练集中与测试样本最接近的k个训练样本, 基于这k个最近的样本进行预测分类。 k值的选择对算法的结果影响较大[26]。 k值取太小, 容易发生过拟合。 k值取得太大, 会导致分类模糊(欠拟合)。 本研究选取欧氏距离作为距离的度量方法, 采用交叉验证法来选取最优k值。 实验结果表明, 当k=15时, 分类效果最好, 测试集的准确率达96%, 灵敏度和特异性分别为89.47%和100%。
决策树(DT)的关键是如何选择最优划分属性, 信息熵是一种常用的度量样本集合纯度的指标[27]。 假设当前样本集D中第k类样本所占比例为Pk=(k=1, 2, …, |y|), 样本集D的信息熵可以定义为式(10)
信息熵值越小, 样本集和D的纯度就越高。 该模型对肺癌患者和健康受试者分类结果的特异性、 灵敏度和准确率分别为75%、 93.33%和86%。
随机森林(RF)算法是由多个决策树组成的分类器, 能够有效降低过拟合的风险, 具有较好的泛化能力。 算法由随机选择样本、 随机选取特征、 构建决策树和集成决策树四部分组成[28]。 该模型在区分肺癌患者和健康受试者时, 特异性、 灵敏度和准确率分别显示94.12%、 96.97%和96%。
逻辑回归分类(LRC)是一种线性分类算法, 常用于二分类问题。 其核心思想是通过一个激活函数(通常是sigmoid函数)将线性回归的输出映射到概率空间, 从而实现分类[29]。 sigmoid函数的数学表达式为
式(11)中, P(Y=1/X)表示给定输入特征X时, 数据点属于正类的概率, ω 是权重向量, b是偏置项。 逻辑回归模型的优点在于快速、 计算效率高、 可解释性强。 本研究中测试集分类结果的特异性、 灵敏度和准确率分别为88.24%、 93.94%和92%。
贝叶斯分类(Bayes)是以贝叶斯定理为基础的统计分类方法。 假设特征之间是独立的, 并且通过计算后验概率来进行分类。 该算法简单且易于理解, 而是它的一个主要限制是假设特征之间是独立的, 与实际应用中要解决的问题不成立, 存在一定的局限性[30, 31]。 其对肺癌患者和健康受试者的分类特异性高达100%, 但是灵敏度和准确率较低, 分别为50%和66%。
所有分类模型评价指标见表2。 SVM模型的分类结果最好, 准确率最高(达到98%), 灵敏度和特异性分别为94.44%和100%, F1分数为97.14%。 Bayes的分类效果较差, 特异性高达100%, 但准确率和灵敏度仅为66%和50%。
| 表2 分类模型的评价指标 Table 2 Evaluation index of the classification models |
健康受试者和肺癌患者血清的拉曼光谱曲线分峰拟合结果显示, 肺癌患者在964、 1 005、 1 091、 1 158、 1 522和1 586 cm-1附近吸收子峰面积百分比较健康受试者有明显改变, 表明肺癌患者血清中类胡萝卜素、 核糖、 核酸、 氨基酸等生化物质发生一定改变。 在对血清的拉曼光谱进行PCA降维处理的基础上, 进一步采用SVM、 KNN、 RF、 DT、 LRC和Bayes算法建立分类模型对健康受试者和肺癌患者的光谱进行数据挖掘, 从而实现对肺癌患者与健康受试者的判别。 对比六个模型的评价指标, SVM模型的分类效果最好, 灵敏度、 特异性和准确率分别达到94.44%、 100%和98%。 其F1分数显示97.14%, 说明SVM模型的判别效果较好。 研究表明, 机器学习算法结合拉曼光谱对血清进行研究, 是肺癌诊断的一种潜在方法。 该方法具有创伤小、 便捷和灵敏度高等优点。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|


