作者简介: 孙浩然, 1992年生, 吉林交通职业技术学院讲师 e-mail: 1515754043@qq.com
肉类是人类饮食中重要的蛋白质和营养物质来源, 而肉类食品掺假问题已成为我国食品安全领域的重大问题, 针对传统肉类食品检测方法存在操作复杂、 程序耗时、 设备造价高昂的问题, 提出利用激光诱导击穿光谱技术(LIBS)结合深度学习网络模型的方法对多品种肉类组织进行快速检测与分类识别。 通过LIBS技术分别采集了牛肉、 羊肉、 猪肉3种肉类光谱数据, 选取5种元素9个波段谱线作为分析谱线及模型输入量进行建模识别, 设计了1种以ResNet18为主干网络, 同时设计3种机器学习模型, 对光谱数据进行建模识别。 结果表明, 深度学习网络模型识别能力最优, 精确度达到了98.1%。 120组光谱数据中, 牛肉、 羊肉、 猪肉光谱数据分别有117、 119、 117组光谱数据识别正确, 在与同为深度学习网络模型的横向对比中, ResNet18网络模型对肉类光谱数据的识别能力优于GoogLeNet、 Vgg16、 ResNet50的3种深度学习模型, 在此基础上通过重新采集的数据对模型泛化性进行验证, 精度达到98.9%, 说明模型能够在较高识别精度的基础上, 保持较强的跨数据集识别能力, 具有良好的泛化性与一致性。 以上研究表明, 通过LIBS与卷积神经网络结合的方式能够在多品种肉类分类识别任务中提供不同品种肉类客观量化差异信息, 并具有快速和原位诊断不同种类肉类组织的潜力。
Meat is an important source of protein and nutrients in the human diet, and the adulteration of meat has become a major problem in the field of food safety in China. To address the problems of complex operations, time-consuming procedures, and high equipment costs in traditional meat detection methods, this paper proposes using Laser-induced Breakdown Spectroscopy (LIBS) combined with a deep learning network to detect and classify multi-variety meat tissues quickly. Spectral data for beef, mutton, and pork were collected using LIBS. Nine spectral lines of five elements were selected as the analysis spectral lines and model input for modeling and recognition. A ResNet18 backbone network was designed, and three machine learning models were designed to model and recognize the spectral data.The results show that the deep learning network achieves the best recognition performance, with an accuracy of 98.1%. Among the 120 groups of spectral data, 117, 119, and 117 groups of beef, mutton, and pork spectral data were identified correctly, respectively. In the horizontal comparison using the same deep learning model, the ResNet18 model was superior to the three deep learning models, GoogLeNet, Vgg16, and ResNet50, in the recognition of meat spectral data. On this basis, the model's generalization was verified using re-collected data, and the accuracy reached 98.9%, indicating that the model maintains strong cross-data-set recognition ability. It has good generalization and consistency. The above research shows that the combination of LIBS and a convolutional neural network can provide objective, quantitative information on differences between meat varieties in multi-variety meat classification and recognition tasks and has the potential to quickly and in situ diagnose different types of meat tissue.
肉类在人类日常饮食中占据着不可或缺的地位, 它不仅为人体提供丰富的营养, 而且是全球范围内重要的食品资源[1]。 在当前社会背景下, 肉类食品的消费量持续增长, 消费者不仅追求肉类的口感和美味, 更关注其背后的安全与健康。 目前肉类食品掺假已成为我国食品安全领域中的重大问题, 某些不良商家绕过必要的检验和检疫程序, 用相对便宜的肉类代替较贵的肉类出售, 这一现象不仅增加了肉类携带有害细菌、 病毒、 以及过敏原的可能性, 还会破坏消费者对食品安全的信心, 导致正规企业连带受损, 使劣质肉流入市场引发连锁反应, 影响整个供应链的信誉, 甚至导致出口贸易壁垒, 如2013年欧洲“ 马肉冒充牛肉” 丑闻导致多国召回产品, 行业损失超数十亿欧元。 此外, 未经声明的跨物种掺假, 如猪肉混入清真食品, 可能违反宗教规范, 引发社会矛盾[2, 3, 4]。 因此迫切需要对肉类食品进行快速准确分析与识别, 以促进肉类市场的标准化, 确保食品安全。 针对目前食品安全领域中的肉类掺假问题, 多种技术已被开发并应用于识别肉类品种, 传统的肉类检测技术包括毛细管凝胶电泳、 聚合酶链反应、 气相色谱质谱、 DNA条形码等方法[5, 6, 7, 8, 9, 10], 然而以上方法存在操作复杂、 程序耗时、 设备造价高昂等缺点, 且需要专业的技术人员进行操作, 检测结果存在一定程度上的主观性, 以上问题限制了肉类检测技术在食品安全领域中的应用。 研究表明, 不同肉质中元素含量存在差异, 如钙、 镁、 钾、 钠是生命体正常运作所必需的营养物质, 从肉质的角度来看, 钙、 镁、 钾和钠元素含量与肉质嫩度有关。 因此可将肉类中元素含量差异映射到具体数据中, 从而对不同肉类进行有效识别。
激光诱导击穿光谱技术(laser-induced breakdown spectroscopy, LIBS)是一种基于样本内部元素含量检测的光谱分析技术, 以其快速响应、 无需复杂样品预处理以及实时分析的显著优势, 在多个领域如水质监测、 金属加工、 土壤分析和航天科技中得到了广泛应用[11, 12, 13, 14, 15]。 在生物组织的研究领域, Bilge等[16]利用LIBS技术对各类肉质进行分析, 通过主成分分析实现了对肉品的定性鉴别, 准确率高达83.37%。 Kanawade等[17]运用LIBS与线性判别算法对猪肉的不同组织成分进行分类识别, 检测精度达到90%。 Casado-Gavalda等[18]通过LIBS对掺入牛肝的碎牛肉中铜含量进行分析, 并采用偏最小二乘回归建立了校正和验证模型, RMSEP达到了36.8 ppm。 Velioglu等[19]采用LIBS技术对牛肉和内脏样品进行鉴别, 结合偏最小二乘法确定掺假比例, 检出限达到了3.8%。 以上研究突出了LIBS技术在肉类检测与分析领域的潜力, 表征了LIBS技术在肉类食品检测中能够提供快速和准确检测结果的能力, 在生物组织光谱数据分析领域, 化学计量法是当前研究的主流方法。 然而生物组织光谱数据通常具有高维特征性, 并伴随特征冗余以及特征相关性, 使机器学习模型在进行数据处理过程中存在数据挖掘困难, 特征提取能力差等问题, 从而导致模型识别精度降低。 与机器学习模型相比, 卷积神经网络模型通过卷积操作能够将数据信息映射到深度网络结构中, 并通过优化网络深度与宽度来提升模型特征提取能力[20]。 因此本文结合LIBS与卷积神经网络, 对3种常见的肉类样品(猪肉、 牛肉、 羊肉)进行检测, 利用肉类元素差异和深度学习网络模型特征提取产生肉类样本信息, 在解释其固有特性的同时帮助降低肉类光谱数据检测的复杂性, 并建立3种机器学习模型进行比对, 通过10倍交叉验证的方式获得模型精确度、 F1-score、 Recall、 Kappa等参数对模型进行评估, 最终提高对3种肉类检测的识别精度。
实验选取长春市前进大街欧亚超市采购的新鲜肉类, 具体信息如表1所示。
| 表1 样品制备信息 Table 1 Sample preparation information |
将9种新鲜肉类切成尺寸为4 cm× 2 cm× 1 cm的切片, 如图1所示。 每种肉类制备10片样品, 每组激光烧蚀尽量作用于样品中心区域, 同时烧蚀点之间保持一定距离, 平均15束脉冲收集1幅光谱, 每片样品采集12组光谱数据, 每种肉类共120组光谱数据, 共360组光谱数据。
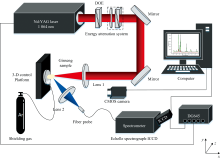
实验装置结构如图2所示。 光源为Nd:YAG激光器(SLII-10, Continuum, 1 064 nm), 激光能量、 脉宽、 重复频率分别设置为65 mJ、 10 ns、 10 Hz。 通过中阶梯光栅光谱仪(Me5000, Andor)采集等离子体, 采集范围为200~1 100 nm, 共可采集27 370个波段数量, 分辨率< ± 0.05 nm。 实验温度22 ℃, 相对湿度27%。 通过三维平移台(M-410.DG, PI)对样品位置进行调整, 以避免存在烧蚀重复点。
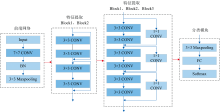
为了在LIBS特征光谱分析中保留尽可能多的原始信息并消除噪声干扰, 首先对光谱数据进行降噪处理。 结合US NIST原子光谱数据库及LIBS特征光谱, 选取元素谱线。 选择ResNet18网络模型作为主干模型进行建模识别, 在利用模型残差边有效传输原始数据特征的同时剔除冗余信息。 网络具体架构如图3所示, 主要由3个Block组成, 其中1个Block带有1× 1残差边。 模型Learning rate设置为0.000 1, epoch设置为30轮。
除构建深度学习模型外, 本文还构建了3种机器学习模型进行比对, 结合主成分分析提取关键信息, 减少数据冗余, 并将光谱数据转化为低维度空间特征, 为机器学习模型提供更清晰、 简洁的数据输入, 从而帮助模型训练和预测。 模型评估方面, 选择10倍交叉验证的方法, 将机器学习模型的学习程度达到最大化, 以解决光谱信息分布不均, 导致模型识别精度受原始数据集选择的影响。 选择Accuracy、 Kappa、 Precision、 Recall、 F-score、 MCC(Matthews correlation coefficient)来对模型整体识别能力进行评估, 分别表示为
$\text { Accuracy }=\frac{T P+T N}{T P+F P+F N+T N~~~} $(1)
$\text { Precision }=\frac{T P}{T P+F P~~~}$(2)
$\text { Recall }=\frac{T P}{T P+F N~~~}$(3)
$\mathrm{~F} \text {-Measure }=\frac{2 \times \text { Recall } \text { × Precision }~~~~~~}{\text { Recall }+ \text { Precision }~~~}$(4)
$\mathrm{MCC}=\frac{T P \times T N-F P \times F N}{\sqrt{(T P+F P)(T P+F N)(T N+F P)(T N+F N)}~~~~~~}$(5)
式中, TP、 TN、 FN、 FP分别表示真阳性、 真阴性、 假阴性、 假阳性。 Kappa作为衡量数据与模型一致性指标, 表示为
$\mathrm{Kappa}=\frac{~~\mathrm{Po}-\mathrm{Pe}~~}{1-\mathrm{Pe}~}$(6)
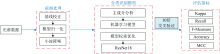
式(6)中, Po为模型整体分类精度, Pe为实际与预测数量的乘积之和除以样本总数的平方。 论文整体技术路线如图4所示。
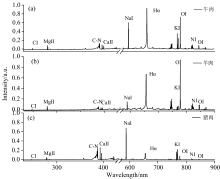
图5显示了三种肉类LIBS特征光谱。 从图中可以看出, 3种肉类组织中存在多种金属元素, 如在279.8、 393.4、 588.99和766.5 nm处存在Mg、 Ca、 Na、 K元素谱线。 此外非金属元素C、 H、 O也可以在LIBS光谱中发现, 从图中可以看出不同肉类样品中各元素谱线强度存在一定差异, 这些元素谱线强度的差异实际上反映了它们在不同种类肉类组织中的含量差异, 为识别3种肉类组织的光谱数据提供了基础。
在选择谱线时依据以下原则: (1)特征谱线激发概率大、 无干扰谱线; (2)避免相邻且无法分辨的谱线; (3)避免具有自吸收或自反转特性特征谱线。 可以看出特征光谱存在Mg、 Ca、 Na、 K等元素谱线, 从肉质角度来看, 镁、 钾和钠的含量与肉质的嫩度有关, 例如镁在神经肌肉活动中起着重要作用, 有助于肌肉放松, 减少压力对肉质的影响。 而钾元素是肌肉收缩和神经冲动的必需元素, 在细胞内部, 钾元素与钠元素一起用于维持细胞内液体的适当平衡。 通过图谱分析发现在猪肉组织光谱图中Ca Ⅱ 393.4 nm谱线强度显著高于牛肉和羊肉; 而羊肉光谱图中Na Ⅰ 588.99、 Na Ⅰ 589.59 nm谱线强度则低于其他两种肉类。 此外, 3种肉类中的K Ⅰ 466.49 nm元素谱线也呈现出不同程度的差异, 其中Ca、 Mg、 K和Na含量差异可能与饮食和管理的差异以及土壤特征如有机质水平、 矿物质组成有关, 因此本文将Ca、 Na、 K元素作为分析谱线。 C— N谱带强度能够有效反映不同肉类中蛋白质、 糖、 脂肪等有机质含量的差异, 这些差异是生物体生理代谢活动的反映, 因此C— N谱带强度可以被视为识别不同品种肉类的一个可靠指标。 最终本文选择了5种元素9个波段谱线进行分析, 如表2所示。
| 表2 特征谱线选取 Table 2 Library of selected emission lines |
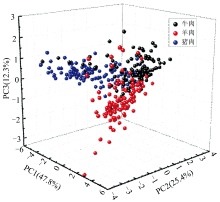
首先通过PCA对3种肉类光谱数据进行处理, 剔除相关性较高的冗余特征, 并将降维处理后的数据作为机器学习模型输入变量, 从而达到精简特征, 降低维度的效果。 图6为降维后光谱数据矩阵各主成分贡献率及其累积贡献率, 其中前3个主成分可占总方差的85.5%。
图7为基于前三个主成分构建的三维散点图, 可以看出3种肉类组织光谱数据点在图中呈现出严重的重叠现象, 并且数据点分布较为分散。 这表明传统聚类分析方法未能有效区分不同肉类的光谱特征, 因此需结合机器学习算法模型对3种肉类组织的光谱数据信息进行深入分析, 从而更有效地挖掘光谱数据中的细微差异, 实现精确分类识别。
首先通过Accuracy、 F1-score、 Recall等参数对机器学习模型的整体识别能力进行深入分析, 结果如表3所示。 3种机器学习模型在识别不同肉类组织方面均表现出较高的识别精度, 均在90%以上。 以SVM算法为例, 其对牛肉、 羊肉、 猪肉的光谱数据识别精度分别高达92.5%、 96.7%、 94.2%, F1-score也分别达到了94.5%、 95.1%、 93.8%。 这一现象表明, 通过PCA降维处理后的光谱数据, 与机器学习算法模型相结合, 能够初步实现对3种常见肉类的分类识别。 然而模型识别能力并不均衡, 例如KNN模型对牛肉与羊肉的识别精度分别达到95.8%与94.2%, 而对猪肉的识别精度仅为90.8%, 同时SVM和BaggingTree模型同样存在此类问题, 说明以机器学习算法作为肉类识别手段的泛化性不强, 容易对单一品种肉类造成混淆。
| 表3 机器学习模型Accuracy、 F1-score、 Recall参数 Table 3 The Accuracy, F1-score, Recall of machine learning models |
针对机器学习模型对多品种肉类识别能力不均衡的问题, 本文构建了以ResNet18为主干网络的深度学习模型对3种肉类光谱数据进行分析, 一方面提高模型识别精度, 另一方面提高模型跨品种数据集的模型泛化能力, 从而达到对多品种肉类精确识别的目的。 结果如表4所示。 模型对3种肉类光谱数据的识别精度分别达到了97.5%、 99.2%以及97.5%; F1-score分别达到了98.7%、 97.9%及97.5%, 模型整体识别能力明显高于其他3种机器学习模型算法, 同时模型对3种肉类光谱数据识别能力较为均衡。 表5显示了4种模型的整体识别精确度、 Kappa系数以及MCC系数, 体现了模型整体识别能力、 识别泛化性以及一致性。 4种模型的整体识别精度分别达到了94.4%、 93.6%、 95.0%以及98.1%。 可以看出ResNet18网络模型在保证模型泛化性的同时, 其整体识别精度同样高于机器学习模型, 此外模型Kappa及MCC同样高于机器学习模型, 这一现象说明了以深度学习为思想的网络架构在多品种肉类识别场景下, 其模型样品内部数据挖掘和特征学习方面明显强于机器学习模型, 能够保证多品种肉类识别任务中的模型泛化性及识别精度。
| 表4 ResNet18模型Accuracy、 F1-score、 Recall参数 Table 4 The Accuracy, F1-score, Recall of ResNet18 |
| 表5 ResNet18模型与机器学习模型Accuracy、 Kappa、 MCC参数 Table 5 The Accuracy, Kappa, MCC of ResNet18 and machine learning models |
为进一步直观展示ResNet18网络模型性能, 通过混淆矩阵对模型的识别结果进行展示, 结果如图8所示。 4种模型中, KNN模型识别效果最低, 在360组数据中, 共有23组光谱数据识别错误, 说明以计算距离为函数思想的机器学习模型, 与平面分割(SVM)或集成思想(Bagging Tree)相比, 并不适用于高维度, 且特异性较高肉类组织光谱数据。 相比之下, ResNet18网络模型在进行多品种肉类识别任务中精度明显提高, 牛肉、 羊肉、 猪肉光谱数据分别有117、 119、 117组光谱数据识别正确, 这表明深度学习网络模型在将LIBS光谱数据映射到网络深层架构中后, 能够通过卷积以及池化的方式对特异性较强、 复杂度较高的肉类组织LIBS光谱数据信息进行充分挖掘, 从而获得更好的局部特征提取能力以及跨数据集泛化能力。
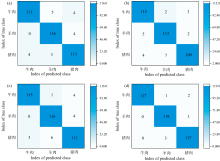 | 图8 模型混淆矩阵 (a): SVM; (b): KNN; (c): Bagging Tree; (d): ResNet18Fig.8 Confusion Matrix of 4 models (a): SVM; (b): KNN; (c): Bagging Tree; (d): ResNet18 |
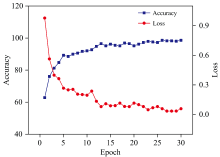
图9展示了ResNet18模型在训练过程中的可视化过程。 在30次训练迭代过程中, 模型Loss值在前10轮迭代过程中呈逐步下降的趋势, 并在20轮之后损失函数逐渐稳定并趋近于0。 模型精确度在13轮前逐步上升, 在13~20轮区间内存在一定程度的波动现象, 并在20轮之后趋于稳定, 表明在多品种肉类识别任务中, 在对3种LIBS光谱数据分析过程中, 以ResNet18为主干网络的网络模型具有优秀的学习能力与稳定性。
表6显示了GoogLeNet、 Vgg16、 ResNet50三种深度学习模型与本文方法对比结果, 从表中可以看出, GoogLeNet、 Vgg16、 ResNet50精确度分别为96.4%、 96.7%、 97.5%, 通过横向比较, 表明本文方法对肉类光谱数据识别的能力优于其他深度学习模型。
| 表6 深度学习模型Accuracy、 Kappa、 MCC参数 Table 6 The Accuracy, Kappa, MCC of ResNet18 |
为了验证模型跨样本集、 跨设备、 跨环境的泛化能力, 本文采用了不同光谱仪, 激光器对3种肉类样品进行重新采集, 其中猪肉、 牛肉、 羊肉样品分别采集120组LIBS特征光谱, 共360幅特征光谱, 并通过ResNet18模型对重新采集的数据进行建模识别, 结果如表7所示, 模型整体识别精度达到98.9%, 牛肉、 羊肉、 猪肉组织模型精确度分别为97.5%、 100%、 99.2%, 从结果可以看出, 本文所建立的ResNet18模型具有良好的跨样本集、 跨设备、 跨环境泛化识别能力。
| 表7 ResNet18模型跨数据集Accuracy、 F-Score、 Recall参数 Table 7 The Accuracy, F-Score, Recall of ResNet18 cross-dataset |
由于LIBS在探测元素方面的具有强大的灵敏性, 同时不同种类肉类组织中存在着明显的元素差异, 而卷积神经网络能够对肉类LIBS光谱数据中的非线性特征进行深度挖掘, 因此本文搭建了LIBS-ResNet18杂化体系, 将元素信息嵌入到网络的深层架构中, 作为神经网络的输入, 对整个肉类组织的光谱信息进行深入挖掘, 以便快速提取肉类组织内部差异信息, 本文的研究目标在于首次评估LIBS技术与深度学习结合, 并在快速原位识别肉类物种方面的潜力。 通过LIBS技术可以检测到样品元素组成差异, 而深度学习网络模型能够对内部元素的光谱数据特征进行有效挖掘, 将LIBS与深度学习技术联用, 就能够在元素层次上实现对复杂物质的快速无损识别和分类。
通过LIBS技术收集了牛肉、 羊肉、 猪肉三种肉类的光谱信息, 在此基础上建立了3种机器学习模型和1种深度学习网络模型, 采用10倍交叉验证对模型进行评估, 数据分析表明, 以ResNet18为主干网络的深度学习网络模型在分类精确度、 模型一致性、 泛化性上均优于传统机器学习模型, 模型识别精度可达到98.1%。 在LIBS-ResNet18模型中, 元素差异可以从LIBS光谱中确定, 肉类样品内部的局部复杂关键特征可以通过ResNet18模型进行有效挖掘, 利用两种技术的互补性的组合系统可以为识别生物组织特征提供有价值的信息, 可作为提高肉类食品安全检测精度的有效手段。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|