作者简介: 黄莲飞,女, 1999年生,贵阳学院食品科学与工程学院硕士研究生 e-mail: 18185950411@163.com
高光谱成像技术凭借其能够获取样品连续且丰富光谱信息、 有效反映内部成分特征的独特优势, 为果蔬品质关键理化指标的快速无损检测提供了新的技术路径。 基于此, 以番茄为研究对象, 聚焦其关键理化指标——包括可溶性固形物(SSC)、 番茄红素含量和维生素C含量, 探索其快速无损预测方法。 为提高预测模型的准确性与稳健性, 首先对高光谱数据采用标准正态变换(SNV)进行预处理, 以削弱散射和光谱漂移的干扰, 随后基于遗传算法(GA)筛选有效特征波段。 进一步分别构建了反向传播-人工神经网络(BP-ANN)与极限学习机(ELM)预测模型, 对比其在不同品质指标预测中的性能差异。 结果表明, ELM在番茄红素、 SSC及维生素C的预测中均优于BP模型。 与BP模型相比, ELM的预测集相关系数(
Hyperspectral imaging technology, with its unique advantages of acquiring continuous and abundant spectral information of samples and effectively reflecting internal compositional characteristics, provides a new technical pathway for the rapid and non-destructive detection of key physicochemical indicators of fruits and vegetables. Based on this, this study focuses on tomatoes as the research object, targeting their key physicochemical indicators—including Soluble Solids Content (SSC), lycopene content, and vitamin C content—to explore rapid and non-destructive prediction methods for these indicators. To enhance the accuracy and robustness of the prediction model, the hyperspectral data were first preprocessed using the Standard Normal Variate (SNV) transformation to mitigate the interference of scattering and spectral drift, followed by the selection of effective feature bands based on the Genetic Algorithm (GA). Further, Backpropagation Neural Network (BP) and Extreme Learning Machine (ELM) prediction models were respectively constructed to compare their performance differences in predicting various quality indicators. The results showed that ELM outperformed the BP model in predicting lycopene, SSC, and vitamin C. Compared to the BP model, the correlation Coefficient of the Prediction set (
番茄作为全球重要的经济作物, 其品质评价主要为可溶性固形物含量(soluble solids content, SSC, 反映甜度)、 番茄红素(主要的抗氧化成分)及维生素C(决定营养价值与保鲜能力)等关键理化指标[1]。 然而, 目前传统检测果蔬品质理化指标的方法, 如高效液相色谱法测定番茄红素、 折射仪法测SSC等, 通常具有破坏性、 检测周期长且成本较高, 难以满足现代农业生产实时、 快速、 大规模无损检测的迫切需求[2]。 近年来, 高光谱成像技术(hyperspectral imaging, HSI)凭借其“ 图谱合一” 的优势, 能够同步获取样本的空间分布信息与连续、 高维的光谱数据, 已成为农产品品质无损检测的研究热点。 与传统检测技术相比, 高光谱成像不仅实现了对样品的快速、 无损检测, 同时具备对内部理化属性预测的潜力, 为农产品品质评价提供了更加高效、 智能的技术路径[3]。 针对番茄品质快速检测的需求, 高光谱成像技术为实现番茄关键品质指标(如SSC、 番茄红素及维生素C含量)的实时、 无损预测提供了新的技术手段, 具有广阔的应用前景。
然而, 高光谱数据通常具有高维度、 波段间强相关性以及显著的噪声干扰(如基线漂移与散射效应), 这显著增加了建模复杂度, 并在一定程度上限制了预测模型的准确性与稳定性[4]。 为减少噪声干扰并提升模型性能, 通常根据数据特性选择合适的光谱预处理方法, 包括多元散射校正(multiplicative scatter correction, MSC)、 标准正态变量变换(standard normal variate, SNV)、 去趋势以及一阶/二阶导数等[5]。 本研究中, 由于高光谱数据主要受光程变化及样品表面不均匀性影响, 产生了显著的散射效应, 因此采用 SNV 进行散射校正, 以有效抑制基线漂移并提升数据质量[6]。
此外, 光谱特征变量提取方法种类丰富, 包括竞争性自适应重加权算法、 连续投影算法、 随机森林(random forest, RF)以及基于优化算法的特征筛选方法等[7]。 在这些方法中, 遗传算法(genetic algorithm, GA)作为最经典且应用最广泛的基于优化算法的变量选择方法, 能够通过模拟自然选择与遗传机制实现全局搜索, 在处理高维、 高相关性光谱数据时具有较强的鲁棒性和优化能力[8]。 故选用GA进行特征波段选择, 旨在从较大波段集合中高效筛选出对预测任务贡献显著的特征, 以降低数据冗余、 简化模型结构并减少过拟合风险。
关于建模, 传统的线性方法如偏最小二乘回归(partial least squares regression, PLSR)在处理高光谱数据时, 因其假设变量间存在线性关系, 往往难以捕捉光谱数据中存在的复杂非线性特征, 导致模型的预测性能受限[9]。 为克服这一不足, 近年来非线性建模方法受到广泛关注。 极限学习机(extreme learning machine, ELM)作为一种单隐层前馈神经网络的训练方法, 通过随机初始化隐藏层权重和偏置, 进而采用最小二乘法直接求解输出权重, 实现非迭代训练[10]。 该方法自提出以来已被广泛应用于模式识别与回归分析领域, 近年来也逐步引入光谱分析研究, 用以提高光谱建模的效率与预测精度[11]。 相比传统的反向传播-人工神经网络(back-propagation artificial neural network, BP-ANN), ELM显著简化了训练过程, 提高了训练速度, 且在一定程度上提升了模型的泛化能力, 是农业高光谱数据分析中的重要方法之一[12]。 然而, 现有研究中针对ELM与传统BP-ANN在番茄品质预测中的系统性对比仍较为有限, 尤其缺乏基于优化预处理与特征选择协同建模的深入探讨。 因此, 进一步开展高光谱数据预处理、 特征选择与非线性建模方法的集成优化研究, 对提升番茄品质快速无损检测的预测性能具有重要意义。
光谱技术因其快速、 无损、 可同时检测多种品质参数的优势, 在果蔬品质检测中表现出广阔应用前景。 已有研究表明, 高光谱成像技术结合机器学习方法, 能够有效预测番茄的SSC、 硬度及部分营养成分。 例如: Shao等[1]利用可见-近红外高光谱成像开发了番茄综合品质指数(comprehensive quality index, CQI), 实现了对多项品质参数的稳健预测。 Polder等[13]通过高光谱成像结合PLSR, 实现了番茄SSC和硬度的无损检测。 Xiang等[4]提出一种基于一维卷积神经网络(one-dimensional convolutional neural network, 1D-CNN)与长短期记忆网络(long short-term memory, LSTM)融合的高光谱成像(400~1 700 nm)无损检测方法, 通过联合提取空间-光谱特征, 实现了番茄SSC和硬度的同步预测。 Duckena等[14]采用光谱技术结合PLSR, 对80个品种番茄的内部品质进行快速无损检测, 建立的模型对番茄红素和干物质含量的预测准确性达到R2=0.90, 且对风味指数、 类黄酮、 β -胡萝卜素及总酚也具有较好预测能力。 Rahman等[15]利用高光谱成像技术预测番茄中的水分含量、 pH值和SSC含量。 研究发现, 基于一阶导数预处理光谱的PLSR回归模型在预测这些参数方面表现出良好的效果, 相关系数分别为0.81、 0.69和0.74。 张若宇[16]等利用高光谱成像技术检测番茄SSC含量。 通过对比不同姿态下的检测效果, 发现组合姿态C1C2C3在3个波段区域的检测效果优于单一姿态, 其验证集均方根误差(root mean square error of prediction, RMSEP)分别为0.299%、 0.133%、 0.151%, 相关系数(Rp)分别为0.42、 0.89、 0.90。 尽管已有研究在番茄品质检测方面取得了重要进展, 但仍存在以下不足:
(1) 检测指标单一: 多数研究集中在SSC或硬度等单一物理参数, 缺乏针对多种营养品质参数(如番茄红素、 SSC、 维生素C)的协同预测;
(2) 特征波段选择不足: 现有工作多依赖全波段建模或简单波段选择, 导致模型复杂度高、 实时应用可行性不足;
(3) 建模方法优化不足: 预处理、 特征提取与机器学习模型优化往往分散进行, 缺乏系统化、 协同优化的建模流程;
(4) 泛化性能验证不足: 少有研究在多指标预测下系统评估模型的稳定性与实际应用潜力。
针对上述问题, 本研究提出了基于“ SNV预处理-GA特征选择-ELM模型优化” 的建模协同优化策略, 将其应用于番茄多指标(番茄红素、 SSC、 维生素 C)快速无损检测, 旨在: (1)验证SNV对番茄高光谱数据散射噪声的校正效果; (2)通过GA精准筛选与各指标显著相关的特征波段, 降低模型复杂度、 提升预测精度; (3)对比ELM与BP-ANN在番茄红素、 SSC和维生素C预测中的建模性能差异; (4)利用关键定量指标, 如决定系数(coefficient of determination, R2)、 均方根误差(root mean square error, RMSE)与剩余预测偏差(residual predictive deviation, RPD)等对模型的准确性和稳定性进行定量评估, 同时, 采用散点图对模型预测结果与真实观测值的对应关系进行可视化分析, 辅助理解模型拟合效果, 以全面论证模型的预测性能和泛化能力。 研究结果有望为高光谱技术与机器学习在智慧农业中的深度融合提供了理论依据与技术参考以推动果蔬品质快速无损检测技术的发展与应用。
选取云南省主栽的两个市售番茄品种“ 水果番茄” 与“ 大红番茄” , 于2024年采收季采摘成熟果实各120个(共240个); 采收标准为果实表面≥ 90%面积转红[17]。 剔除损伤及病虫害果后, 用无菌软布擦净表面杂质, 置于实验室[温度(20± 1) ℃, 湿度80%± 5%]中静置12 h以平衡样本状态。 后续高光谱采集均在相同环境下进行。
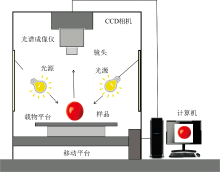
高光谱图像采集采用“ GaiaField” 高光谱成像系统(GaiaField-F-V10, 双利合谱科技有限公司), 该系统主要由高光谱成像仪、 镜头, 光源、 样品移动平台以及配备高光谱数据采集软件SpecView的专用计算机等部件组成。 成像系统的光谱采集波长范围为400~1 000 nm, 包含256个连续波段, 能够全面覆盖可见光近红外波段的光谱范围, 光谱分辨率为0.2 nm。 设备结构及样品放置示意见图1, 采集的数据可同时提供光谱信息与图像信息, 实现光谱与空间的有机融合。
为获取清晰且不失真的高光谱图像, 实验过程中, 高光谱图像采集系统的各项关键参数进行了统一设置: 曝光时间设定为15 ms, 移动平台的前进速度为1 cm· s-1, 回退速度为2 cm· s-1, 镜头与样品间的距离保持为20 cm。 在高光谱图像采集过程中, 受光敏单元自身响应差异、 暗电流以及偏置等因素的影响, 导致高光谱图像的输出强度不均匀, 从而影响后续特征提取与建模分析的准确性[18]。 为减少高光谱成像系统中暗电流等噪声对图像的干扰, 对采集图像进行了黑白校正, 将光强度数据转换为光反射率数据进而实施黑白校正, 黑白校正如式(1)所示。
式(1)中, I为校正后的反射率, B为原始高光谱图像, W为暗参考图像, R为白参考图像。 通过黑白校正处理, 有效消除了系统误差及环境光干扰, 为后续光谱分析提供了可靠的数据基础。
1.4.1 可溶性固形物(SSC)测定
高光谱图像采集完成后, 使用榨汁机对番茄样品进行充分榨碎处理。 将所得番茄浆液通过200目过滤袋过滤, 并收集滤得的番茄汁。 随后, 采用ATAGOPAL-α 折射仪(日本ATAGO公司)测定样品的SSC含量。 每个样品重复测量三次, 取平均值作为该样品的最终SSC测定结果。
1.4.2 维生素C含量测量
维生素C含量的测定参考Wu等[19]报道的方法。 采用2, 6-二氯靛酚滴定法测定维生素C的含量。 称取5 g番茄匀浆, 稀释在2%草酸溶液中, 使用标准化的2, 6-二氯靛酚溶液进行滴定, 直至样品颜色刚好褪色为终点。 根据式(2)计算样品的维生素C的含量。
式(2)中, V为滴定样品所消耗的2, 6-二氯靛酚钠溶液的体积(mL); T为1 mL 2, 6-二氯靛酚钠溶液相当于抗坏血酸的毫克数; m样品质量。
1.4.3 番茄红素的含量测量
番茄红素含量的测定参照Shao等[1]的方法。 以无水乙醇、 正己烷以及2%二氯甲烷混合溶液作为萃取剂, 对番茄匀浆进行番茄红素的提取。 提取完成后, 运用紫外分光光度计(型号: UV-2600, 日本岛津公司)测定所提取样品中的番茄红素含量。 在测定过程中, 以径长为1 cm的石英池作为比色容器, 同时选用正己烷和2%二氯甲烷的混合溶液作为空白对照调零。 记录番茄红素提取液在503 nm波长处的吸光度, 结合相关标准曲线进行含量计算, 最终实现番茄红素的定量分析。
使用ENVI 5.6软件(ResearchSystemsInc., Boulder, CO, USA)对每个样品的感兴趣区域(region of interest, ROI)进行手动选取, 并计算该区域的平均光谱值, 生成各样品的光谱数据矩阵。 为消除散射效应及样品表面不均匀性对光谱数据的干扰, 提升数据质量, 对原始光谱采用标准正态变量变换(SNV)进行预处理, SNV 预处理算法如式(3)所示:
式(3)中, xi第i个样本的光谱向量; ui该样本所有波段反射率均值; σ i该样本反射率标准差。
为降低高光谱数据的冗余性与建模复杂度, 利用遗传算法(GA)对SNV预处理后的光谱数据进行特征波段筛选。 GA通过模拟自然界生物进化过程选择、 交叉和变异等遗传操作, 迭代搜索最优特征, 以最大化预测模型的性能[7]。 特征波段的最优组合依据交叉验证所得的均方根误差(root mean square error of cross validation, RMSECV)最小原则进行确定。
选择反向传播-人工神经网络(BP-ANN)和极限学习机(ELM)作为预测模型的算法, 分别构建番茄理化指标的预测模型。 BP-ANN通过多层非线性结构深入挖掘光谱数据与目标指标之间的复杂映射关系。 相比之下, ELM随机生成隐藏层权重与偏置, 通过解析解直接计算输出权重, 大幅提升训练速度, 且具有较强的泛化能力[20]。
为全面评估模型的拟合效果与预斥能力, 采用决定系数(R2)、 均方根误差(RMSE)与剩余预测偏差(RPD)作为模型评价指标, 以全面评估模型的拟合效果与预测能力。 对应公式如式(4)— 式(6)所示。
$\mathrm{RMSE}=\sqrt{\frac{1}{n} \sum_{i=1}^{n}\left(y_{j}-\hat{y}_{j}\right)^{2}}$ (5)
式(4)— 式(6)中, n样本数; yi为理化指标的实际测量值;
为确保预测模型结果的可靠性评估具备充分的数据基础, 在建模分析前对番茄样品的品质指标进行了统计分析(表1)。 结果显示, 番茄红素含量范围为1.41~2.89 mg· (100 g)-1, SSC含量范围为4.71~7.36° Brix, 维生素C含量范围为18.69~30.14 mg· (100 g)-1。 各指标的标准差反映出样品间存在一定差异性, 可为后续建模分析提供必要的数据支撑。
| 表1 番茄品质指标含量分析 Table 1 Analysis of quality index content of tomato |
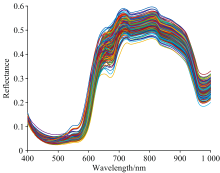
图2展示了所采集的番茄样品的原始光谱数据, 曲线整体呈现丰富且连续的光谱信息, 不同波长区域的反射率的变化特征与番茄品质指标(番茄红素、 SSC以及维生素C)等具有紧密的相关性。
其中, 番茄红素作为番茄中重要的类胡萝卜素, 其分子结构决定了其在可见光区(400~700 nm)光谱中的吸收特性。 筛选出的波长与番茄红素的电子跃迁吸收密切相关。 类胡萝卜素分子具有共轭双键体系, 在吸收特定波长的光能后, 会发生电子从基态到激发态的跃迁。 具体而言, Soret带和Q带吸收峰是类胡萝卜素光谱特征的典型代表。 Soret带通常位于紫外-可见光区的较短波长处, 具有较高的摩尔吸光系数, 而Q带则位于相对较长波长处, 吸收强度相对较弱。 在番茄红素的光谱分析中, 这些吸收峰的位置和强度能够反映其分子结构和含量的信息[21]。 因此, 在400~700 nm范围内筛选出的特征波段, 极有可能是对应于番茄红素分子中电子跃迁的关键波长, 通过该波段的光谱信息, 可有效预测番茄红素的含量和结构特征。
对于维生素C, 其抗氧化活性主要来源于其分子结构中的羟基(— OH)和羰基(C=O)基团。 SSC主要由糖类组成, 其分子结构仔富含的C— H和O— H化学键在红外光谱区域所变现出显著的振动吸收特性。 研究表明, 970~926 nm 波段与芳香族醚、 碳水化合物及多糖中的C— O— C伸缩振动相关, 855 nm处的特征峰可归因于醇基团的振动, 而833~794 nm波段则反映了芳香族C— O— H伸缩振动及芳香族醚/酚类中C— O— C的振动模式[22, 23]。 通过分析这些波段的光谱信息, 可以准确地反映番茄中维生素C和SSC的含量, 符合维生素C和SSC的光谱特性。 综上所述, 番茄样品在可见光与近红外区域所呈现的光谱特征, 与其主要品质指标(番茄红素、 SSC、 维生素C)具有良好的相关性。 本研究所提取的特征波段不仅符合化学机理, 也为后续预测模型的构建提供了坚实的数据基础。
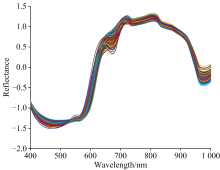
在光谱数据分析中, 光谱预处理对有效提取关键化学信息, 降低物理干扰及提升模型预测性能具有重要作用。 标准正态变换(SNV)作为一种常用的光谱预处理方法, 主要用于消除光谱数据中的基线漂移和物理散射效应的影响, 从而突出样品光谱中的化学成分信息。 图3给出了经过SNV处理后的光谱数据。
从图2未经处理的原始光谱数据图中可以观察到, 原始光谱存在明显的基线漂移现象, 这种现象主要源于样品的物理属性差异(如颗粒大小、 分布不均匀性等)以及测量过程中光程变化、 仪器噪声等外部因素等引起的[5]。 基线漂移不仅会掩盖光谱中的吸收峰和特征信息, 还会影响后续的特征提取分析和建模分析。 此外, 原始光谱曲线在不同样本之间存在较大离散性, 尽管这种差异部分反映了样品间的化学组成差异, 但也不可避免地混杂了散射效应和其他物理干扰, 增加了光谱解析和建模的复杂度。 相比之下, 图3给出了经过SNV处理后的光谱数据。 经过SNV预处理后, 光谱的基线明显得到了校正, 曲线整体趋于平稳, 不同样本之间的离散性显著减小, 光谱重叠度大幅提高。 以550 nm附近的吸收峰为例, 原始光谱中该吸收峰由于基线漂移影响, 形态模糊、 强度不稳定, 难以准确表征目标化学组分。 而经过SNV处理后, 吸收峰变得更加尖锐, 强度和位置更加明确, 从而能更准确地反映样本中特定化学成分的吸收特性。 此外, 在700~1 000 nm波段内的“ 红边” 区域, SNV处理也显著增强了光谱特征的表达能力, 使该区域的吸收特性更加清晰, 有助于后续对相关理化指标的精确建模。 综上所述, SNV预处理有效提升了光谱数据的信噪比和分析价值, 为后续特征波段提取及预测模型的构建奠定了良好基础。
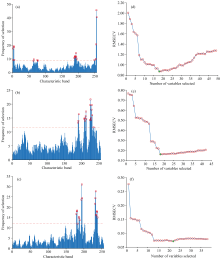
本文中采用遗传算法(GA)结合交叉验证均方根误差(RMSECV)对预处理后的全光谱(波长400~1 000 nm, 256个波段)进行特征波段筛选, 提取出与番茄品质指标显著相关的特征波长, 特征波长提取见表2。
| 表2 选定的特征波段 Table 2 Selected characteristic bands |
如表2所示, 针对番茄红素、 SSC、 维生素C中三个理化指标, GA 分别提取出22、 18和18个关键特征波段, 与全波段相比, 所选的特征波段的数量分别占全波段的比例分别为8.59%, 7.03%, 7.03%。 与全光谱建模相比, GA显著降低了输入变量维度, 减少了冗余信息和噪声干扰, 有助于降低模型复杂度并提升预测稳定性。 另外, 从特征光谱带分布来看, 在光谱起始段(2~6波段, 对应约400~410 nm), 属于可见光区的短波段, 可能与番茄红素分子在紫外-可见区的电子跃迁相关, 尤其是Soret吸收带, 表明该区域对番茄红素的浓度变化较为敏感。 在波段64~77(对应约520~540 nm)处于番茄红素特征吸收峰的主区域, 进一步验证了该波段与番茄红素含量的密切关系。 波段188~195(对应约780~810 nm)处于“ 红边” 过渡区, 可能与类胡萝卜素的光谱衰减特性和样品组织结构有关。 249~256(对应约960~1 000 nm)处于近红外区域, 可能与水分、 糖类、 酯类及其他分子的C— H、 O— H、 N— H振动吸收有关, 表明番茄红素的分布不仅与色素吸收有关, 还可能受到细胞结构和基质环境的影响。
为了进一步验证特征波长提取的合理性, 图4进一步给出了特征波长的筛选过程以及相应RMSECV的变化趋势, 为遗传算法特征选择提供了清晰的可视化依据。
从图4中可以看出, 随着所选特征波段数量的增加, RMSECV显著下降, 说明初期变量增加对模型性能提升有明显贡献。 当选取的特征波段达到一定数量后(番茄红素22个波段, SSC 18个波段, 维生素C 18个波段), RMSECV 曲线趋于平稳, 继续增加波段数量对模型精度提升有限, 甚至可能引入冗余信息。 虽然不同品质指标所选特征波段存在部分重叠, 表明番茄红素、 SSC与维生素C在光谱响应上存在一定相关性, 但每个指标也具备独立的特征信息。 这种RMSECV的下降趋势充分验证了遗传算法在有效剔除冗余波段、 提取关键特征方面的优越性。 特征波段的优化不仅简化了模型结构, 也为后续建立更具普适性和实际应用价值的预测模型提供了基础。 特征波段选择结果与Zhao等[24]的报道一致, 进一步验证了通过合理选择关键波长能够显著提升高光谱建模的预测性能和泛化能力。
为了分析BP和ELM两种模型在番茄番茄红素、 SSC及维生素C含量预测中的建模性能, 表3给出了不同模型预测不同指标的模型性能评估系数。
| 表3 BP和ELM两种模型性能对比 Table 3 Performance evaluation of various models |
从表3可以看出, ELM模型在番茄红素、 SSC以及维生素C三项品质指标预测中, 整体优于BP-ANN模型。 具体表现在以下几个方面: (1)ELM在校正集
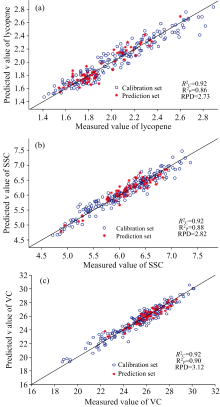
为直观验证极限学习机(ELM)模型在番茄品质预测中的性能, 给出了真实值与预测值的散点图, 其中校正集为蓝色圆点, 预测集为红色点, 如图5所示。
从图5中可以看出三个图中的数据点均高度集中于直线线附近, 表明模型的预测值与实际测量值具有较强的一致性。 从图5(a)番茄红素预测结果来看, 决定系数(
基于高光谱成像技术, 结合标准正态变换(SNV)、 遗传算法(GA)和极限学习机(ELM)模型, 建立了番茄关键品质指标(番茄红素、 SSC和维生素含量)的快速无损检测方法。 实验结果表明: 在400~1 000 nm波段范围内获取的高光谱信息与各品质指标均存在显著相关性, 验证了高光谱技术在番茄品质检测中的可行性与有效性。 通过SNV预处理, 有效消除了散射效应和基线漂移, 显著提升了光谱数据质量; GA成功筛选出与各品质指标显著相关的特征波段, 相比全波段建模, 降低了模型复杂度并提升了预测效率; ELM模型在多指标预测中表现优异, 最高
与已有集中于单一指标预测或单一算法优化的研究不同, 本研究实现了多指标协同预测、 特征波段精准筛选与建模方法优化的有机结合, 形成了一条完整的“ SNV预处理— GA特征筛选— ELM建模” 协同优化流程。 这不仅提升了预测精度与模型稳定性, 为实际检测设备的波段选取和算法部署提供了可行方案, 也为智慧农业和食品品质在线检测的深入研究与推广应用提供了理论依据与技术支撑, 具有良好的应用前景。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|