作者简介: 孙浩然, 1992年生, 吉林交通职业技术学院讲师 e-mail: 1515754043@qq.com
肺癌作为世界范围内发病率和死亡率最高的恶性肿瘤, 其诊断结果直接影响患者的治疗方案及术后复发几率, 传统诊断方式往往依靠医生主观判断, 且过程复杂, 检测周期较长, 因此急需一种能提供客观量化指标且快速检测的肺部肿瘤诊断方法。 本文的目的是首次评估激光诱导击穿光谱技术(LIBS)结合深度学习网络模型作为快速和原位诊断肺部病变组织的潜力。 基于癌变组织中元素成分定量变化, 通过LIBS对45名患者提供的肺部肿瘤及正常组织进行快速检测, 从而获取病变及正常组织间的元素差异信息, 在此基础上通过多变量分析法选取了Ca、 Na、 K等6种元素共12条特征谱线作为模型输入量进行分析, 针对肿瘤及正常组织光谱数据存在复杂预处理和特征提取困难的问题, 设计了1种以ResNet18为主干网络的深度学习光谱特征处理体系, 同时设计了3种机器学习模型, 结合随机森林特征提取方法, 建立了二次递进式的机器学习光谱特征处理体系进行比对。 结果表明, 深度学习网络模型识别能力明显高于其他3种机器学习模型, 其精确度、 灵敏度、 特异度分别达到了99.6%、 100%、 99.3%, 分别有140组肿瘤组织, 139组正常组织光谱数据识别正确, 模型F-Measure、 Recall以及Kappa系数分别达到了99.6%、 99.7%以及99.3%, 说明模型在保证识别精度的同时, 对不同种类光谱数据的识别能力较为均衡, 具有良好的泛化性与鲁棒性。 以上研究表明, 针对肿瘤及正常组织中高维且抽象的光谱特征信息, 与传统化学计量方法相比, 深度学习网络模型中卷积与池化区域能够更高效地挖掘非线性特征, 从而将肿瘤及正常组织内部中相似信息和差异信息快速找出。 通过LIBS与深度学习结合的检测方式能够在肺癌诊断场景中提供病变组织的客观量化信息, 可以作为一种快速、 精确和鲁棒性的肺部肿瘤诊断方法。
Lung cancer is the most deadly form of cancer worldwide, with a high morbidity and mortality rate. The accuracy of a patient's diagnosis directly impacts their treatment plan and the likelihood of recurrence post-surgery. Conventional diagnostic methods are often dependent on the subjective assessment of medical professionals and are time-consuming. Consequently, there is an urgent need for a lung tumor diagnostic method that can provide objective and quantitative metrics, facilitating rapid detection. The objective of this study is to assess the viability of a novel approach that integrates laser-induced breakdown spectroscopy (LIBS) with deep learning network models for the expeditious and in situ diagnosis of diseased lung tissues. The LIBS technique was employed to quantitatively analyze the elemental composition of cancerous tissues, lung tumors, and normal tissues from 45 patients. This analysis enabled the rapid detection of lung tumors and the acquisition of elemental differences between diseased and normal tissues. A total of 12 characteristic spectral lines of 6 elements, including Ca, Na, K, etc., were selected as inputs to the model through multivariate analysis. To address the challenges posed by the intricate preprocessing and feature extraction in tumor and normal tissue spectral data, a deep learning spectral feature processing system with ResNet18 as the primary network was developed. In conjunction with this system, 3 machine learning models were designed to be utilized in conjunction with the Random Forest feature extraction method. To facilitate a comprehensive comparison, a quadratic recursive machine learning spectral feature processing system was also established. The findings indicate that the deep learning network model exhibits a substantially superior recognition capability compared to the other three machine learning models. Its accuracy, sensitivity, and specificity reach 99.6%, 100%, and 99.3%, respectively. The model demonstrated a 99.6% accuracy, 100% precision, and 99.3% recall in the recognition of spectral data from 140 tumor tissues and 139 normal tissues. The model's balanced ability to recognize different spectral data types while ensuring recognition accuracy, in conjunction with its demonstrated generalization and robustness, is noteworthy. The study above demonstrates that, in the context of high-dimensional and abstract spectral feature information in tumors and normal tissues, the convolution and pooling regions in the deep learning network model exhibit a superior capacity to extract nonlinear features in comparison to traditional chemometric methods. This enhanced capability enables the expeditious identification of similarity and difference information in tumors and normal tissues. The integration of LIBS with deep learning has been demonstrated to facilitate the acquisition of objective, quantitative data concerning diseased tissue in the context of lung cancer diagnosis. This approach has been demonstrated to provide a rapid, precise, and robust method for identifying lung tumors.
肺癌作为全球范围内最常见的癌症类型之一, 其发病率和死亡率一直高居不下[1]。 据国家癌症中心报告显示, 2022年中国肺癌确诊人数达到1 060 600例, 死亡人数达到733 300例, 分别占全国癌症确诊人数、 癌症死亡总人数的22%和28.5%, 同时位列癌症发病率及患癌致死率首位, 远高于第二位结直肠癌确诊人数(517 100例)及肝癌死亡人数(316 500例), 且明显呈上升、 年轻化趋势。 吸烟是肺癌最主要的致病因素, 研究表明约有 85%~90%肺癌患者属于吸烟者或有吸烟史, 且随吸烟量增加、 吸烟年龄提前、 烟龄增长等因素, 肺癌概率逐渐增大[2]。 此外职业环境接触、 空气污染、 饮食营养等都可成为肺癌发病诱因[3, 4, 5, 6]。
传统肺癌检查方法中, 病理学检查被视为诊断的“ 金标准” , 包括常规病理组织切片、 免疫组织化学和图像分析等技术[7, 8, 9, 10]。 然而这些检查流程繁琐, 耗时较长, 尤其在手术中的快速组织切片病理分析场景中, 医生需要在短时间内完成诊断。 此外, 目前常规检查缺乏客观量化指标, 诊断结果主要依赖于医生的宏观评估, 而患者的治疗方案和长期生存率在很大程度上取决于诊断时的疾病程度, 这导致了较高的误切和术后复发风险。 同时肿瘤组织自身特征, 如病变不典型[11]、 交界性等因素[12], 也会对诊断结果产生一定影响。 因此开发一种敏感且可靠的肺部肿瘤诊断方法显得尤为重要。
研究表明生命体病变过程会导致生物体内部元素分布发生变化, 例如钙离子作为细胞内的“ 第二信使” , 可同时调节同一种细胞的不同功能, 表征生物体内各元素对癌症发展过程至关重要, 因此可将肿瘤组织中各元素含量差异作为判断依据来对病变组织进行进一步探索。 激光诱导击穿光谱技术(laser-induced breakdown spectroscopy, LIBS)是一种基于元素探测的光谱技术, 具有检测周期短、 多元素检测、 破坏性小、 无需预处理等优点, 目前已被广泛用于冶金、 矿产、 航天等领域中不同基质物质的定性或定量测量研究[13, 14, 15]。 在疾病诊断方面, 因其对生物组织中各元素敏感性较高, Kiss等[16]通过LIBS对皮肤恶性黑色素瘤、 鳞状细胞癌、 肿瘤基底细胞癌, 血管瘤进行分析, 以元素图像的方式展示了健康和肿瘤组织之间的差异性。 Teng等[17]通过LIBS结合支持向量机和特征选择对脑胶质瘤进行识别, 识别率达到95%。 Chen等[18]使用LIBS结合化学计量法对淋巴瘤和多发性骨髓瘤血清进行检测, 整体判别率达到了96.0%。 Chu等[19]通过LIBS与极限学习机对鼻咽癌血清进行鉴别, 准确率达到了98.3%。 以上研究表征了LIBS技术对病变组织检测的可行性, 然而以上研究工作大多采用机器学习模型来处理光谱数据, 其模型性能主要依赖于特征选择方法, 但不同特征选择方法都存在自身局限性, 同时生物组织内部元素种类及含量复杂, 且光谱数据量庞大, 其数据高维性及特异性会导致光谱数据处理过程中存在数据挖掘困难, 特征提取能力差等问题, 同时复杂的光谱预处理和特征选择会延长分析时间, 并导致光谱失真及潜在的精度降低。
近年来基于深度学习的卷积神经网络凭借其强大的数据特征提取能力在计算机检测与回归任务中得到广泛应用, 与传统方法相比, 卷积神经网络能够通过卷积、 池化等操作将数据各通道信息映射到深度网络结构中, 从而提高网络信息挖掘能力[20]。 因此提出采用LIBS结合深度学习的方式, 对肺部肿瘤、 正常组织光谱数据进行诊断。 首先通过LIBS采集肺部肿瘤及正常组织LIBS特征光谱, 从而获取病变及正常组织间的元素差异信息, 在此基础上建立深度学习模型(ResNet18)和3种机器学习(SVM、 KNN、 Bagging Tree)模型, 并通过交叉验证的方式进行建模识别, 最终通过判定模型准确度、 灵敏度、 ROC曲线等指标对模型识别能力进行全面评估, 从而达到对肺部病变组织快速诊断的目的。
肺部肿瘤组织切片及正常组织切片如图1所示。 选取长春市中日联谊医院病理科登记的肺部肿瘤患者作为研究对象, 共计45名, 其中男性20名, 女性25名, 年龄介于39至80岁之间。 样本采集在离体30 min后完成采集。 为确保样本清洁和减少外界污染, 使用生理盐水进行彻底冲洗, 以去除血液、 粘液等杂质。 随后将样本置于专用内旋式冻存管中, 并立即转移至-80 ℃的液氮柜进行长期保存。 整个样本处理过程均在严格控制的洁净环境中进行。
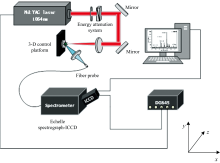
LIBS实验装置结构如图2所示。 激光光源为输出波长1 064 nm的Nd:YAG激光器(SLII-10, Continuum), 激光能量65 mJ, 脉宽10 ns, 重复频率10 Hz, 光束直径约为6 mm, 光束模式为TEM00。 等离子体通过光纤耦合至配备有ICCD的中阶梯光栅光谱仪(Me5000, Andor)进行探测。 实验环境为标准大气压, 温度22 ℃, 相对湿度27%。 样品被放置在载玻片上并通过竖直放置的三维平移台(M-410.DG, PI)进行调整, 以避免存在烧蚀重复点。 平均15束脉冲收集1幅光谱, 肿瘤组织及正常组织分别采集140幅LIBS特征光谱。
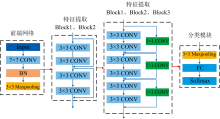
首先通过小波阈值降噪对光谱数据内部信息的重构与分解, 以保证在有效降噪的同时尽量保留谱线原始信息。 选取多变量分析的方法, 结合US NIST atomic spectroscopy database及LIBS特征光谱对元素谱线进行选取。 在深度学习模型构建方面, 选择ResNte18网络模型作为模型主干网络进行建模, ResNet18是一种基于残差思想的网络模型, 通过在网络模型中引入残差边, 保留原始网络层输出, 从而避免网络层学习冗余信息, 并减少网络计算量, 其网络结构如图3所示。
网络模型分为前端网络、 特征提取、 分类模块3个部分。 前端网络主要包括1个输入层, 1个7× 7的卷积层, 1个池化层。 在特征提取部分首先由2个不带有残差边的Block进行特征提取, 共4个卷积层, 每个卷积层使用3x3的卷积核和ReLU激活函数来提取光谱数据局部特征。 之后通过3个带有1× 1残差边的Block进行特征提取, 每个残差块由两个卷积层和一条跳跃连接构成, 通过全连接层将学到的特征表示映射到样本标记空间, 最后使用Softmax激活函数生成类别的概率分布。 实验以Pytorch框架为基础, GPU采用NVIDIA-3060Ti, 学习率设置为0.000 1, 训练周期为35轮次。
除构建深度模型外, 还构建了3种机器学习模型对肿瘤及正常LIBS光谱数据进行识别, 分别从平面分割(SVM)、 距离结构(KNN)、 集成思想(Bagging Tree)3个数学角度进行建模。 此外生物组织光谱数据集存在大量冗余、 不相关和噪声特征及复杂相互作用等问题, 由于输入变量在建模中起着至关重要的作用, 无用信息变量会对分析结果产生有害影响, 然而机器学习算法并不具有卷积神经网络强大的特征提取与网络算力, 因此本文首先通过随机森林特征向量选择对光谱数据特征进行选取, 以此来提高光谱信息挖掘效率同时降低无效变量干扰。
在模型评估方面, 为消除样本之间可能存在的相关性, 选择10倍交叉验证实现平衡偏置和方差。 将准确度、 灵敏度、 特异度作为评估指标。
准确度(accuracy)表示为
灵敏度(Sensitivity)表示为
特异度(Specificity)表示为
式(1)— 式(3)中, TP、 TN、 FN、 FP分别表示真阳性、 真阴性、 假阴性、 假阳性。 通过Kappa、 Precision、 Recall、 F-Measure、 MCC(matthews correlation coefficient)来对模型整体识别能力进行评估。 Kappa作为衡量数据与模型一致性指标, 表示为
式(4)中, Po为模型整体分类精度, Pe为实际与预测数量的乘积之和除以样本总数的平方。 Precision、 Recall反映了分类模型的精确度, F-Measure用来衡量上述两种指标, MCC描述了实际分类与预测分类之间的相关系数, 它的取值范围为[-1, 1]。 Precision、 Recall、 F-Measure、 MCC计算公式分别为
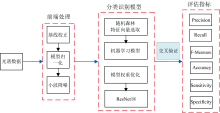
具体流程如图4所示。
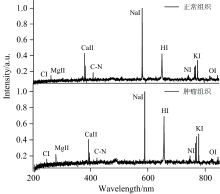
图5显示了同一患者肺部正常与肿瘤组织LIBS特征光谱图, 光谱范围在200~900 nm之间。 首先观察到的元素包括金属元素(Mg、 Na、 K、 Ca)和非金属元素(C、 H、 O、 N)以及分子键C— N带。 其中金属元素如Ca、 Mg在肿瘤组织中的强度高于正常组织, 作为细胞酶中的重要成分, 金属微量元素含量改变可使细胞功能及状态发生变化, 导致细胞代谢、 繁殖异常, 研究表明, 由于病变引起的细胞元素积累, 会导致不同阶段和类型的肺癌组织中金属微量元素含量有着很大的差别。 通过对比两种组织特征光谱结果表明元素差异与肿瘤病变信息密切相关, 然而, 仅通过比较光谱特征峰很难对病变组织进行精确诊断, 因此需要进行深入研究探索更多信息。
由于光谱数据中包含大量的无用数据, 同时LIBS检测技术需尽量选择高灵敏度及低LOD元素来进行检测, 从而保证在有限的检测限下获得合理的结果。 在综合考虑变量之间的相关性以及谱线的物理意义的基础上, 通过观察肿瘤及正常LIBS谱线图, 结合NIST数据库, 首先选择了4种金属元素和2种非金属元素作为深度学习及机器学习模型特征向量进行研究, 如表1所示。
| 表1 特征谱线选取 Table 1 Library of selected emission lines |
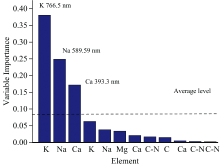
由于机器学习算法并不具有卷积神经网络强大的特征提取与网络算力, 为避免无用信息变量会对分析结果产生影响, 需要进一步筛选机器学习模型的输入特征, 本文采用随机森林算法(Random forest, RF)对光谱各谱线权重进行计算, 结果如图6所示, 谱线权重平均值为0.083, 其中K 766.5 nm、 Ca 393.3 nm和Na 588.59 nm谱线权重大于平均值, 为0.38、 0.25和0.17。 因此将以上3条特征谱线作为机器学习模型的输入变量, 以减少谱线中冗余特征所带来的干扰信息。
本文首先通过Precision、 F-Measure等参数对模型整体识别能力进行讨论。 首先构建了3种机器学习模型, 并将通过随机森林特征选择后的特征作为模型输入量, 表2显示了3种模型对肺部组织光谱数据的Precision、 F-Measure等参数, 体现了3种机器学习模型对不同种类以及总体光谱数据的识别能力。 与其他两种机器学习模型相比, SVM模型对光谱识别能力较差, 仅有91.4%, 由于SVM模型是一种基于结构风险最小化和有限样本学习的统计学习算法, 主要通过超平面对数据进行分割, 然而针对生物组织光谱数据的高维特性以及内部复杂特性, 使得光谱数据非线性程度较高, SVM算法并不能进行有效的抑制, 导致模型精度较低。
| 表2 RF-KNN、 RF-SVM、 RF-Bagging Tree模型Precision、 F-Measure、 Recall、 MCC Table 2 The Precision, F-Measure, Recall, MCC of RF-KNN, RF-SVM, RF-Bagging Tree model |
与SVM模型相比, KNN模型与Bagging Tree模型识别精度虽然有所提高, 但模型识别能力不均衡, 例如Bagging Tree模型对肿瘤组织光谱数据识别准确率为92.5%, 但对正常组织光谱数据识别准确率为96.3%, 两者差异较大, 说明以机器学习算法作为肺部肿瘤病变组织的诊断手段并不具有较强的泛化性, 由于本文主要针对病变组织精确诊断场景, 模型识别精度直接影响到患者的治疗方案以及术后病变组织的复发概率, 因此, 模型精度仍有待提高, 以确保其在临床诊断中的可靠性。
为进一步提高模型识别精度, 从而达到病变组织精确诊断的精度要求, 本文构建了以ResNet18为主干网络的深度学习模型。 模型识别结果如表3所示。 ResNet18网络模型识别精确率达到了99.7%, F-Measure与召回率分别达到了99.6%以及99.7%, 结果明显高于其他三种机器学习模型算法。 表4显示了4种模型的识别精确度、 灵敏度、 特异度以及Kappa系数, 体现了模型识别的泛化性以及一致性。 可以看出, CNN模型在总体精确度高于其他3种机器学习模型的基础上, 其对两种组织光谱数据的识别能力较为均衡, 灵敏度以及特异性分别达到了100%和99.3%, 证明了CNN模型的数据挖掘和特征学习能力能够满足肺部肿瘤光谱数据精确识别的要求, 同时深度学习模型不需要预先进行特征选择, 可有效简化实验过程, 避免人为因素导致光谱失真的风险。 此外CNN模型Kappa为99.3%, 说明深度学习模型在检测精度以及模型一致性方面优于机器学习模型算法。
| 表3 ResNet18 模型Precision、 F-Measure、 Recall、 MCC Table 3 The Precision, F-Measure, Recall, MCC of ResNet18 model |
| 表4 四种模型精确度、 灵敏度、 特异度、 Kappa系数 Table 4 The accuracy, sensitivity, specificity, Kappa of 4 models |
由于本文使用大量光谱数据导致表2、 表3、 表4的模型识别精度在数值水平上差异并不直观, 因此本文进一步讨论了4种模型的混淆矩阵, 以便更加直观展示CNN模型的最佳性能。 如图7所示。 在所有模型中, SVM模型识别结果最差, 280组数据中, 共有24组光谱数据被错误识别, 而KNN和Bagging Tree模型分别共有18组和16组光谱数据识别错误, 同时可以发现机器学习模型对肿瘤光谱数据识别较为敏感, 以KNN模型为例, 肿瘤光谱数据只有3组识别错误, 灵敏度达到了97.9%, 这一现象说明机器学习算法在提取特征较为明显的数据(如肺部肿瘤识别场景)更具有优势。 相比之下, ResNet18网络模型在对肺部肿瘤以及正常组织光谱数据预测时精确度明显提高, 其中肿瘤组织光谱数据全部识别正确, 仅将1组正常组织光谱数据误判为肿瘤组织光谱数据, 这表明CNN模型在识别样本和训练学习样本方面都有很大的性能提升。 以上研究表征了传统模式识别仍然需要复杂的预处理和特征选择工程来提高其分类性能, 而CNN模型中卷积层和池化层可作为特征提取器, 全连接层作为优化改进分类器, 可通过密集挖掘多个局部特征以获得更好的分类效果。
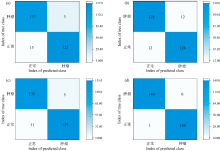 | 图7 模型混淆矩阵 |
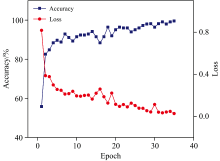
为展示CNN模型训练效果, 本文将ResNet18模型训练学习过程可视化。 在35次迭代过程中, ResNet18模型每一步学习精度与损失率如图8所示。 在19次计算时, 模型快速收敛, 学习精度可达到92%, 在20次计算之后模型最高精度可超过99%, 表明该模型具有优秀的学习能力与稳定性。 另一方面, 经过20次迭代后损失函数值逐渐趋近于0, 这意味着模型预测值逐渐接近真实值。
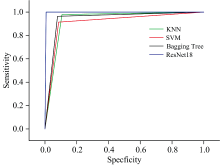
图9显示了SVM、 KNN、 Bagging Tree、 ResNet18模型对于肺部肿瘤组织-正常组织光谱数据的ROC曲线, 与SVM、 KNN以及Bagging Tree模型曲线相比, ResNet18模型曲线拐点更靠近左上方, 说明ResNet18模型在对各种光谱数据识别的能力要高于其他模型, 在保证模型精确度的同时, 整体识别能力较为均衡。
本文的目的是首次评估LIBS结合深度学习网络模型作为快速和原位诊断肺部病变组织的潜力。 将两种技术连用可分别从原子或离子结构层次以及光谱数据中高维抽象特征信息层次进行数据挖掘。 数据分析表明, LIBS技术对于金属元素含量存在差异的病变以及正常组织可以表现出很好的光谱特征差异, 而卷积神经网络中卷积区域能够挖掘更高效的非线性特征, 从而将组织内部的相似信息和差异信息快速找出。 结果表明, LIBS结合深度学习技术获得的信息可以在一定程度上克服其他技术在肺部病变组织诊断的潜在局限性。
本文介绍了一种利用深度学习网络模型和LIBS的肺部肿瘤组织快速诊断方法。 首先通过比较两种组织的LIBS光谱差异, 在此基础上建立了以ResNet18为主干网络的深度学习模型以及3种机器学习模型, 并通过随机森林算法对光谱特征向量权重进行评估, 选择了特征权重大于平均值的特征向量作为机器学习模型输入量。 结果表明, 深度学习网络模型在模型精确度、 泛化性上均比传统机器学习模型体现出更好的能力, 模型精度可以达到99.6%。 在LIBS-ResNet18检测模型中, 原子或离子组成可以从LIBS光谱中确定, 而光谱高维的抽象特征信息可以通过深度学习技术进行挖掘。 本研究表明, 为了肺部肿瘤组织进行完整的分析和识别, 通过LIBS与深度学习结合的方法可以为识别人体病变组织特征提供有价值的信息, 是提高病变组织诊断精度的有效途径。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|