作者简介: 谢 星, 1993年生, 江西师范大学健康学院食品营养与健康系讲师 e-mail: 13609092537@163.com
采用光谱分析结合分子模拟技术探究了鞣花酸(EA)及其代谢产物尿石素A~D(UA~D)与人血清白蛋白(HSA)的相互作用机理, 有助于解析其药理毒性和药效。研究结果表明EA和UA~D能与HSA以1∶1的摩尔比例结合并通过静态方式猝灭HSA的荧光。UA和UC与HSA结合是氢键和范德华力驱动的放热过程, 而EA和UD与HSA结合疏水相互作用驱动的吸热过程。三维荧光图谱分析表明UC~D和EA与UA~B分别增加了HSA色氨酸和酪氨酸微环境的亲水性和疏水性。分子模拟分析结果表明, EA和UA~D与HSA的活性氨基酸残基Lys436、 Asp187、 Lys432、 Arg485、 Leu430、 Leu4、 Ile388、 Tyr411等形成氢键, 与氨基酸残基Ala191、 Val456、 Lys199和Trp214之间存在疏水相互作用, 证明其主要通过氢键和范德华力与HSA结合, 屏蔽HSA的糖基化位点, 抑制其糖基化。可为EA和UA~D作为糖基化抑制剂用于治疗糖尿病并发症奠定理论基础。
Spectroscopy and molecular simulation technologies investigated The interaction mechanism between ellagic acid (EA) and urolithin A~D (UA~D) with HSA, which helped explore its pharmacotoxicity and efficacy. The results indicated that EA and UA~D could bind with HSA at a molar ratio of 1∶1 and quench its fluorescence via a static mechanism. The binding of UA and UC with HSA was exothermic and driven by hydrogen bonding and van der Waals force. The binding of EA and UD with HSA was endothermic and driven by hydrophobic interaction. The three-dimensional fluorescence spectrum analysis exhibited that the addition of EA and UC, UD, UA, and UB enhanced the hydrophilicity and hydrophobicity of tyrosine and tryptophan microenvironments of HSA, respectively. The molecular simulation analysis showed that EA and UA~D formed hydrogen bonds with active amino acid residues Lys436, Asp187, Lys432, Arg485, Leu430, Leu4, Ile388 and Tyr411, and formed hydrophobic interaction with active amino acid residues Ala191, Val456, Lys199 and Trp214, which proved that they were majorly bound to HSA by hydrogen bonds and van der Waalsforce, and then screened glycation sites and inhibited HSA glycation. This study could provide a theoretical basis for developing EA and UA~D as non-enzyme glycation inhibitors to treat diabetic complications.
人血清白蛋白(human serum albumin, HSA)是人体血浆中含量最丰富的蛋白质, 约占血浆总蛋白的60%, 包含585个氨基酸, 分子量为66kD, 可起到运输药物、 毒素、 营养素、 激素和代谢物等疏水小分子的作用[1]。糖尿病患者长期处于高血糖状态, 血浆中的葡萄糖与HSA发生非酶糖基化反应, 导致HSA结构和功能的改变, 生成大量的晚期糖基化产物(advanced glycation end products, AGEs)[2]。AGEs与蛋白质发生交联, 促进活性氧自由基产生, 增加动脉粥样硬化、 老年痴呆症、 肾病、 白内障等糖尿病并发症的发病率[3]。抑制HSA糖基化可有效减少AGEs的产生和降低糖尿病并发症的发病率。
氨基胍(aminoguanidine, AG)是常用的糖基化抑制剂, 长期摄入会导致胃肠道紊乱、 血管炎、 贫血和流感样等症状, 并具有毒性和促氧化作用, 临床上已被禁止使用[4]。天然来源的糖基化抑制剂具有低毒、 高效和副作用小的特点, 是当前的研究热点。研究发现单宁和黄酮可以与HSA自发发生非共价相互作用, 起到保护蛋白质结构和抑制非酶糖基化的作用[5]。鞣花酸(ellagic, EA)是由鞣花单宁水解产生的单宁, 广泛存在于石榴、 覆盆子、 核桃和草莓等植物、 水果和坚果中, 具有抗氧化、 抗菌、 抗炎、 抗肿瘤、 降血糖等多种生物活性[6]。在胃肠道消化过程中, 只有小部分EA被吸收, 大部分进入结肠酵解代谢成尿石素类化合物, 易被结肠粘膜吸收进入血液中, 可发挥多种生物活性[7]。研究发现EA和尿石素类化合物可降低HSA糖基化体系中AGEs的形成, 但其与HSA的相互作用机理尚不明确, 亟需进一步的探究。
紫外和荧光光谱是研究小分子与蛋白质相互作用的常用手段, 小分子化合物的加入会增强蛋白质的紫外吸光值和猝灭其荧光, 通过内源荧光的变化可分析小分子化合物与蛋白质结合过程中的的结合常数和热力学参数等, 外源荧光可研究小分子化合物对蛋白质空间构象和疏水微环境的影响, 这对解析小分子化合物的药理作用具有重要的意义[8]。通过光谱分析结合分子模拟技术探究EA和尿石素A~D(urolithin A~D, UA~D)与HSA的相互作用机理, 即采用紫外光谱分析两者之间是否发生了结合, 采用内源荧光、 同步荧光和三维荧光探究EA和UA~D的荧光猝灭机理、 结合参数和相互作用力等, 及其对HSA空间构象和微环境的影响, 采用分子模拟技术分析EA和UA~D与HSA相互作用的分子机制。为EA和UA~D作为糖基化抑制剂进行开发提供科学依据。
EA、 UA~D、 NaH2PO4和Na2HPO4(上海源叶生物科技有限公司); HSA(美国sigma公司)。
U-2910紫外可分光光度计和F-7000荧光分光光度计(日本日立有限公司); PE28 pH计[梅特勒-托力多仪器(上海)有限公司]。
1.2.1 紫外光谱分析
取3 mL 2 mg· mL-1 HSA溶液(0.1 mol· L-1 pH 7.4的PBS缓冲溶液配制)于比色皿中, 添加20 μ L 1 mmol· L-1的EA和UA~D溶液, 混匀后于25 ℃水浴5 min, 用紫外分光光度计扫描样品。具体参数: 波长190~450 nm, 扫描速度200 nm· min-1, 步幅5 nm, 狭缝宽度为1.5 nm。
1.2.2 内源荧光光谱分析
采用F-7000荧光分光光度计研究EA和UA~D对HSA内源荧光的影响。取3 mL 2 mg· mL-1 HSA溶液于1 mL比色皿中, 向其中添加20 μ L 1 mmol· L-1的EA和UA~D溶液, 混匀后25 ℃水浴5 min, 在280 nm激发波长下, 记录发射光谱300~400 nm的荧光光谱, 激发与发射波长的狭缝宽度均设为2.5 nm, 电压为700 V。
1.2.3 荧光滴定实验
取3 mL 1 mg· mL-1的HSA溶液于荧光比色皿中, 分别加入10 μ L 1 mmol· L-1的EA、 UA~D溶液, 重复滴加6~8次, 混匀后分别于298、 304和310 K下水浴5 min, 测定其荧光光谱和最大荧光强度值。具体参数为: 激发波长280 nm, 发射波长300~450 nm, 电压700 V, 激发与发射波长的狭缝宽度均设为2.5 nm。
采用Stern-Volmer方程计算EA和UA~D的荧光猝灭常数(Ksv)和猝灭速率常数(Kq), 公式如式(1)[9]
式(1)中, F、 F0分别表示体系添加和不添加EA和UA~D时HSA的荧光强度; [Q]为EA和UA~D的浓度; τ 0(10-8 s)为不加猝灭剂时HSA的平均荧光寿命。
EA和UA~D与HSA的结合常数(Ka)和结合位点数(n)可通过双对数方程Double-Logarithmic计算[10], 见式(2)
通过van’ t Hoff方程计算EA和UA~D热力学参数, 包括焓变(Δ H° )、 熵变(Δ S° )和酶自由能(Δ G° )变化, 见式(3)和式(4)
式中, R为气体常数; T为反应体系的温度。
1.2.4 同步荧光测定
采用F-7000荧光分光光度计分析EA和UA~D对HSA色氨酸和酪氨酸微环境的影响。分别往3 mL 2 mg· mL-1的HSA溶液中逐滴加入1 mmol· L-1的EA和UA~D, 混匀后25 ℃水浴反应5 min, 测定样品的荧光光谱和最大荧光吸收值。激发起始波长分别设为280与310 nm, 发射波长分别设为265~400和250~400 nm, 激发和发射波长的狭缝宽度均设为5.0 nm。
1.2.5 三维荧光测定
分别取3 mL 2 mg· mL-1 HSA溶液于比色皿中, 添加20 μ L 1 mmol· L-1的EA和UA~D溶液, 混匀后25 ℃水浴5 min, 测定其在激发和发射波长扫描范围均为200~600 nm时的荧光光谱, 激发和发射波长的狭缝宽度均为5.0 nm, 扫描速率为12 000 nm· min-1。
1.2.6 分子对接
采用AutoDocktools 1.5.6软件研究HSA与EA和UA~D相互作用的分子机制。HSA的三维结构图(1AO6)从Protein Data Bank (https://www.rcsb.org/)下载, EA、 UA~D的3D结构图由ChemBio3D Ultra 14.0软件绘制, 并以最低能量保存为pdb文件, 并用AutoDockTools 1.5.6软件优化处理结构图, 保存为PDBPT文件后, 进行分子对接模拟分析。网格盒设置为60× 60× 60, 网格间距为0.375 Å , 采用拉马克遗传算法(Lamarckian genetic algorithm, LGA)进行分子对接计算, 其他参数均为默认值, 对接次数为100, 选择结合能量最低的蛋白质-配体复合物为目标, 采用Discovery Studio Visualizer软件对其进行可视化处理。
1.2.7 数据分析
所有实验重复3次, 结果以平均值(标准偏差表示, 采用SPSS Statistics 22.0软件对数据进行统计分析, 采用Origin 2019绘制图谱, 并以Duncan进行显著性差异分析, p< 0.05认为具有显著性差异。
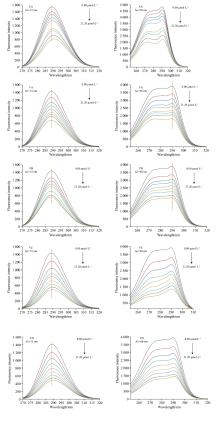
HSA蛋白分子中含有带苯环的芳香族氨基酸如酪氨酸和色氨酸, 因此其在紫外区(190~450 nm)有吸收, 可通过紫外吸收值的高低判断其是否与小分子化合物发生了结合[11]。由图1(a), 天然HSA的紫外吸收值最低, 加入EA和UA~D后, 其紫外吸收值由1.072分别增加至1.145、 1.164、 1.218、 1.369、 1.275。以上结果表明EA和UA~D与HSA发生结合, 并形成了复合物, 而添加UC后HSA的紫外吸收值增加最多, 说明UC与HSA的结合能力最强。
 | 图1 EA和UA~D对HSA紫外光谱(a)和荧光图谱(b)的影响Fig.1 Effect of EA and UA~D on the UV (a) and fluorescence spectrum (b) of HSA |
EA、 UA~D对HSA内源荧光强度的影响如图1(b)所示, 天然HSA具有最大荧光吸收, 添加EA和UA~D后, 其峰值分别从3 870降至2 811、 3 079、 3 422、 1 808和2 019, 表明EA和UA~D能猝灭HSA的内源荧光, 其与HSA发生了结合并形成了复合物[12], UC的猝灭效果最强, 其次为UD。UA使HSA的最强荧光吸收峰发生了蓝移(左移1 nm), UC和UD使最强荧光吸收峰发生了红移(分别右移0.4 和0.8 nm), 说明UC、 UD与UA的加入分别导致HSA的微环境向亲水和疏水方向移动[13]。
2.2.1 荧光动力学分析
当激发波长为280 nm时, 小分子化合物通过激发态反应、 分子重排、 能量转移、 形成基态络合物等方式猝灭蛋白质的荧光, 采用荧光滴定实验可分析EA和UA~D与HSA相互作用关系[14]。如图2所示, 在不同的温度下, Stern-Volmer曲线呈现良好的线性关系, 说明EA和UA~D均通过单一的方式猝灭HSA的荧光。如表1所示, EA、 UA、 UC和UD的KSV值随反应温度的升高而降低, 在304 K时, 其Kq的值分别为9.59× 1012、 7.69× 1012、 9.84× 1012和20.27× 1012 L· mol-1· s-1, 远大于最大猝灭速率常数, 表明EA、 UA、 UC和UD是通过与HSA结合形成基态络合物而静态猝灭HSA的荧光[12]。
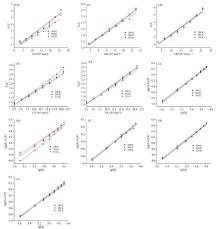 | 图2 在298、 304和310 K下, EA和UA~D猝灭HSA荧光的Stern-Volmer曲线和双对数曲线Fig.2 The Stern-Volmer and double logarithmic plots forthe fluorescence quenching of HSA by EA and UA~D at 298, 304 and 310 K |
| 表1 不同温度下HSA与EA和UA~D相互作用的荧光猝灭常数、 结合常数、 和热力学参数 Table 1 The quenching constants, binding constants, number of binding sites and thermodynamic properties for the interaction of HSA with EA and UA~D at different temperatures |
2.2.2 结合常数和热力学常数分析
如表1所示, 在298、 304和310 K下, EA和UA~D的结合位点数为1.052~1.375之间, 接近于1, 表明其在HSA上有一个或者一类结合位点, 两者之间结合的摩尔比例为1∶ 1。随着温度的升高, UA~C的Ka值出现降低的趋势, 表明UA~C-HSA复合物的稳定性随温度的升高而降低, UB的Ka值最大, 其与HSA结合力最强[3]。
由表1, UA和UC与HSA相互作用的Δ S° 、 Δ H° 和Δ G° 值均小于零, 说明UA和UC与HSA的结合是一个自发进行的放热过程, 氢键和范德华力是其主要驱动力[15]。UB与HSA相互作用的Δ H° 和Δ G° 值小于零, 而Δ S° 值大于零, 表明两者的结合是自发进行的放热过程, 其主要驱动力为静电相互作用力。EA和UD与HSA相互作用的Δ S° 和Δ H° 值均大于零, 而Δ G° 值小于零, 说明EA和UD与HSA结合是一个自发进行的吸热过程, 疏水相互作用是形成EA/UD-HSA复合物的主要相互作用力[16]。
同步荧光光谱可以反映荧光基团微环境的变化, Δ λ =15和60 nm分别表示EA和UA~D对HSA酪氨酸和色氨酸微环境的影响[17]。如图3所示, 随着EA和UA~D浓度的增加, HSA酪氨酸和色氨酸残基的最大荧光吸收值呈现降低的趋势, 其对色氨酸的猝灭效果显著强于酪氨酸, 表明EA和UA~D主要与HSA的色氨酸残基发生了结合。EA、 UC和UD使HSA酪氨酸和色氨酸残基的最大荧光吸收峰发生了红移(0.8~1.4 nm), UA和UB使酪氨酸的最大荧光吸收峰发生了轻微的蓝移(0.2 nm), UB使色氨酸的最大荧光吸收峰发生了轻微的红移(0.2 nm)。以上结果说明, EA、 UC和UD会增加HSA色氨酸和酪氨酸微环境的亲水性, UB会增加HSA色氨酸和酪氨酸微环境的疏水性, UA则会增加HSA酪氨酸微环境的疏水性, 其中EA对HSA微环境的改变效果最显著, 其次是UC和UD, UA的影响最弱[9]。
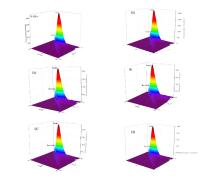
EA和UA~D对HSA三维荧光的影响如图4所示, Rayleigh峰(Ex=Em)表示瑞利散射峰, 峰1(Ex=280 nm, Em=330 nm)为酪氨酸和色氨酸残基的光谱特征峰, 其能反映HSA多肽骨架的结构的改变[18]。添加EA和UA~D之后, HSA的Rayleigh峰的荧光强度显著增加, 峰1的荧光强度显著减低, 且UC对峰1的影响最大, UB和UC次之。以上结果表明EA和UA~D与HSA的结合引起了其构象的变化。峰1荧光强度值的降低可能是因为EA和UA~D与HSA的结合位点靠近酪氨酸和色氨酸残基[19]。
采用分子对接技术进一步探究了EA和UA~D与HSA相互作用的分子机制。HSA有两个主要结合位点, Ⅱ A子结合域为位点1, Ⅲ A子结合域为位点2, 本研究主要模拟分析了EA和UA~D与HSA位点1和位点2的结合作用, 发现EA和UA~D与HSA位点1和位点2的结合能分别是-5.74、 -5.98, -6.11、 -6.78和-6.99、 -6.92, -6.12、 -6.37, -5.55、 -6.5。EA、 UA、 UC和UD与HSA结合位点2有最低结合能, UB则与HSA结合位点1有最低结合能, 采用Discovery Studio软件对最低结合能的对接模型进行可视化分析, 其结果见图5(a, b)。
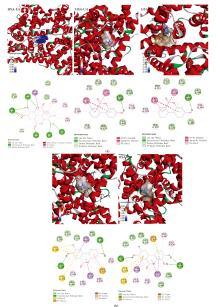 | 图5 EA (a)和UA~D (b)与HSA相互作用的二维和三维结构图Fig.5 Tertiary (a) and two-dimensional structure (b) of the interaction between EA and UA~D with HSA |
EA与HSA的氨基酸残基Lys436、 Asp187、 Lys432、Asn429、 Gln459结合形成7个氢键, UA与氨基酸残基Arg410、 Arg485和Leu430形成3个氢键, UB与氨基酸残基Leu481形成1个氢键, UC与氨基酸残基Ile388、 Tyr411形成2个氢键, UD与氨基酸残基Ile388、 Tyr411形成2个氢键。EA和UA~D分别与4、 6、 6、 11和9个HSA氨基酸残基发生疏水相互作用, 涉及的氨基酸残基包括Ala191、 Val456、 Lys199和Trp214等。结果表明氢键和疏水相互作用是EA和UA~D与HSA结合的主要作用力, 与热力学研究结果一致。EA和HSA能形成更多的氢键, 表明EA-HSA复合物的结构最稳定[20]。计算机模拟发现参与EA、 UC和UD非共价结合的氨基酸残基显著多于UA和UB, 与前期研究发现的EA、 UC、 UD对HSA微环境影响程度强于UA与UB一致。EA和UA~D可通过与HSA的活性氨基酸结合而起到屏蔽HSA糖基化活性位点的作用。
EA和UA~D与HSA的相互作用机理: 自发的情况下, EA和UA~D可通过氢键、 范德华力、 静电相互作用力和疏水相互作用等驱动力与HSA的活性氨基酸进行结合, 并以静态的方式猝灭HSA的荧光, 通过改变HSA酪氨酸和色氨酸的微环境而影响其空间构象, 进而屏蔽HSA的糖基化位点, 达到抑制糖基化的作用。EA含有的羟基数目最多, 与HSA形成的复合物更稳定, 表明羟基对其抑制糖基化能力具有重要的贡献。UA~D具有相似的结构, 但UC和UD与HSA的结合能力显著强于UA和UB。相比于UA~B, UC和UD的B环上多了2个羟基, 形成了更多的氢键, 说明B环上的羟基能促进其与HSA的结合。Zhang等和Ma等的研究也有相似的结果[21, 22]。
通过紫外和荧光光谱结合分子模拟研究了EA和UA~D与HSA的相互作用机理。紫外和荧光光谱分析表明EA和UA~D与HSA结合形成稳定的复合物, UC的结合能力最强。荧光动力学分析发现EA和UA~D会静态猝灭HSA的荧光, 结合位点的数目为1。EA和UA~D与HSA结合是一个自发的过程, 形成UA/UB/UC-HSA复合物、 EA-HSA复合物和UD-HSA复合物的驱动力分别是氢键和范德华力、 静电相互作用力和疏水相互作用。同步荧光和三维荧光分析均表明EA和UA~D与HSA的相互作用改变其构象, 改变酪氨酸和色氨酸微环境的亲水性和疏水性, 其中EA对微环境的影响最大。分子模拟结果表明EA和UA~D与HSA结合分别形成了7、 3、 1、 2、 3个氢键, 其中EA形成的氢键最多, EA-HSA复合物的结构最稳定。EA和UA~D可通过与HSA的活性氨基酸结合而改变其结构, 屏蔽HSA的活性位点, 起到抑制HSA糖基化的效果。本研究可为理解EA和UA~D作为抗糖基化的药代动力学和药效作用提供借鉴, 为其在糖尿病预防和治疗领域的开发利用奠定理论基础。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|