作者简介: 李 璇, 1999年生,昆明理工大学国土资源工程学院硕士研究生 e-mail: 15912550925@163.com
利用高光谱数据对湿地植被进行识别历来是植被遥感研究的重点之一。 高光谱遥感数据包含更加细致的植被光谱特征, 为高光谱植被识别提供了强有力的手段。 以洱海东岸海滨为研究区, 测取了3种典型湿地植被(菰、 芦、 槐叶蘋)的高光谱数据作为目标样本。 对原始光谱进行一阶微分、 包络线去除变换并分析其光谱特征, 采用连续投影(SPA)、 竞争性自适应重加权采样(CARS)两种特征变量选择算法选取原始光谱及其变换光谱中的特征波长, 最后基于全波段数据以及特征波长选取后的数据分别建立支持向量机(SVM)、 随机森林(RF)、 径向基(RBF)神经网络的识别模型。 结果表明: SPA与CARS算法对高光谱数据都有良好的降维效果, 选取出的特征波长数量在5~18之间。 对比组合不同的光谱变换处理与特征波长提取方法进行模建实验, 包络线去除-SPA-SVM模型识别三类目标样本表现最好, 其识别精度为0.937 5, 此时选取用于输入建模的特征波长数量仅为10个, 占全波段的4.7%, 极大的降低了模型的运算时间, 而且选取的特征波长中, 70%都位于特征吸收带内, 其分布可以较好的反应植被化学成分差异导致的光谱吸收特征规律。 实验结果表明利用光谱变换、 特征选择后建模的高光谱植被识别是可行的, 可以为其他湿地植被识别方法提供参考。
The identification of wetland vegetation using hyperspectral data has traditionally been one of the focuses of vegetation remote sensing research. Hyperspectral remote sensing data contains more detailed spectral features of vegetation, providing a powerful means for identifying hyperspectral vegetation. In this paper, the hyperspectral data of three typical wetland vegetation species, Zizania latifolia; Phragmites australis Salvinia natans; They were measured as target samples in the study area of the east coast of Erhai Lake. The original spectra were transformed by first-order differentiation and envelope removal and analysed for their spectral features. The feature wavelengths in the original spectra and their transformed spectra were selected using two feature variable selection algorithms, namely, successive projection (SPA) and competitive adaptive reweighted sampling (CARS), and the support vector machine (SVM) and random forest (RF) were finally established based on the full-wavelength data as well as the feature wavelengths after the selection-Recognition models. The results show that both SPA and CARS algorithms have a good dimensionality reduction effect on hyperspectral data, and the number of selected feature wavelengths is between 5 and 18. Comparing the combination of different spectral transform processing and feature wavelength extraction methods for modelling experiments, the envelope removal-SPA-SVM model performs the best in identifying the three types of target samples, with a recognition accuracy of 0.937 5. At this time, the number of feature wavelengths selected for input modelling is only 10, which accounts for 4.7% of the full wavelength range, which greatly reduces the model's computation time and of the selected feature wavelengths, 70% of the selected characteristic wavelengths are located in the characteristic absorption bands. Their distribution can better reflect the spectral absorption characteristic law caused by the differences in the chemical composition of vegetation. The experimental results show that the hyperspectral vegetation identification modelled by spectral transformation and feature selection is feasible and can provide a reference for other wetland vegetation identification methods.
湿地、 森林以及海洋合称为全球三大生态系统, 而湿地被誉为“ 地球之肾” , 对保护物种多样性、 调节河川径流、 促进沉积物沉降、 改善水质和维持区域水循环等有重要作用[1]。 高光谱数据具有波段窄、 波段多等特点, 容易获取地物的局部精细信息, 对光谱细节特征具有良好的表现能力, 在分析湿地植被的反射光谱差异性方面有较大的潜力[2]。
利用高光谱数据可以捕捉湿地地物目标的细微光谱响应差异[3], 对不同植被进行精准识别。 王美玲等[4]测取了曹妃甸湿地的两种典型植被芦苇和翅碱蓬的高光谱数据, 并对其展开时间差异和种间差异分析, 得出了更适合区分植被的光谱区间; 杨迈等[5]围绕滇池测取了芦苇、 香蒲、 菰和风车草的高光谱数据, 基于光谱“ 三边” 参数构建多元逐步回归模型, 并在氮含量估算模型中获得最大精度; 李世波等[6]基于东洞庭湖自然保护区的典型植被的实测光谱数据精细降维与分类处理, 得出了随机森林和径向基内核支持向量机保持较高的稳定性的结论。 目前针对淡水湿地植被光谱分类研究的成果较少。 湿地植被光谱特征存在较多的复杂与不确定因素[7], 植被光谱相似度高, 植被间反射光谱曲线的差异性不明显。 不同种类植被的光谱特征曲线受生化组分和冠层结构等因素的影响, 表现出一定程度的差别[8]。
以洱海海滨东岸菰、 芦、 槐叶蘋3种典型植被高光谱数据为研究对象, 对原始光谱作一阶微分和包络线去除变换扩大其光谱特征, 采用连续投影算法(successive projections algorithm, SPA)、 竞争性自适应重加权采样(competitive adaptive reweighted sampling, CARS)两种变量选择算法选取原始光谱与变换光谱的特征波长, 基于特征波长数据分别建立支持向量机(support vector machine, SVM)、 随机森林(random forest, RF)和径向基神经网络(radio basis function neural network, RBF)识别模型, 对比建模结果找出最准确、 高效的湿地植被高光谱识别方法。 在前人的研究经验中, 湿地植被存在较多不确定因素, 且植被光谱的相似度较高, 所以关键是寻找出能够放大湿地植被光谱特征的光谱变换办法以及筛选出鲁棒性更强、 识别效率更高的模型。
研究区位于云南省大理州洱海东岸海滨(金梭岛附近), 洱海(25° 36'— 25° 58'N, 100° 06'— 100° 18'E)发源于洱源的茈碧湖, 是云南省第二大淡水湖泊, 其水域面积约为253 km2, 径流面积达2 565 km2, 南北长达40.5 km, 东西宽约在3~9 km之间, 湖岸线长129.14 km, 而在水域中岛峭面积为0.748 km2, 主要有巷山十八溪、 金星河、 波罗江、 罗时江、 弥直河和永安江等河流和溪水流入[9]。 通过调查并结合已有资料, 洱海湖滨带有维管植物47科108属145种, 含属种数较多的科有禾本科、 菊科、 莎草科、 水鳖科等。 浮叶植物7种、 沉水植物26种以及湖滨带乔灌木十五种[10]。
高光谱影像数据的采集时间为2023年9月9日上午十点到下午两点之间, 飞行条件良好。 数据图像采用大疆M300RTK型无人机搭载FigSpec60高光谱成像仪获取。 无人机设置飞行高度为100 m, 设定航速7 m· s-1, 航向和旁向图像重叠度85%。 FS60高光谱相机的像素为5.86 μ m, 图像大小1 920× 1 080, 地面分辨率约5 cm。 飞行前采集白板数据用于辐射校正。
对采集到的光谱图像使用FigSpec1.0软件进行裁剪拼接, 再利用FigSpec Studio软件进行反射率矫正, 将得到的图像作为原始信号。 接着使用FigSpec Studio获取感兴趣区, 每个感兴趣区选择10个植被特征明显的像素, 通过计算植被的平均光谱反射率, 以绘制光谱曲线。 由于受测量时环境及传感器设备等因素的影响, 所测光谱存在抖动和噪声, 因此去掉受干扰严重的光谱波段, 截取后的光谱波段范围为400~850 nm, 然后采用SG平滑(Savitzky-Golay smoothing)进行平滑降噪, 得到原始光谱。
在光谱分析中, 单一的原始光谱曲线有时对光谱特征的反映不够突出, 故采用一阶微分及包络线去除法分别对原始光谱进行光谱变换。 一阶微分变换能减少背景噪声的影响、 增强光谱曲线斜率的细微变化, 强化谱带特征[11], 计算公式为
式(1)中, R'(λ i)为波长λ i处的一阶微分光谱; R(λ i+1)、 R(λ i-1)为波长λ i+1、 λ i-1处的原始光谱反射率; Δ λ 为波长λ i+1、 λ i-1之间的差值。 包络线去除变换在野外高光谱应用中表明, 能有效突出光谱曲线吸收特征[12]。 计算公式为
式(2)中, CR(λ i)为λ i处的包络线去除值; R(λ i)为λ i处光谱反射率; RH(λ i)为包络线上波长为λ i的光谱反射率。
高光谱数据量大, 冗余度高, 可能会影响模型运行的效率与准确度, 因此使用特征波长选择算法对数据进行降维。 采用SPA、 CARS两种特征选择算法分别选取原始光谱及两种变换光谱的特征波长, 将全波段与所选取的特征波长数据作为模型的输入变量。 SPA是使矢量空间共线性最小化的前向变量选择算法, 通过将波长投影到其他波长上, 选择投影向量最大的波长为待选波长, 然后建立多元线性回归模型选出最佳特征波长[12]。 使用SPA算法时设置特征波长为2~25个。 CARS是基于蒙特卡洛采样和偏最小二乘模型(partial least square, PLS)回归系数的特征波长选择方法, 采用自适应加权采样保留PLS模型中回归系数权重值较大的波长变量再次建立PLS模型, 选择交叉验证均方根误差最小的子集作为最佳特征波长变量子集[13]。
为了准确评估模型的识别效果, 采用Kennard-Stone算法[14]划分样本集, 将其按照2∶ 1的比例分为训练集和预测集。 随后, 我们基于训练集的样本数据建立了三种不同的植被分类模型: 支持向量机(SVM)、 随机森林(RF)以及径向基(RBF)神经网络。 接着, 我们利用这些模型对预测集的样本进行识别, 将识别精度作为评价模型的标准。 识别精度被定义为正确识别的预测集样本数与预测集样本总数的比值。 当识别精度接近1时, 表明模型的识别效果越好。 通过比较不同模型的识别精度, 我们可以选出最优的识别方法。
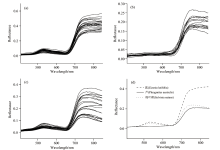
三种主要植被样本的原始光谱如图1(a, b, c)所示, 图1(d)为均值曲线。 从图中可以看出, 三种植被的光谱曲线变化走势基本相同, 但是由于其所含的叶绿素、 水分等生化成分不同, 导致其在一些波段上的光谱特征有所差异。 菰的总体反射率在0.01~0.56之间, 芦的反射率在0.01~0.26之间, 槐叶蘋的反射率在0.015~0.37之间。 三种植物在450和650 nm附近都形成了吸收谷, 在650~750 nm的红边波段附近, 反射率都急剧上升, 形成明显差异, 其中菰在这些吸收带中的特征差异最为明显。 芦与槐叶蘋在750 nm附近还存在一个弱吸收谱带。 在550 nm附近, 菰与槐叶蘋形成一个反射峰, 而芦在图中基本无法判断, 这主要是由于三种植物体内所含叶绿素浓度引起的, 这也与此季节肉眼观察芦为紫色相一致。
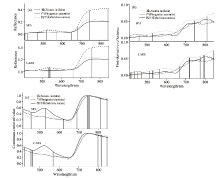
图2(a)和图2(b)分别是对原始光谱进行一阶微分和包络线去除变换后的光谱曲线。 在图2(a)中, 其纵坐标表示原始光谱曲线的变化速率, 正值表示正变化速率, 负值反之, 这样可以凸显出光谱曲线的曲率变化。 在进行包络线去除变换后的图2(b)中, 光谱反射率归一化到0~1之间, 形成了一些较为明显的吸收谷, 放大了光谱曲线的特征。
使用SPA算法与CARS算法分别对样本的原始光谱、 一阶微分光谱、 包络线去除光谱进行特征波长的选取, 选取出的特征波长在整个波段的分布如图3(a, b, c)所示。 两种算法从原始光谱中分别选取10个和6个特征波长, 分别占全波段的4.7%和2.8%; 从一阶微分光谱中分别选取5个和18个波长, 分别占全波段的2.3%和8.6%; 从去包络光谱中分别选取了10个和8个特征波长, 分别占全波段的4.7%和3.8%。 在与原始数据进行对比以后发现, 两种特征波长选择算法对原始数据都有很好的降维效果。
所选的特征波长分布可以比较好的对应植被光谱的吸收特征区间。 以原始光谱为例, SPA算法从原始光谱中选择了10个特征波长, 其中两个分别位于中心波长为450与650 nm附近的红蓝谱带内, 一个位于540 nm反射峰附近, 两个位于红边波段内, 特征谱带附近的特征波长数量占了总特征波长数的60%; 而CARS提取的特征波长中特征谱带附近的特征波长所占的比例达到了83%。 表明植物体内生化含量差异所导致的光谱特征规律对构建识别模型有重要作用, 是光谱信息识别植被类型的基础。
对训练集原始光谱、 一阶微分光谱和去包络变换光谱的全波段光谱(full-band spectrum, FS)数据, 以及经过SPA/CARS算法选取出的特征波长光谱数据, 分别建立SVM、 RF以及RBF的植被识别模型。 SVM模型采用径向基函数作为核函数, 使用交叉验证和网格搜索方法寻找最佳惩罚因子c和核函数g。 RF模型使用交叉验证和学习曲线方法寻找最佳决策树数量n以及叶子数量f。 在RBF神经网络模型中, 扩展速度(spread)值变大会使得拟合越平滑, 但逼近误差也会增大, 变小则反之, 所以需要通过对不同spread值的尝试寻找出适应模型的参数(RDF_spread, rs)。 不同模型的确定参数见表1。 模型对预测集的识别结果见表2。
| 表1 不同SVM和RF模型的参数 Table 1 Parameters of different SVM and RF models |
| 表2 基于不同特征波长选择方法建立模型的识别结果 Table 2 Recognition results of models based on different characteristic wavelength selection methods |
由表2可以看出: 在SVM和RF模型对全波段数据的识别结果中, 原始光谱的识别精度分别为0.842 1和0.789 4, 一阶微分变换后光谱的识别精度都为0.894 7, 包络线去除变换后光谱的识别精度分别为0.875 0和0.842 1。 经过变换处理后的光谱的识别精度相较于原始光谱明显提高, 这说明对原始光谱进行适当的光谱变换可以提升建模的精度。
RBF模型的训练时间明显低于其他两种模型, 且包络线去除-CARS-RBF的识别模型精度达到了0.904 7, 这可能归功于RBF模型的局部逼近特性, 在处理复合问题具有较强的适应性。 但是对全波段特征提取之后的识别精度相较于原始光谱却有一定的下降, 这可能是由于全波段包含的噪声大于特征提取后波段造成的。
对特征波长光谱数据的识别结果中, 识别精度最高的是去包络变换-SPA-SVM模型, 识别精度达到了0.937 5, SPA共选取出了10个波长, 其中3个分别位于450、 550和650 nm附近的蓝绿红谱带, 4个位于760 nm的红边波段附近, 因此该模型可以较好地反映植被生化成分差异导致的光谱吸收特征规律。 识别精度最低的是一阶微分-CARS-RF模型, 识别精度为0.684 2, 经分析认为一阶微分光谱在较多波段的共线性比较高, 而且模型的超参数还有待调整, 信息量较少, 导致最终的建模效果欠佳。
针对洱海东岸海滨三种典型植被测得的高光谱数据, 对其原始光谱进行一阶微分和去包络变换并分析其光谱特征, 采用SPA和CARS分别选取原始光谱以及两种经过光谱变换后的特征波长, 然后将特征波长提取前的全波段数据以及特征波长选取后的部分波长数据作为变量输入, 构建了植被识别模型, 对比不同组合的精度选取出最佳的建模方法。 结果表明: (1)在以全波段数据为基础建立的识别模型中, 一阶微分-FS-RF模型与一阶微分-FS-SVM的识别精度最高, 都达到了0.894 7, 适当的光谱变换可以有效提升全波段数据的建模效果。 (2)相比于用全波段数据进行建模, 部分特征波长选取算法可以在有效减少建模输入量的同时, 提升建模的精度, 保证模型的效果优越。 有的虽然减少了模型的输入量, 但使得模型的效果降低, 不利于对模型进行优化。 包络线去除变换-SPA-SVM模型的表现最好, 其识别精度达到了0.937 5, 而输入模型的波长数量只占全波段的4.7%, 能够准确、 迅速的对湿地植被进行精准识别。
针对洱海东岸海滨三种植被的光谱分析与识别方法对于推动高光谱数据在湿地植被分类识别方面的应用具有重要意义。 同时也为湖泊湿地植被及湖泊生态环境的保护决策提供了科学依据。 然而, 植被光谱分析常常受到大气空间和时相变化的影响。 在本实验中, 我们只使用了单一时期的样本数据, 这可能会限制识别模型的适用性。 因此, 在未来的研究中, 我们将增加不同时期的样本数据, 并尝试采用更具创新性的光谱分析方法, 以提高模型的普适性和精度。 这将有助于更准确地捕捉湿地植被光谱特征, 并提升模型在不同条件下的可靠性, 从而更有效地支持湖泊湿地生态环境的管理与保护工作。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|