作者简介: 张微微,女, 1984年生,滁州学院生物与食品工程学院副教授 e-mail: 249541998@qq.com
牛奶因其丰富的营养成分和易消化吸收的特点, 受到消费者的青睐。 牛奶掺杂行为的产生使得牛奶制品质量备受关注, 快速、 便捷地鉴别乳品质量对于乳制品行业经济的健康发展具有重要意义。 利用同步荧光光谱对掺杂牛奶进行检测, 寻求一种高效判别掺杂乳品方法。 采用分子荧光分光光度计测定激发波长(Ex)为220~600 nm, 激发-发射间隔波长(Δ λ)为10~180 nm的纯牛奶、 复原乳粉(全脂、 脱脂)及其掺杂样本的牛奶样品的三维荧光光谱数据, 利用平行因子分析方法(PARAFAC)降维获取特征光谱, 通过支持向量机建立了掺杂牛奶的判别模型。 结果所有乳品样品在激发波长为225~300 nm范围内都有一个特征荧光峰, 荧光峰在280 nm附近, 为色氨酸类物质, 但该处纯牛奶荧光强度明显高于两种乳粉, 且脱脂乳粉要强于全脂乳粉, 这说明牛奶的主要成分都一样, 发光基团一致, 但由于处理方式不一致, 使得其浓度存在差异。 两种复原乳粉在350~400, 450~500 nm之外存在荧光物质, 主要为维生素A和类胡萝卜素, 且脱脂乳粉比全脂乳粉对应区域荧光强度要强, 主要在于脂肪物质散射使得荧光强度增强。 为更好获取乳品样本特征, 通过PARAFAC对三维数据进行降维之后, 显示当组分数为6, Δ λ为40 nm时载荷值最大, 该处样本信息差异显著。 提取Δ λ为40 nm特征波长和掺杂乳品品类值作为输入数据的支持向量机(SVM)分类器, 采用了遗传算法(Ga-SVM)、 粒子群优化算法(Pso-SVM)和网格搜索算法(Grid-SVM)三种SVM算法对掺杂牛奶进行分类识别。 结果显示Grid-SVM模式交叉验证(CV)准确率为98.91%, 其训练集和测试集的分类准确率均为100.00%, 且模型运行时间仅6.724 s, 显著优于另两种分类器。 结果表明荧光光谱与PARAFAC-SVM方法相结合, 是一种简单且高效判别掺杂牛奶的方法。
Milk is favored due to its high nutritional value and consumption rate. Authenticity is a common concern for value assessment. Recently, non-invasive and rapid identification methods have been preferred for the dairy industry. This work proposed a quick method using synchronous fluorescence (SF) spectroscopy and a support vector machine (SVM) for the identification of raw milk. With this aim, SF spectra of milk were recorded between 220 and 600 nm excitation range with Δ λ of 10 to 180 nm, in steps of 10 nm. All the milk showed the same fluorescence excitation at band position 280 nm, which corresponded to tryptophan. However, the fluorescence intensity of pure milk at this location was significantly higher than that of the two types of milk powder, and skimmed milk powder was stronger than whole milk powder. It indicated that the same main components were in milk. However, there were differences in their concentrations by different treatment methods. Two types of reconstituted formula milk were differentiated based on intensity variations at wavelengths 350~400 and 450~500 nm. The excitation at these wavelength positions corresponds to vitamin A and carotenoids. At these bands, the skimmed milk powder had a stronger fluorescence intensity in the corresponding region than whole milk powder, mainly due to the scattering of fatty substances, which enhanced the fluorescence intensity. Parallel factor analysis (PARAFAC) was found to reduce three-dimensional SF spectroscopy to two-dimensional data, resulting in a better understanding of the characteristics of dairy products. When the suitable components were6, the maximum load value was at Δ λ with 40 nm, where the difference in sample information was more significant. Then, the Δ λ with 40 nm and the value of contaminated dairy products as input data were used to classify and identify adulterated milk for the support vector machine (SVM) classifier. The three SVM methods were the genetic algorithm for support vector machine (Ga-SVM), particle swarm optimization support vector machine (Pso-SVM), and grid search algorithm (Grid-SVM). The results showed that the optimal classification accuracy for the Grid-SVM mode training set, test set, and cross-validation (CV) accuracy were 100.00%, 100%, and 98.91%, respectively, with a model running time of only 6.724 seconds. The study demonstrated that SF spectroscopy with PARAFAC and SVM methods is a promising tool and can potentially become a rapid and nondestructive analytical technique for identification of adulteration milk.
牛奶是常见的乳制品之一, 其丰富的营养价值使得人们对乳制品的需求量与日俱增。 为了牟取高额利润或减少供需差距, 不法商贩在牛奶中掺入水、 混加入一些乳粉或者其他动物奶等[1, 2]。 这些掺杂行为使得消费者和食品供应链企业更加注重乳品品质。 迄今为止, 已有多种技术用于检测牛奶样本的掺杂情况, 例如色谱法[3]、 PCR[4]、 电泳法[5]等, 但这些技术中样本制备步骤复杂且耗时, 而且仪器操作需要专业人员, 更不适宜规模工业化应用。
荧光光谱技术因其快速、 简单和高效的特点被广泛应用于食品分析, 其在乳品领域中主要用于鉴别牛奶来源, 定性定量检测奶中物质等[6, 7, 8]。 与传统荧光光谱技术相比, 同步荧光光谱在发射和激发波长之间保持恒定的间隔波长(Δ λ )进行扫描, 提高了荧光光谱的分析能力[9], 因而具有高选择性和低干扰性。 Velioglu等利用同步荧光光谱结合多变量方法快速鉴别和检测水牛奶和牛奶掺假[10]。 Genis等通过同步荧光光谱方法鉴别发酵乳制品中牛、 水牛、 山羊和母羊乳种[11]。 由于牛奶复杂的检测环境, 机器学习算法可以更好提取荧光光谱数据中特征信息从而提高判别准确率。 支持向量机(support vector machines, SVM)是基于统计学理论的一种新的机器学习算法, 具有较好的分类性能, 常用于分析小样本。
针对牛奶分析精度和方法的需求, 采用同步荧光光谱技术结合三个基于SVM的优化算法建立掺杂牛奶的判别模型, 综合比较各模型性能, 寻求一种运行时间短、 分类准确度高的最优方法, 为快速、 高效判别牛奶真伪提供参考。
荧光分光光度计(Cary Eclipse, 美国瓦里安有限公司); 超纯水机(UP300-E UAF, 上海和泰仪器有限公司); 乙酸-乙酸钠缓冲液: 分析纯, 国药集团化学试剂有限公司。
纯牛奶、 复原乳粉(全脂、 脱脂)由滁州当地小牧场提供, 其中复原乳粉为非商用乳粉, 经处理后牛乳直接喷粉获得。
掺杂样品的配制: 将乳粉与纯牛奶按1∶ 1、 1∶ 2、 1∶ 3, …, 1∶ 20(V/V)比例混合, 共138个样本。
将0.5 mL上述乳品样品加入4.5 mL乙酸-乙酸钠缓冲液(0.1 mol· L-1, pH 4.6)中, 以4 500 r· min-1离心15 min, 用1 mL的注射器吸取离心管中的上清液, 用针筒过滤器进行过滤(滤膜为0.22 μ m), 4 ℃下储存, 备用[12]。
将滤液移入四面通光的石英比色皿, 使用荧光分光光度计, 设定同步荧光光谱扫描参数: 激发波长(Ex)扫描范围为220~600 nm, 激发狭缝为5 nm, 发射狭缝为5 nm, 扫描速度1 200 nm· min-1, 间隔2 nm。 激发-发射间隔波长(Δ λ )扫描范围为10~180 nm, 间隔10 nm。 每个样本采集记录3次并保存光谱数据, 取平均光谱作为样本分析光谱, 绘制等高线图。
1.5.1 数据降维
平行因子法(parallel factor analysis, PARAFAC)被证实是一种高效降维方法, 是通过最小化残差平方和(Eijk)来实现的[13]。 三维同步荧光光谱数据由激发光谱i、 荧光强度j和激发-发射间隔波长k组成三维数据, 记为i× j× k的三维响应矩阵X。 利用该方法将三维荧光光谱矩阵分解为3个载荷矩阵A、 B、 C, 数学表达式如式(1)
式(1)中, Xijk为三维数据矩阵X的一个元素; Ain、 Bjn、 Ckn分别为A、 B、 C中的元素; Eijk为误差矩阵; N为模型因字数, 即对应模型的最佳组分数。
数据降维在MATLAB 2014a中的DOMFluor工具箱环境下运行。
1.5.2 判别模型建立与性能指标评价
SVM建立非线性判别模型的有效分类技术。 SVM分类模型的有效性和结果除数据集性质影响外, 其核函数尤其径向基函数惩罚参数c和核参数γ 优化决定着整个模型的效率。 为避免机器学习过程存在的过拟合现象, 获得最佳参数, 可采用交叉验证(cross validation, CV), 以获得最好的分类精度[14]。 本项目采用遗传算法(genetic algorithm, Ga)、 粒子群优化算法(particle swarm optimization, Pso)、 网格搜索算法(grid search, Grid)等3种方法进行参数优化, 以提高支持向量机的分类性能。
项目将牛奶特征荧光光谱数据和牛奶类别值输入支持向量机分类器。 所有的数据随机打乱, 自行按照比例划分训练集和测试集, 通过训练数据训练支持向量机模型, 并通过测试数据对所构建的支持向量机模型进行评估。 利用分类准确率和耗时作为模型的评价指标。
数据在MATLAB 2014a中Libsvm工具箱环境下运行。 Libsvm工具箱由台湾大学林智仁教授实验室开发。
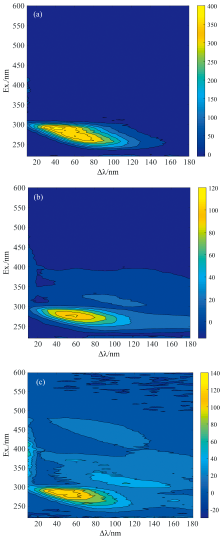
通过不同Δ λ 扫描激发光谱, 采集不同类别牛奶样品的同步荧光光谱, 研究牛奶荧光光谱特性, 结果见图1。 图1清晰显示三种类别牛奶荧光光谱存在显著差异。 三种牛奶在激发波长区225~300 nm有1个显著特征荧光峰, 最强峰位置为280 nm左右, 该物质为生色团色氨酸[15, 16]。 图1(a)纯牛奶该处荧光强度显然强于图1复原乳(b)和(c), 而且峰位置存在些许差异。 这说明牛奶中主要成分相同, 发光基团一致, 但由于发光团所处的环境不同, 导致总光谱形状出现差别[17]。 荧光区域激发波长为300~550 nm, 主要表示维生素A、 部分色氨酸、 核黄素和类胡萝卜素等组分[18]。 全脂乳粉在(Δ λ , Ex)为(70 nm, 323 nm)附近有1个次级荧光峰, 主要为β -胡萝卜素。 脱脂乳粉除却320 nm附近荧光峰外, 在(Δ λ , Ex)为(50 nm, 380 nm)还存在1个弱荧光峰, 主要表示色氨酸、 脂溶性维生素和β -胡萝卜素[19, 20]。 图中激发波长300~400 nm扫描区域显然发现脱脂乳粉荧光强度要高于全脂乳粉。 主要由于牛奶中的脂肪对荧光有散射作用, 且脂肪对长波段的荧光散射作用效果较明显, 光的散射作用使得荧光在溶液中通过的光程较长, 增加了被吸收的概率[21]。 因此, 在较长激发光激励下, 脂肪散射作用明显, 使得全脂乳粉荧光峰强度弱于脱脂乳粉[22]。 图1研究结果得出三维同步荧光光谱能较为全面的描述并区分乳制品类别, 清楚地表明了同步荧光光谱在牛奶成分鉴别方面的潜力。 由于乳样结构中的芳香族氨基酸和核酸含有共轭双键, 同步荧光光谱技术可以成功地用于乳品分析。
2.2.1 光谱数据组分数分析
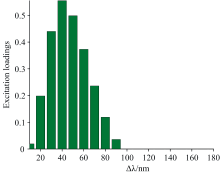
随着与光谱解析手段平行因子方法等的结合, 可以确定模型的最适组分数进一步解析三维荧光光谱[23, 24], 组分模型2到组分模型7的误差平方和结果见图2。 分析结果表明, 组分6和组分7的模型较为适合, 但考虑运行时间等综合选择组分6进行平行因子建模分析, 开展进一步的研究。 通过图3发现当组分为6时, Δ λ 为40 nm时模型的载荷值最大, 产生样本间的差异最显著。
2.2.2 二维荧光光谱分析
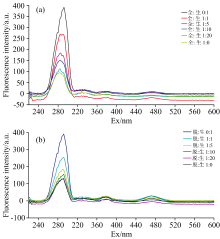
通过载荷得分分析, 使用PARAFAC法将同步荧光光谱的三维数据分解为二维数据, 牛奶掺杂不同比例复原乳样品的典型同步荧光激发光谱如图4(a, b)所示。 比较不同浓度的掺杂样本, 从图中很容易观察到所有掺混牛奶在250~300 nm波段具有相似的光谱峰。 除了光谱峰的分析外, 光谱强度在不同掺混的液体牛奶中也有显著的差异。 牛奶荧光光谱峰值大小随着纯牛奶中掺混物质的浓度增加而降低。 主要因为溶液浓度的改变引起发光基团浓度的改变, 荧光强度也相应改变。 而在激发波长为350~400和450~500 nm区域荧光峰位置和强度并无变化, 主要因为当激发波长增加超过色氨酸的最大激发波长时, 色氨酸对总荧光光谱的影响较小。 牛奶中物质位于280 nm波段位置具有高度选择性, 可以作为鉴别掺混牛奶的一个重要指标。 该二维光谱的结果显示与三维同步荧光光谱的解析一致。
为了将牛奶进行分类, 设定完好纯牛奶类别值为1, 掺杂全脂乳粉为2, 掺杂脱脂乳粉为3。 从牛奶样品中选取了138个样本, 分为一组训练样本(即92个样本)和一组测试样本(即46个样本)。 为了检测SVM模型的性能, 选取Grid-SVM模型、 Ga-SVM模型和Pso-SVM模型进行比较, 将PARAFAC提取的特征波长值和类别值作为输入, 通过综合考虑模型的训练效果与预测效果, 分类模型结果见表1所示。
| 表1 支持向量机分类器结果(Δ λ =40 nm) Table 1 The classification results by different SVM methods at 40 nm |
由表1可知, 3种优化模型的CV准确率均超过了95%, 说明PARAFAC法为有效的提取特征荧光光谱方法。
基于PARAFAC-Grid-SVM所建模型的CV准确率最高, 为98.91%, 该条件下训练集和测试集的分类准确率都为100%, 而Ga-SVM模型和Pso-SVM模型CV准确率分别为95.65%、 96.73%, 训练集分类准确率分别为100%、 98.91%, 但其测试集都为97.83%。 机器学习分类器结果训练集准确率与测试集准确率越高并且两者越接近, 表明模型的精度高、 可靠性好, 因此Grid-SVM判别模型最优, Pso-SVM模型次之, Ga-SVM模型最差。 但在运行时间上, Ga-SVM模型、 Pso-SVM模型和Grid-SVM模型的运行时间分别为19.453、 58.438和6.724, 很显然Grid-SVM最快, 仅为Ga-SVM模型的34.6%, Pso-SVM模型的11.5%。 综合考虑准确率和效率, Grid-SVM模型作为掺杂牛奶品类判别的最优模型。
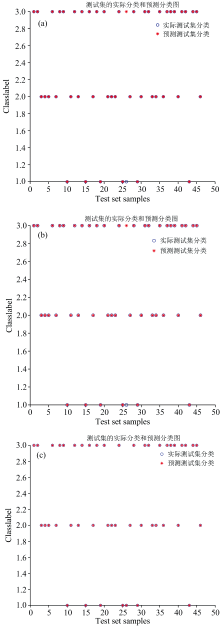
图5(a, b, c)分别是Ga-SVM模型、 Pso-SVM模型和Grid-SVM模型的预测判别结果。 图5可以看出Ga-SVM模型和Pso-SVM模型测试集的仅有1个被误判, 将纯牛奶判定为掺杂脱脂奶粉, 主要原因为该样本纯牛奶特征荧光基团强度与掺杂低浓度脱脂乳粉荧光峰强度缺少显著差别, 导致模型误判。
为了快速鉴别掺杂牛奶, 采用同步荧光光谱技术对牛奶样本进行判别, 将同步荧光光谱结合支持向量机分类器, 分别建立Ga-SVM模型、 Pso-SVM模型和Grid-SVM模型进行对比分析, 主要结论:
(1)三维同步荧光光谱能直观获取样品特征信息, 通过PARAFAC法对三维光谱数据降维分析得到Δ λ =40 nm特征波长, 并作为建模数据输入值, 用于后续的建模分析。
(2)采用Ga-SVM、 Pso-SVM和Grid-SVM等3种模式识别, 探讨判别模型性能。 结果表明Grid-SVM模型最优, 训练集、 测试集分类准确率均为100%, 且其交叉验证准确率为98.91%, 总体显著高于Ga-SVM和Pso-SVM模型。
(3)实际生产中, 确保模型准确度前提下, 效率也很重要。 在Ga-SVM、 Pso-SVM和Grid-SVM等3种判别模型中, Grid-SVM模型耗时小, 仅为6.724 s。 综合考虑准确度和效率, 选定Grid-SVM模型为鉴定牛奶类别的最理想方法。
荧光光谱技术结合PARAFAC和SVM是一种快速、 非破坏性的具有高度的训练和测试准确度的鉴别掺混牛奶方法。 研究结果也可为其他食品种类判别提供一条便捷、 无损且精准的有效途径。
从模型准确率及误判结果分析, 仍存再进一步深入研究问题, 牛奶成分复杂的光谱检测环境需要引入多元统计方法提高检测精度、 灵敏度和检测范围, 以期为精准识别掺杂物质提供技术支持。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|