作者简介: 李 浩, 1994年生, 中国农业大学“智慧农业系统集成研究”教育部重点实验室博士研究生 e-mail: caulh@cau.edu.cn
卷积神经网络(CNN)在数据特征提取方面具有巨大优势, 能充分获取数据特征, 相较于传统模型具有更好的泛化性。 基于CNN开展了土壤有机质(SOM)含量高光谱预测方法及模型研究。 以北京市昌平区上庄实验站的320个土壤样本为研究对象, 提取可见光-近红外(VIS-NIR)350~1 700 nm内的807个光谱波段, 通过多元散射校正(MSC)和一阶微分变换进行光谱数据去噪和变换。 分别使用连续投影算法(SPA)、 竞争性自适应重加权算法(CARS)筛选敏感波长实现光谱数据降维。 为解决传统手段泛化性差以及深层CNN网络复杂且负载过大的问题, 基于CARS与SPA算法, 提出一种基于6层卷积层的浅层CNN模型预测, 并对比具有不同卷积尺寸和卷积数量的1D-CNN1、 1D-CNN2以寻找最优网络参数。 通过对比VGG16、 支持向量回归(SVR)、 最小二乘回归(PLSR)、 随机森林(RF)建立预测模型在特征波长以及全波段的表现确定最佳模型。 结果表明, 相比于全谱波段和SPA筛选算法, 基于CARS筛选特征波长建立的模型整体表现更好, 波段数量被压缩至全波段的8%, 有效实现了光谱数据的降维。 对比全波段数据, 基于CARS筛选波长的1D-CNN1、 1D-CNN2的表现更好, 模型预测 R2分别提升了0.028, 0.018; RMSE分别降低了0.150和0.107 g·kg-1。 整体上, 基于CARS的1D-CNN1模型表现最好, 预测 R2=0.846, RMSE=3.145 g·kg-1, 降低了网络负载的同时提高了模型精度, 同时也证明了小尺寸卷积的表现优于更多数量的大尺寸卷积, 能够更好的获取数据特征。 通过CARS筛选特征波长结合浅层CNN建立SOM含量预测模型, 为建立高精度的SOM含量预测模型提供了方法与参考。
Convolutional Neural Network (CNN) has a great advantage in data feature extraction, as it can fully acquire data features and has better generalization than traditional models. This study used a hyperspectral prediction method and modeling of Soil Organic Matter (SOM) content based on CNN. Using 320 soil samples from Shangzhuang Experimental Station, Changping District, Beijing, 807 spectral bands within 350~1 700 nm in the visible-near-infrared (VIS-NIR) were extracted, and the spectral data were denoised and transformed by the multivariate scattering correction (MSC) and the first-order differential transform. Successive projection algorithm (SPA) and competitive adaptive reweighted Sampling (CARS) were used to screen the sensitive wavelengths to realize the dimensionality reduction of the spectral data, respectively. To solve the problems of poor generalization of traditional means as well as the complexity and overload of deep CNN networks, based on the CARS and SPA algorithms, a shallow CNN model prediction based on 6 convolutional layers is proposed, and 1D-CNN1 and 1D-CNN2 with different convolutional sizes and number of convolutions are compared to find the optimal network parameters. By comparing the performance of VGG16, Support Vector Regression (SVR), Partial Least Squares Regression (PLSR), and Random Forests (RF) to build a prediction model in the feature wavelength and the full waveform. The optimal model was determined. The results show that compared with the full-spectrum band and SPA filtering algorithms, the model based on CARS filtering feature wavelength modeling performs better, and the number of bands is compressed to 8% of the full-wavelength band, which effectively realizes the dimensionality reduction of the spectral data. Comparing the full-band data, 1D-CNN1 and 1D-CNN2 based on CARS screening wavelengths performed better, with the model predicted R2 improved by 0.028 and 0.018, respectively, and the RMSE reduced by 0.150 and 0.107 g·kg-1, respectively. Overall, the 1D-CNN1 model based on CARS performs the best, with the predicted R2=0.846 and the RMSE decreased by 0.150 g·kg-1, respectively 0.846, and RMSE=3.145 g·kg-1, which reduces the network load while improving the model accuracy, and also proves that small-size convolution outperforms a larger number of large-size convolutions for better acquisition of data features. The SOM content prediction model is established by CARS screening feature wavelengths combined with shallow CNN, which provides a method and reference for establishing a high-precision SOM content prediction model.
利用光谱学分析方法检测土壤有机质(soil organic matter, SOM)含量是替代传统化学检测方法的有效手段之一[1, 2, 3]。 近些年, 基于光谱学原理预测SOM含量备受关注, 相关研究也取得了诸多进展。 但相较于传统化学方法, 已有手段在精度上仍然存在一定差距; 同时受限于土壤组成的复杂性, 面对较大的数据样本, 传统建模方法也存在着模型精度下降的情况。 提高模型的预测能力和泛化性也成为了当下研究的重点[4, 5]。 整体上, 基于光谱学原理预测SOM含量主要分为数据预处理、 敏感波长选取和建立模型三个部分[6]。 预处理通过去噪、 微分变换等手段对原始光谱进行转换, 有助于消除不相关因素对光谱曲线的干扰, 提升真值与光谱波段的相关性。 敏感波长的选取能够有效实现光谱数据降维, 避免数据冗余[7, 8]。 选择更好的建模方法, 对于提高预测结果精度和提升模型的泛化性具有决定性影响。
国内外已有诸多学者在可见光-近红外区间内对SOM含量预测展开相关研究, 主要围绕消除外在因素干扰、 寻找最佳建模方法、 减少光谱数据冗余三个方面开展研究。 Guindo等使用粒子群算法筛选光谱特征, 实现了光谱数据的有效降维, 证明该方法可以提升模型的预测精度[9]。 章涛等在高光谱数据预处理阶段使用小波能量去除高光谱数据的噪声, 并对比未处理数据建立模型, 证明该方法对模型预测效果具有一定提升, 模型最终预测R2=0.77[10]。 孟珊等[11]通过使用竞争性自适应重加权算法(competitive adaptative reweighted sampling, CARS)将光谱波段数量降低了97%, 结合BP神经网络实现了SOM的高效预测。 Liu等[12]通过连续投影算法(successive projections algorithm, SPA)实现光谱降维, 对比全波段数据建立多种预测模型, 最终证明基于SPA降维后的波段结合RF预测效果最佳, R2=0.82。 然而, 以上工作都是基于小样本数据, 模型的泛化性仍需要进一步验证。 光谱数据与有机质含量之间的关系较为复杂, 仅考虑二者之间的线性关系容易导致模型的精度受限[13]。 随着数据样本的增加, 模型的性能也会受到影响, 结合硬件实地测试, 模型精度也会下降, 限制了相关手段的推广与应用。 相比之下, 深度学习模型具有泛化性强的特点, 能更充分地挖掘到数据特征, 模型的实际应用性更强。 钟亮对比多种经典深度学习模型预测SOM含量, 证明网络层数更少的VGG模型表现最优, 具有更好的泛化性, 相较于传统机器学习模型优势明显, 但较深层CNN模型也容易出现性能下降和运行效率慢的情况[14]。
整体上看, 已有研究主要通过预处理方法结合机器学习模型, 或是通过CNN模型完成SOM含量的预测。 相较于传统机器学习方法, CNN模型展现出了更好的泛化性和预测能力, 但是CNN模型广泛存在的网络复杂和模型负载较大的情况依然未得到有效解决, 相关工作仍需要开展更加深入的研究[15]。 在此基础上, 以SPA以及CARS作为波段筛选算法结合CNN展开研究, 旨在建立具有更高效率和更强预测能力的CNN预测模型。
实验地点位于中国农业大学海淀区上庄实验站(40.141 1° N, 116.189 4° E), 实验区属于温热带季风气候, 夏季高温多雨, 秋冬季寒冷干燥, 年平均气温在8~13 ℃左右, 年均降雨量500 mm, 主要种植小麦、 玉米。 共随机采集土壤样本320份, 每份样品挖取5~15 cm深度浅层土样300 g, 用牛皮纸袋密封后带回实验室分析。 采回的样品在24 h内完成过筛、 去除杂质、 烘干、 研磨、 称量等步骤。 使用芬兰SPECIM公司生产的Gaia Sorter成像式高光谱仪, 搭配了400~1 000和900~1 700 nm镜头, 光谱分辨率分别为2.8和5 nm; 样本放置于移动台实现扫描式拍照成像, 可采集350~1 700 nm区间的高光谱数据。 结合已有研究[16], 土壤高光谱数据选择(Savitzky-Golay, SG)滤波进行平滑预处理, 采用多元散射校正(multiplicative scatter correction, MSC)对原始光谱进行去噪, 并使用一阶微分对原始数据进行变换。
使用灼烧法测定SOM含量, 该方法准确且易于操作, 首先称量烘干后的4 g土样放入燃烧炉中在660 ℃燃烧4 h后静置放凉, 随后称量计算燃烧前后差值即为土壤有机质含量。 表1为所采集的320份土壤样本SOM含量的统计结果, 按照8∶ 2的比例分为建模集和测试集, 样本整体SOM含量区间为9.3~46.4 g· kg-1, 建模集样本区间为9.5~46.4 g· kg-1, 测试集样本区间为9.3~43.2 g· kg-1, 建模集涵盖了预测集的含量范围, 总体变异系数为43%, 属于中等变异。
| 表1 土壤SOM含量统计 Table 1 Soil organic matter content statistics |
在已有研究的基础上, 采用目前表现较好的竞争性自适应重加权算法(CARS)和连续投影算法(SPA)对全谱数据进行敏感波长的筛选。 CARS通过自适应重加权采样(ARS)技术剔除权重最小的波长点, 对回归系数绝对值最大的点进行提取, 结合交叉验证均方根误差(root mean square error cross validation, RMSECV)与交叉验证(cross validation, CV)进行子集选取完成最优变量组合的选取。
SPA算法以均方根误差(root mean square error, RMSE)为指标, 选取矢量空间共线性最小化前向变量, 通过计算RMSE最小值对应的波长个数确定敏感波段个数。 敏感波长筛选算法能够有效减少全谱波段数量, 剔除不相关波段的影响, 有效提升模型的运算精度和效率。
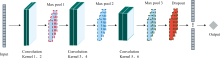
与传统算法相比, CNN具有强大的数据特征提取能力和更好的泛化性。 近些年来, CNN在SOM预测领域也展现出了巨大的优势, 取得了相较于传统机器学习模型更好的表现。 整体上看, CNN由输入层、 卷积层、 激活层、 池化层和全连接层组成。 针对卷积层数过深的网络在光谱数据处理中易出现模型效率低下、 性能下降的情况, 设计了一种具有6层卷积网络的1D-CNN光谱预测模型。 1D-CNN模型结构如图1所示。
模型使用ReLU激活函数提升卷积网络性能, 引入Dropout方法进行结构优化, 学习率设为0.001, 设置优化器Adam。 为研究卷积核尺寸与数量对模型的性能影响, 设计1D-CNN1与1D-CNN2两种模型对比分析。 1D-CNN1和1D-CNN2模型结构相同, 只在卷积尺寸和数量上进行差异化设定。 具体参数如表2所示。 实验环境通过Python中的sk-learn库完成搭建, 实验平台硬件运行内存为16G, CPU型号为i7-12700, GPU型号为RTX3060(6G)。
| 表2 卷积神经网络参数 Table 2 Convolutional neural network parameters |
基于成像高光谱相机完成土壤光谱反射率的采集, 通过ENVI软件实现数据黑白校正后, 选择图像中心区域作为敏感区域进行光谱反射率的提取, 得到土样在350~1 000和900~1 700 nm波段的反射率数据。 根据SOM的含量高低, 将SOM含量分为3个等级, 分别为等级Ⅰ (0 g· kg-1≤ SOM< 20 g· kg-1)、 等级Ⅱ (20 g· kg-1≤ SOM< 40 g· kg-1)、 等级Ⅲ (40 g· kg-1≤ SOM< 50 g· kg-1)。 图2为剔除边缘等无效波段后, 不同等级在350~1 700 nm区间内的VIS-NIR光谱均值曲线。 由图2可知, 不同SOM含量的VIS-NIR光谱反射曲线形态基本一致, 在1 400 nm附近存在明显的水分波谷。
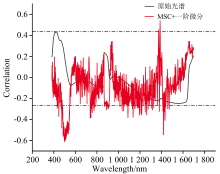
针对原始光谱数据以及经过变换后的光谱数据开展了与SOM实测值之间的相关性分析, 结果如图3所示。 原始光谱数据与SOM含量相关性绝对值最高为0.4, 大部分波段相关系数绝对值在0.2以内, 主要敏感区间为420~540、 860~940和1 630~1 680 nm。 经过多元散射校正与一阶微分变换后, 波段的整体相关性明显提升, 绝对值最高达到0.6, 主要区间为450~600、 750~790、 950~980、 1 350~1 490和1 620~1 670 nm, 基本涵盖了原始光谱的敏感区间波段。
经过运算, CARS最终迭代次数为18时RMSECV值最小, 总波段数从807降为64个, 特征波长数量占原始总波长数量的8%, 过程如图4所示; 采用SPA算法对高光谱数据进行敏感波段选取, SPA将总波段数量由807降为48个, 特征波长数量占原始总波长数量的6%。 SPA算法运行过程如图5(a)所示。
 | 图4 CARS敏感波段筛选 (a): 运行次数与变量数值; (b): 运行次数与误差分析Fig.4 CARS characteristic band screening (a): Number of runs and variable values; (b): Number of runs and error analysis |
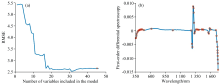 | 图5 SPA敏感波段筛选 (a): 运行次数与变量数值; (b): 一阶微分处理后的敏感波段分布Fig.5 SPA characteristic band screening (a): Number of runs and variable values; (b): Distribution of Sensitive bands after 1st derivative processing |
基于CARS与SPA的敏感波长筛选分布与重要性的结果如图6和图7所示。 从图6中可以看到, 基于CARS算法筛选的敏感波长, 主要集中在430~550、 560~900、 980~1 280、 1 351~1 520和1 621~1 680 nm炮, 波段的整体重要性在0.5附近; 从图7中能够看到, 基于SPA算法筛选的敏感波长主要集中在400~520、 1 000~1 280、 1 400~1 500和1 600~1 680 nm内, 敏感波段的重要性在0.4~0.5之间, SPA与CARS算法筛选后的敏感波段和已有文献报道的相关结果一致[14]。 相比于SPA算法, 基于CARS筛选的特征波长的整体重要性更高且CARS筛选波长分布更加分散, SPA筛选的波长则更多集中在400~550、 1 000~1 300和1 400~1 700 nm区间, 忽略了600~900 nm之间的光谱特征, 这可能会对模型的精度造成一定的影响。
基于全波段数据以及SPA、 CARS两种敏感波长筛选后的特征数据, 对比本研究提出的1D-CNN1, 1D-CNN2与在SOM预测方面表现效果较好的VGG16[14]以及机器学习中的PLSR、 支持向量回归(support vector regression, SVR)、 随机森林(random forests, RF)六种方法建立的模型, 以回归模型中的决定系数(coefficient of determination, R2)、 均方根误差(root mean square error, RMSE)、 相对分析误差(residual predictive deviation, RPD)三个指标综合评价模型表现。 最终建模与预测结果如表3所示。
| 表3 不同模型训练结果对比 Table 3 Comparison of training results of different models |
首先, 对比不同筛选算法的效果来看, 以SPA筛选的敏感波段建立的模型, 波段数量被压缩到全波段的6%, 在预测集上, 1D-CNN1与1D-CNN2、 VGG16和SVR模型能够实现SOM含量的有效预测(R2大于0.8, RPD大于2), 1D-CNN1模型表现最好, R2=0.812, RMSE=3.318 g· kg-1。 对比全波段建模, SVR模型在R2和RPD上提升了0.043和0.012, 其余模型的预测精度相比全波段建模均有所下降; 相较于全波段数据, 1D-CNN1, 1D-CNN2, VGG16, RF, PLSR的R2下降了0.006, 0.007, 0.006, 0.002和0.040, RPD下降了0.013, 0.066, 0.054, 0.041和0.178。 相比之下, 以CARS筛选敏感波段建立的模型中, 除了RF模型表现较差, 其余模型均能实现SOM含量的有效预测。 对比全波段建模, 除了PLSR与RF在预测集上R2下降了0.024, 0.004, RPD下降了0.121, 0.025; 1D-CNN1, 1D-CNN2, VGG16和SVR的R2分别提升了0.028, 0.018, 0.005, 0.026, RPD提升了0.039, 0.034, 0.012, 0.077。
SPA与CARS两种敏感波长筛选算法分别将全谱数据降至6%和8%, 两种敏感波长筛选算法都能够实现光谱数据的有效降维, 剔除不相关波长的干扰, 保留敏感波长。 从模型表现来看, CARS筛选敏感波段整体表现优于全波段数据和SPA敏感波段, SPA虽然整体表现不如全波段数据, 但是模型表现差距很小, 降维后波段数仅为全波段数据的6%, 这有助于提升模型的运行效率, 为边缘计算的研究奠定基础。
在模型方面, 对比不同模型基于敏感波段和全谱数据的表现, CNN模型整体表现相比传统模型更好, 在基于CARS和SPA以及全波段数据上, 都能做到精准预测。 对比18种模型, 基于CARS的1D-CNN1表现最佳, R2=0.846, RMSE=3.145 g· kg-1。
提出的具有6层卷积层的浅层CNN模型, 相较于VGG16表现更好, 有效减少了网络层数, 同时提升了运行效率和预测能力。 对比模型不同参数的表现, 具有更小卷尺寸的1D-CNN1表现优于具有更多数量的大尺寸卷积的1D-CNN2, 证明小尺寸卷积对于模型性能的提升更为明显。 相比之下, 机器学习整体表现明显低于CNN网络, 不同数据的表现具有明显差异, 主要是因为光谱数据与土壤属性之间存在更多非线性因素, 将会直接影响模型的预测精度, 同时模型整体泛化性较差, 容易导致不同数据的表现呈现出明显差异。
对高光谱数据降维并建立具有更好的SOM预测效果的模型是本研究的重点。 基于采集的320份耕层土壤, 提取350~1 700 nm区间的SOM高光谱数据, 通过MSC和一阶微分对光谱反射率进行变换, 结合1D-CNN1、 1D-CNN2、 VGG16和PLSR、 SVR、 RF六种模型分别建立基于全波段数据和SPA、 CARS敏感波段数据的SOM预测模型, 结论如下:
(1)相比于PLSR、 SVM、 RF等传统经典模型, 基于CNN的预测模型精度更高、 模型泛化性更强, 本研究提出的浅层CNN表现优于VGG16, 有效提升了预测精度, 降低了模型负载。
(2)基于CARS敏感波长的SOM预测模型整体表现优于SPA和全波段数据, 筛选过后的敏感波段数量仅为全波段数量的8%, 证明了基于敏感波长算法, 不仅能有效降低高光谱数据的维度, 也提高了模型的预测能力。
(3)基于CARS的1D-CNN1在所有模型中表现最好, 优于1D-CNN2, 证明3× 3卷积相比于5× 5, 7× 7等大尺寸卷积效果更佳, 预测R2 =0.846, RMSE=3.145 g· kg-1, 为快速检测土壤SOM提供了新思路。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|