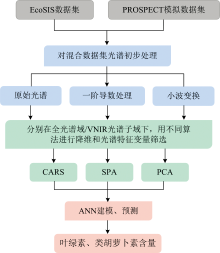
作者简介: 汤馥睿, 1999年生, 江苏大学物理与电子工程学院硕士研究生 e-mail: hwrem12345@163.com
叶绿素和类胡萝卜素含量是评价植物健康状况的一个重要指标。 PROSPECT模型与机器学习耦合反演植被生化特性已得到广泛应用。 但由于叶片方向半球反射率因子(DHRF)光谱和二向反射率因子(BRF)光谱之间的差异, 耦合模型的应用范围受到限制。 为此, 以北美地区植物叶片光谱数据库(EcoSIS)作为实验数据集, 提出PROSPECT模型作为机器学习的附加约束形成混合数据集, 对此混合数据集利用连续小波变换(CWT)产生的小波系数谱和一阶导(FD)产生谱, 提出三种全光谱域和VNIR光谱子域下的植物叶片叶绿素、 类胡萝卜素的光谱特征变量筛选策略, 即是: 竞争性自适应重加权算法(CARS)、 连续投影算法(SPA)和主成分分析法(PCA)。 由此, 基于上述2×2×3=12种不同光谱处理方法、 特征提取方法组合, 分别建立了植物叶片叶绿素和类胡萝卜素含量的人工神经网络(ANN)预测模型。 进而开展了不同模型下的预测精度对比分析, 结果表明: PROSPECT模型约束下的模拟数据一定程度增强了机器学习的训练集质量; 经一阶导、 小波变换处理的光谱能较好地减少DHRF模拟光谱和BRF实测光谱间的偏差, 并在结合特征提取算法CARS后进一步提升了预测表现。 在全光谱域下的FD+CARS组合对叶片叶绿素的反演效果最佳, 测试集 R2为0.806 4, RMSE为2.911 4; 在VNIR光谱子域下的CWT+CARS组合对叶片类胡萝卜素最佳, 测试集 R2为0.797 2, RMSE为0.414 1。 该方法可为研究人员从叶片BRF光谱及其他近端反射率图像更精确、 高效地提取植物叶片生化特征提供参考。
The chlorophyll and carotenoid content is an important indicator for evaluating the health status of plants. The PROSPECT model, coupled with machine learning, has been widely used to retrieve the biochemical properties of vegetation. However, the application of the coupled model is limited due to the differences between the leaf-directional hemispherical reflectance factor (DHRF) spectra and the bidirectional reflectance factor (BRF) spectra. This paper utilizes the leaf spectral database of North American plant (EcoSIS) as the experimental dataset and introduces the PROSPECT model as an additional constraint for machine learning. This approach creates a hybrid dataset by employing wavelet continuous wavelet transform (CWT) to generate the wavelet coefficient spectrum and the derivative spectrum generated by the first-order derivative (FD). Three kinds of feature extraction algorithms, namely competitive adaptive reweighting algorithm (CARS), successive projection algorithm (SPA), and principal component analysis (PCA) were applied to extract spectral features for chlorophylls and carotenoids in the full-spectral domains and the subdomain of VNIR spectroscopy. Based on the above 12 combinations of different methods, artificial neural network (ANN) prediction models for chlorophyll and carotenoids were separately established. The results show that the simulated data under the constraint of the PROSPECT model enhanced the quality of the training set for machine learning to a certain extent. Additionally, the spectra processed by the first-order derivatives and wavelet transforms were able to reduce better the bias between the simulated spectra of the DHRF and the measured spectra of the BRF. The best inversion of leaf chlorophyll is achieved with the FD+CARS combination in the whole spectral domain, yielding a test set R2 of 0.806 4 and RMSE of 2.911 4. Meanwhile, the CWT+CARS combination in the VNIR spectral sub-domain offers the best results for leaf carotenoids, with a test set R2 of 0.797 2 and RMSE of 0.414 1. The proposed method can provide researchers with a reference to extract biochemical characteristics of plant leaves more accurately and efficiently from BRF spectra and other near-end reflectance images.
植被是地球生态系统最重要的组成部分之一, 同时, 植被的生长状态, 包括健康应激状态和功能过程是整个生态系统的一个重要评价因素[1]。 叶绿素和类胡萝卜素含量是光合作用活性的重要指标, 从中可为了解植物光合功能、 动态生长、 营养循环和初级生产提供有价值的信息[2]。 因此, 准确、 快速地反演出它们的含量是研究植被的生长状态与环境胁迫之间关系的一种通用而有效的方法。
植被参数反演方法大致可以划分为三类。 第一类是纯数据驱动式的经验学习方法, 如支持向量机(support vector machine, SVM)、 偏最小二乘法(partial least squares, PLS)、 反向传播神经网络(back propagation neural network, BP-NN)和径向基函数神经网络(radial basis function neural network, RBF-NN)等回归算法[3]。 经验学习方法有较强的数据适应性, 但其忽视物理规律, 因果可解释性差。 第二类是机理模型驱动, 由辐射传输模型(radiative transfer model, RTM)发展形成, 如雷祥祥等[4]基于PROSPECT模型对实测植物叶片反射率光谱进行拟合, 无损、 定量地获取植物叶片的叶绿素含量。 理论模型基于知识及演绎, 能够表征叶片光学特性和其生物化学参数之间的机理关系, 结果可解释性强[5], 不过, 物理建模方法难以精准地描述复杂多变的现实情况, 并且模型所需输入参量较多, 易导致病态反演问题。 第三类为数据与模型的混合驱动, 该方法通常基于机理模型产生模拟数据集, 再利用机器学习对该数据集的植被光谱和相应生化参数进行训练, 最后将训练好的模型迁移应用于实际场景中的参数反演。 如Berger[6]通过耦合PROSPECT-PRO模型与SAIL冠层反射率模型生成训练数据库, 再结合高斯过程(gaussian processes, GP)提取农作物氮含量的最佳光谱波段, 实现作物参数的混合反演。 数据与模型的混合驱动的方法进一步提升定量遥感反演的精度, 已成为植被参数反演研究领域的一个热点[7]。 然而, 在叶片尺度下的植物叶绿素、 类胡萝卜素反演方面, 以真实数据集为基础, 是否能将模拟数据作为物理模型辅助下的机器学习附加数据来源, 既增强数据质量, 又可使PROSPECT模型的物理性先验知识融入训练过程, 以对机器学习起到一定的约束作用, 还有待进一步深入探讨和验证。
除此之外, 尽管叶片光学特性模型PROSPECT及其扩展版本能够表征光与叶片之间相互作用的机理过程, 具有良好的泛化能力, 但是, 模型的输入量为叶片方向半球反射光谱(directional-hemispherical reflectance factor, DHRF), 难以满足现代农业对大田作物施行高效监测的实际需求。 相比之下, 双向反射光谱(bidirectional reflectance factor, BRF)可通过叶片夹或高光谱成像系统来获得, 测量效率更高。 然而, 叶片并非完美的朗伯体, 其表面反射率的存在以及反射率的各向异性导致了叶片DHRF和BRF光谱之间存在差异, 也限制了PROSPECT模型在叶片BRF光谱的应用。 因此, 为提高PROSPECT模型反演精度, 减少表面反射效应的影响是一项值得研究的工作。 为了解决该问题, Dong等[8]开发了基于双差和红边位置的光谱指数, 该指数对镜面反射率的变化不敏感, 可适用于DHRF光谱和BRF光谱反演; Joe等[9]引入光入射角、 照明天顶角和镜面反射参数, 开发了叶片近距离光谱成像模型PROCOSINE, 可以基于PROSPECT模型模拟叶片BRF光谱。 然而, 由于没有考虑水吸收对叶片折射率的影响, 模型精度仍有改进的空间。 Li[10]则将PROSPECT和小波变换结合为PROCWT模型, 抑制了水稻和小麦的表面反射率效应, 相较于PROSPECT模型可有效提高作物生化参数的反演精度, 但其基于叶片BRF和DHRF光谱之间与波长无关的差异假设, 波长依赖的情况尚未得到探索。 Wan[11]考虑了波长影响的情况, 以曼哈顿距离、 欧氏距离为评价函数, 将PROSPECT模型与导数相结合, 发现在不同数据集中, PROSDM模型相较于其他模型均有较大的预测效果提升。 但是该方法采用单一类型光谱反演, 对于BRF光谱与DHRF光谱混合后, 导数和小波变换处理对消除两者差异的有效性仍有待研究; 另一方面, 由于未考虑特征变量维度的影响, 输入模型的光谱变量仍有2 000多个, 较多的变量会加重机器学习负载, 影响模型预测效果。
为提高植物叶绿素、 类胡萝卜素的反演精度, 提高机器学习训练数据的质量, 提出以PROSPECT模型模拟数据实现对真实数据集的约束, 与EcoSIS(ecological spectral information system)数据集形成混合数据集, 对该数据集光谱经过一阶导和连续小波变换的数值处理, 以消除部分BRF和DHRF光谱差异, 组合以不同的光谱特征变量提取算法, 从而提升叶绿素、 类胡萝卜素的反演精度, 同时、 降低模型负载。 本方法通过加拿大植物物种叶片光谱实验数据库的应用验证, 获得了较为精准的叶绿素和类胡萝卜素的反演结果。
本研究使用两个数据集: EcoSIS实验数据集和PROSPECT模拟数据集。
1.1.1 EcoSIS数据集
EcoSIS数据集是由Kothari等[12]、 Wang[13]等于2017年— 2019年间在美国、 加拿大等各野外地区采用PSR-3500型全量程光谱仪测量得到。 其中包含近2 000个植被叶片样本, 灌木、 乔木等100种植物, 包括叶片的反射光谱以及各种生物化学参数, 如叶绿素a/b含量、 类胡萝卜素含量等, 满足光谱的多样性需求。 数据集可以在线获得(https://ecosts.org), 所采用标准化协议参考Asner[14]的研究文献。
1.1.2 PROSPECT模拟数据集
采用改进后的PROSPECT-D模型生成模拟数据集, 该数据集用于与EcoSIS数据集融合构成机器学习建模所需的混合训练集, 达到增强机器学习数据训练的效果。 其中, 叶绿素和类胡萝卜素模拟参数的含量范围由EcoSIS数据集限制, 其他参数参考以往文献设定(见表1)。
| 表1 PROSPECT-D 模型参数设置 Table1 Parameter configurations of PROSPECT-D model |
1.1.3 混合数据集的构建
为了有效构建混合训练数据集, 使用K-S检验(Kolmogorov-Smirnov, K-S)算法, 按3∶ 1的比例将EcoSIS数据集划分为完全独立的A、 B两部分, A部分参与构建建模集, B部分作为模型的验证集。 每次随机挑选PROSPECT模拟数据, 与EcoSIS数据集A部分相混合, 按1∶ 3的混合比例构成机器学习的训练集。 从表2可见, A、 B两部分的最值、 平均值和标准差皆相近, 可以较为均匀地覆盖训练集样本和测试集样本。
| 表2 样本集色素计量数据统计(μ g· cm-2) Table 2 Statistics of pigmentation in the sample set (μ g· cm-2) |
根据作用的不同, 光谱预处理可以分为基线校正、 散射校正、 平滑处理和尺度缩放四类。 其中, 基线校正包括一阶导数(first-order derivative, FD)、 二阶导数(second-order derivative, SD)、 连续小波变换(continuous wavelet transform, CWT)等, 主要应用于消除背景基线漂移对信号的影响。
混合训练集中, DHRF光谱与BRF光谱反射率之间的偏差f会影响最终的反演精度。 偏差f随光谱区域(如可见光和近红外区域)和植物种类而变化, 假设该f值线性可加[11, 15], 同时可以把f看作是一种“ 系统背景” 或者“ 系统漂移信号” , 利用导数和小波变换来消除部分f值对BRF的影响。 即, 用小波系数谱和一阶导数谱代替原反射光谱, 作为混合模型反演时的输入值, 以降低表面反射效应对模型反演效果的干扰。
连续小波变换使用缩放和移位的母小波对反射光谱进行卷积。 高斯函数的二阶导数与植物叶片吸收特征光谱的形状相似[10, 16], 而第四尺度的缩放又在叶片色素反演上有更好的表现, 因此选择高斯函数的二阶导数为母小波函数, 并在第四尺度缩放。 CWT 调用Python3.7版本的pywave库对光谱进行处理; 一阶导数直接在Python软件实现。
BRF与DHRF的关系如式(1)和式(2)描述。
$\text{BR}{{\text{F}}_{\left( {{\theta }_{S}}, \ \ {{\varphi }_{S}}\ ; \ \ {{\theta }_{V}}, \ \ {{\varphi }_{V}}\ ; \ \ \lambda \right)}}=\pi \times \text{BRD}{{\text{F}}_{\left( {{\theta }_{S}}, \ \ {{\varphi }_{S}}\ ; \ \ {{\theta }_{V}}, \ \ {{\varphi }_{V}}\ ; \ \ \lambda \right)}}=\pi \times \frac{\ \ \ \ {{R}_{\left( {{\theta }_{S}}\ , \ {{\varphi }_{S}}\ ; \ {{\theta }_{V}}\ , \ {{\varphi }_{V}}\ ; \ \lambda \right)}}\ \ \ \ \ }{{{I}_{\left( {{\theta }_{S}}\ , \ {{\varphi }_{S}}\ ; \ \lambda \right)}}\ \ \ }\ \ \ \ \ $(1)
$\text{DHR}{{\text{F}}_{\left( {{\theta }_{S}}, {{\varphi }_{S}}; \ \lambda \right)}}=\int\limits_{0}^{\pi }{\underset{0}{\overset{\frac{ \pi }{2}}{\mathop \int }}\, }\text{BRD}{{\text{F}}_{\left(\ {{\theta }_{S}}, \ {{\varphi }_{S}}\ ; \ {{\theta }_{V}}, {{\varphi }_{V}}\ ; \ \lambda \ \right)}}\ \ \text{cos}{{\theta }_{V}}\ \text{sin}{{\theta }_{V}}\ \text{d}{{\theta }_{V}}\ \text{d}{{\varphi }_{V}}\ \ \ $(2)
Jay[9]将二者关系简化为式(3)
其中
反射率由表面反射分量(surface)和漫反射分量(diffuse)构成, 前者描述了叶子的表面反射, 后者描述了叶片内部的吸收和散射过程。 式(4)反映了BRFsurf和DHRFsurf两个表面反射分量之间的差异, 也是DHRF光谱和BRF光谱之间的差异。
考虑到大部分波长与光谱特征相关, 且相邻波段间存在共线性问题, 这些会影响叶片的色素含量分析[2]。 为此, 本文采用竞争性自适应重加权算法(competitive adaptive reweighted sampling, CARS)、 连续投影算法(successive projections algorithm, SPA)、 主成分分析(principle component analysis, PCA)三种算法[17, 18], 对原始光谱和分别经过一阶导处理、 小波变换处理的光谱, 进一步筛选出与叶绿素、 类胡萝卜素最具有相关性的特征变量, 再导入机器学习模型中。
已有研究结果表明在光谱子域检索叶片生化性状, 可能获得更好的反演结果[19, 20], DHRFsuff和BRFsuff之间的偏差f在不同的光谱区域有所差异。 通过对不同特征变量提取方法组合的结果分析(见表3), 综合考虑全光谱域(400~2 400 nm)和可见-近红外光谱子域(visible and near infra-red, VNIR)反演结果可能带来的不同影响, 依据前述FD、 CWT 2种预处理方法, 以及CARS、 SPA、 PCA 3种算法降维后的特征光谱变量, 设计12种组合, 分别对叶绿素和类胡萝卜素含量开展反演。 单独使用原始光谱和经一阶导、 小波变换处理的光谱所构模型作为对照组。 各组合的变量筛选详见后文。
| 表3 不同特征变量提取方法组合 Table 3 Combination of different feature variable extraction methods |
PROSPECT模型约束下的机器学习反演植物叶片胡萝卜素、 叶绿素流程见图1, 其中由人工神经网络模型(artificial neural network, ANN)在混合数据集中分别构建叶绿素和类胡萝卜素含量监测模型, 对比经过单一算法和组合算法处理过的模型性能。 采用测试集决定系数(coefficient of determination of prediction,
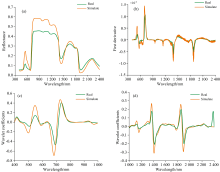
在EcoSIS数据集和仿真数据集中各自选取第一个样本, 其原始光谱如图2(a)所示; 分别进行一阶导和小波变换处理后的图像如图2(b), 图2(c, d)所示。
图2(a)可见, 模拟光谱DHRF与实测光谱BRF之间存在反射率的差异, 难以在振幅上将二者进行直接比较。 并且在反射率空间中, 只能观察到叶绿素在450和680 nm处和水分在1 450和1 950 nm 处较强的吸收特征。 由于叶片色素在可见蓝光、 红光区域的强吸收, 而在绿波段强反射, 因此会在420和680 nm附近出现两个吸收谷, 在约600 nm处出现一个反射峰。 对比图2(a)和(b)可见, 经一阶导处理后的光谱更显著地突出了原始光谱的两“ 谷” 一“ 峰” , 从中可见: 以拐点形式所捕捉到的叶片黄边(560~580 nm)陡然降低、 红边(680~750 nm)陡然增加, 光谱导数在增强局部光谱特征和消除微小的光谱变化方面具有一定优势, 小波变换同样可以较好地捕捉到这些差异。 如图2(c, d)所示, 第四尺度的小波系数谱展示了更为具体的窄带吸收特征。 以对植被光谱特征具有重要指示意义的两个波段, 即可见光黄波段(560~580 nm)和红波段(680~750 mm)为例, 相较于原始光谱, 小波系数谱更易于观察到关于叶片光谱黄边、 红边特征的变化细节, 也意味着可能包含了更丰富的叶片叶绿素、 类胡萝卜素的光谱信息。 这两种处理方法或能有助于减少混合训练集中实测BRF光谱和模拟DHRF光谱之间的差异, 并增强叶片色素的吸收特征, 改善植物叶片叶绿素、 类胡萝卜素的提取效果。
2.2.1 基于CARS的特征光谱变量提取
对样本的一阶导数谱和小波变换系数谱采用 CARS算法提取特征光谱变量, 设置蒙特卡罗(Monte-Carlo, MC)采样50次, 采用5折交叉验证法, 得到一阶导数谱在全光谱域筛选叶绿素含量特征波长的运行结果(见图3)。
 | 图3 一阶导数谱筛选叶绿素特征波长CARS运行结果Fig.3 Screening chlorophyll characteristic wavelengths of FD spectra by CARS |
图3(a)、 (b)和(c)分别表示在CARS算法运行过程中, 特征变量数、 五折交叉验证均方根误差(root mean square error of cross validation, RMSECV)和各特征变量回归系数三者随着采样次数的增加而发生的变化。 从图3(a)可以看出, 变量数经历了先“ 粗选” 后“ 精选” 2个选择过程, 在前5次采样中快速下降, 随后缓慢减少并趋于平稳。 图3(b)展示了RMSECV先逐渐减小后陡然增加的趋势, 在其达到最小值时, 采样运行14次, 各特征光谱变量的回归系数由图3(c)中的“ * ” 号垂线标示。 此后若采样次数继续增加, 光谱中某些包含叶绿素关键信息的重要变量将被剔除, RMSECV也随之增加。 因此, 选择第14次采样提取的52个变量为叶绿素含量的特征光谱变量, 现有维度相比原始维度下降了97.4%。
如图3(d)所示, 特征波段大多数分布于400~800 nm之间, 包含了可见蓝光、 绿光区域, 也是对植物光合作用活性最为敏感的区域。 叶片的“ 黄边” 特征是指在560~580 nm间的窄带, 由于其叶绿素、 类胡萝卜素从绿波段强反射过渡到黄波段强吸收, 光谱反射率曲线陡然降低的特点。 “ 红边” 特征则是指叶片光谱反射率在680~750 nm之间陡然攀升的特点, 也表征了叶片光学特性的主要决定因素从叶绿素对红波段的强吸收, 转变为叶片内部细胞结构对近红外波段的强反射。 CARS算法在黄边位置(560~580 nm区间反射率降低最快的点), 即一阶导数谱的第一个拐点附近筛选出了若干特征波长, 红边位置(680~750 nm区间反射率上升最快的点)即第二个拐点右侧也有少量波长被采集。 使用该算法提取的特征变量基本具备叶绿素的关键光谱信息。
2.2.2 基于SPA的特征光谱变量提取
对样本的一阶导数和小波变换谱采用SPA算法提取特征光谱变量, 计算在不同个数子集的特征光谱变量下的均方根误差RMSE, 选取该值最小者为最优特征变量子集。 以小波系数数谱在VNIR子域的类胡萝卜素敏感波长提取为例, 设置5~50为特征变量数的变化范围, 当提取特征变量个数为15时, RMSE取得最小值3.927。 该15个特征光谱变量占原始波段数的2.5%, 在VNIR光谱子域的分布如图4所示。
 | 图4 基于SPA的特征变量提取 (a): RMSE迭代变化图; (b): SPA特征变量分布图Fig.4 Extracting characteristic variables by SPA (a): RMSE iterative change chart; (b): Distribution of characteristic spectral variables |
2.2.3 基于PCA的主成分提取
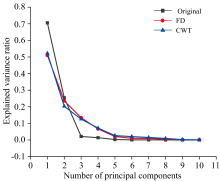
对样本的原始高光谱、 一阶导数和小波变换光谱数据进行主成分分析, 前10个主成分(principal components, PC) 的方差贡献率如图5所示。 以原始光谱的PCA为例, 第1个PC贡献率为72.78%, 是所有PC中贡献率最大的; 前2个PC累积贡献率为86.68%, 之后各PC累积贡献率缓慢增加; 前7个PC的累积贡献率为99.89%, 仅剩0.11%的光谱信息未能表达。 以方差累积贡献率≥ 95%以上为阈值, 原始光谱、 一阶导光谱和小波系数谱均选择最佳PC个数为7, 并以该7个主成分作为特征变量进行建模, 为原始高光谱维度的0.3%。
2.3.1 ANN模型的叶绿素含量预测结果
在人工神经网络ANN模型中, 选取“ Relu” 函数作为神经网络的激活函数, “ Adam” 为权重优化器, 并设置ANN的隐含层神经元个数为50(4层), 最大迭代次数2 000。 用原始光谱及6种光谱处理方法提取的特征变量建立对应的ANN模型, 得到叶绿素含量预测结果见表4。 为避免自变量数值差异过大影响建模精度, 建模前将所有自变量作归一化处理。
| 表4 不同处理方法建立的ANN模型叶绿素含量预测结果 Table 4 Prediction results of chlorophyll content of ANN models established by different treatments |
表4结果表明: 对于叶绿素反演, 无论全光谱域还是VNIR子域, 经过一阶导和小波变换处理均表现出了相似的性能, 相对于原始光谱有更高的准确率。 在全光谱域下, 测试集决定系数
2.3.2 ANN模型的类胡萝卜素含量预测结果
不同处理方法ANN模型的类胡萝卜素含量预测结果如表5。
| 表5 不同处理方法建立的ANN模型类胡萝卜素含量预测结果 Table 5 Prediction results of carotenoid content of ANN models established by different treatments |
从表4、 表5的分析可以发现, 大部分光谱处理组合的模型对两种色素的预测表现都高于原始光谱, 叶绿素的模型预测表现整体高于类胡萝卜素。 总体上看, 叶片的黄边特征、 红边特征不受水分和类胡萝卜素的影响, 但其红边位置, 即680~750 nm区间反射率攀升最快点会随叶绿素含量的增加而红移, 并且在400~800 nm的可见光区域, 叶片的光谱特征由叶绿素的吸收主导, 因此叶绿素的模型预测精度较高。 而类胡萝卜素的反演因其吸收特征会被可见光区域中的叶绿素和短波红外(short wavelength infra-red, SWIR)区域中的水分含量所掩盖[16], 所以类胡萝卜素反演精度低于叶绿素的反演精度。 两种色素的最佳预测结果见散点图6。
2.3.3 特征提取对反演结果的影响
不同组合的特征波长提取结果见图7。 经过降维处理的模型并没有更优异的预测表现。 例如表5的FD+CARS、 CWT+CARS组合, 有效剔除了全光谱域中与叶绿素、 类胡萝卜素相关性小的波段, 提高了信噪比, 在保持准确率基本不变的基础上减少了建模所需要的波长, 但模型很难有比全谱更突出的预测性能。 FD+SPA在VNIR子域所提取波长能覆盖叶绿素、 类胡萝卜素的部分敏感波段, 却使得蓝光波段出现“ 波长聚集” 现象; CWT+SPA在全光谱域提取的特征波长分布较均匀, 然而弱相关波长和噪声较多, 预测效果差。 使用PCA能够较好地解决多重共线性的问题, 各模型性能相对稳定, 但准确率不算最优。 从结果来看, 光谱特征筛选的主要作用是降维并减少模型负载, 降低模型训练所耗时间。
 | 图7 不同组合所提取特征波长位置 (a): VNIR子域下的特征波长位置; (b): 全光谱域下的特征波长位置Fig.7 Characteristic wavelengths extracted with different combinations (a): Within the VNIR subdomain; (b): Within the full spectral domain |
2.3.4 不同光谱域对反演结果的影响
在VNIR子域, 原始光谱和CWT处理过的光谱在反演叶绿素方面有所改善, 拟合度和稳定性得到提高, 且较好地提高了SPA算法组合的
2.3.5 FD与CWT对消除混合光谱中的f的有效性和适用性
Wan等[11]研究结果表明: f对BRF谱的贡献可达20%~100%, 视不同谱域而定; 不同物种叶片的形状、 叶片表面的粗糙程度也会对f造成影响, 例如针叶植物比阔叶植物有更复杂的多向反射[21]; 光滑表面会使叶片产生更高的表面反射率, 影响反演精度。 在本研究中, 通过消除表面反射率改善叶绿素、 类胡萝卜素的反演结果, 可有效减少f对模型反演的影响。 然而, 本方法不能直接用于光谱模拟, 而是通过缩小DHRF和BRF之间的差异来提高叶片生化参数的反演精度。 除此之外, 叶片表面反射率影响着叶子在不同观测角度的定向反射率的大小和分布。 导数和小波变换处理直接将光谱中的表面反射率简化为混合光谱间的差异f[见式(4)], 而未考虑BRF光谱的多角度问题, 对叶片表面特性的表征不充分, 尚不能完全消除f对BRF的影响[22]。 如何进一步提高反演精度, 还有待进一步深入研究。
以多个物种的植物叶片叶绿素、 类胡萝卜素为研究对象, 提出了EcoSIS数据库和PROSPECT合成数据构建混合数据集的方法, 利用导数处理和小波变换处理、 不同特征光谱变量提取方法组合构建不同的模型, 建立了叶片叶绿素和类胡萝卜素精准反演的方法, 其研究结果表明:
(1)用一阶导、 小波变换处理的光谱在叶片叶绿素、 类胡萝卜素的反演中, 相对于原始光谱有更好的表现, 对于叶绿素反演, 测试集决定系数
(2)对比12种组合方法, FD+CARS、 CWT+CARS组合对两种叶片色素有较好的反演效果, 叶绿素含量的FD+CARS+全光谱域组合测试集决定系数
(3)叶绿素的反演精度总体优于类胡萝卜素, VNIR子域不同算法组合所构建模型对类胡萝卜素反演的效果提升大于叶绿素。
(4)综上所述, 一阶导与小波变换可以改善PROSPECT模型约束下的混合训练集中的光谱差异问题, 同时, 光谱特征变量提取算法对机器学习建模有一定的降低训练负载作用。 该方法从叶片BRF光谱的角度出发, 为基于PROSPECT模型提取植被生化特征的研究提供了一种新参考。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|