作者简介: 马焕臻,女, 1997年生,国科大杭州高等研究院硕士研究生 e-mail: mahuanzhen20@mails.ucas.ac.cn
血液是一种受管制的特殊遗传生物资源。 针对传统血液光谱检测中易氧化变质的问题, 采用基于仿生血管的动态共聚焦拉曼荧光光谱, 开展了猪、 马、 鸽、 鸡、 鸭、 鹅等六种家禽家畜的血液物种鉴别研究。 原始光谱的预处理过程包括去基线、 平滑和归一化等。 采用线性判别分析对光谱数据进行降维处理, 继而用支持向量机建立识别模型, 选用高斯核函数, 通过人工鱼群算法优化支持向量机的参数 C和 γ, 使其分类准确率最高, 最优的 C和 γ分别为0.2和0.134。 人工鱼群-支持向量机模型识别准确率达到97.2%, 基于仿生血管的动态共聚焦拉曼荧光光谱可以满足血液安全高效的检测要求, 用人工鱼群算法优化支持向量机参数的算法模型表现出较好的分类效果。
Blood is a regulated exceptional genetic biological resource. In response to the issue of easy oxidation and deterioration in traditional blood spectral detection, dynamic confocal Raman fluorescence spectroscopy technology based on biomimetic blood vessels was used to conduct blood species identification research on six types of poultry and livestock, including pigs, horses, pigeons, chickens, ducks, and geese. The preprocessing process of the original spectrum includes baseline removal, smoothing, and normalization. Linear discriminant analysis is used to reduce the dimensionality of spectral data, and then support vector machines are used to establish recognition models. Gaussian kernel functions are selected, and the parameters C and γ Make their classification accuracy the highest, the optimal C and γ 0.2 and 0.134, respectively. The recognition accuracy of the artificial fish school support vector machine model reaches 97.2%. The dynamic confocal Raman fluorescence spectrum based on biomimetic blood vessels used in this article can meet the requirements of blood safety and efficiency detection, and the algorithm model optimized by the artificial fish school algorithm for support vector machine parameters shows good classification performance.
近年来, 一些不法分子通过非法手段运输血液, 血液种属识别对海关出入境检验意义重大。 国内外使用光谱技术进行血液分类相关的研究很多。 Balzerova Anna[1]采用在玻片上涂抹的方法用表面增强拉曼检测血液中的免疫球蛋白, Gurian等[2]同样采用涂片方法, 用表面增强拉曼观测肝癌患者血液和普通人血液, 采用化学计量学的方法可以将健康细胞和淋巴瘤细胞区分开, Lima等[3]利用衰减全反射傅里叶变换红外光谱技术, 使用GA-LDA模型鉴别血浆和血清样本中生物标志物, 可诊断卵巢癌的不同阶段。 国内将支持向量机用于拉曼光谱血液鉴别人、 犬和兔血, 采集了鸽、 鸡、 鼠、 羊四种动物不同浓度的全血与红细胞荧光光谱数据, 建立BP神经网络分类器对其进行分类, Xue等[4]使用激光诱导光谱击穿技术(LIBS)对全血样本进行淋巴瘤的鉴别。
以上关于血液的研究, 基本采用涂片法, 对于来源不明的血液, 涂片法可能会有传播疾病的风险, 而且激光长时间凝视同一区域, 热效应会导致该区域血液变质, 检测和分类精度大打折扣。 Wang等[5]提出动态仿生血管的方法很好地解决了传播疾病风险大和血液变质的问题, 微流泵通过封闭的仿生血管使血细胞通过凝视区的焦点区域, 仿生血管材质为聚氯乙烯(PVC), 直径3 mm, 管壁厚度0.5 mm, 选用人血和牛血作为实验对象, 采用PCA-LDA算法模型, 实现了准确的识别分类, 并利用导数光谱比较了微弱的拉曼信号差异, 可以有效区分人和非人的血液样本, 但尚未对多物种血液进行鉴别分类。 最近Wang等采用支持向量机进一步对四种家禽的血液进行鉴别[6], 取得良好的效果, 本研究在原来基础上进行了进一步推进, 在分类中加入两种家畜(猪和马), 采用实验室自主研发的生物特征光谱识别仪, 通过仿生血管心脏泵, 使六种家禽家畜的全血样品流动起来, 对其进行动态的拉曼和荧光光谱测量, 有效地避免了血液样品的变质和传播疾病的风险, 且采用LDA对光谱数据进行降维, 用人工鱼群算法找到最优的SVM两个系数, 参数惩罚因子C和γ, 对测试集的分类准确率可达到97.2%。
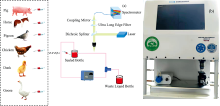
采用实验室自主研发的仪器, 激光器中心波长为532 nm(连续工作模式, 横向工作模式: TEM00, 激光功率为30 mW, 谱线宽度小于0.003 nm)血液样品通过注射器注入密封瓶, 通过仿生心脏在仿生管里流动, 仿生血管直径3 mm, 壁厚0.5 mm, 血液在管中的流速为0.42 mL·(s·mm2)-1, 测试完成后回收到废液瓶统一处理。 实验用到的血液样本由拱北海关提供, 每条光谱的曝光时间为5 s, 采用Nova拉曼光谱仪, 光谱记录范围535~635 nm, 分辨率为0.3 nm。 向后的拉曼信号依次通过40倍奥林巴斯显微镜物镜, 二向色分光镜(DS), 和超长边滤光镜(ULEF), 最后由耦合镜收集到光谱仪中进行记录。 光路示意图和仪器实物如图1(a)和图1(b)所示。
实验前采集的猪、 马、 鸽、 鸡、 鸭、 鹅六种动物的新鲜血液, 每种血液取自30个供体动物, 供体动物雌雄均有, 用PET(聚对苯二甲酸乙二醇酯)塑料试管盛放新鲜血液, 使用EDTA(乙二胺四乙酸)作为抗凝剂, 先将新鲜血液放入-25 ℃冰箱中冷冻处理24 h, 然后对冷冻血液进行解冻处理, 每种解冻的动物血液取1 mL, 将其与10 mL生理盐水按照1∶10的比例混合稀释, 稀释后再次放入-25 ℃冰箱冷冻处理, 预处理后的血液样本可以多次重复使用。 将处理后的样品放入密封瓶, 利用仿生心脏泵使样品流动起来, 对激光聚焦区域进行拉曼光谱检测, 每次采集下一物种血液光谱之前, 仿生血管内通入纯净水, 直至废液瓶中液体完全无色, 防止不同样品之间互相污染。 选取6个物种共536条光谱数据(其中猪94条, 马115条, 鸽89条, 鸡85条, 鸭75条, 鹅78条)。 实验结束后, 通过废液瓶收集血液。
采集得到的光谱信息不仅包括与属性有关的信息, 还有背景噪声等干扰, 所以在对血样进行分类前, 需要对光谱进行预处理。
截取拉曼频移为500~1 800 cm-1, 使用BEADS算法[7](Baseline Estimation and Denoising with Sparsity)对原始的536条光谱数据进行去基线, 去除荧光背景和噪声, 使用Python自带的numpy库的convolve函数对光谱数据平滑, 窗口设为1, 最后对去噪后的数据进行归一化处理。
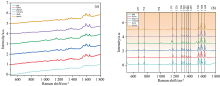
图2(a)为原始光谱, 图2(b)为预处理后的光谱图, 六种动物共有的特征峰位于677, 754, 974, 1 174, 1 228, 1 306, 1 340和1 377 cm-1等位置, 通过查阅文献, 这些谱峰对应的分子振动模态如表1[8, 9, 10, 11, 12], 未在图中标注的峰代表血红蛋白、 氨基酸等血液成分中的生物分子的振动, 仅从特征峰的位置无法准确分辨, 需要通过算法进一步特征提取和分类。
| 表1 六种动物全血特征峰对应分子振动模态 Table 1 Molecular vibration modes corresponding to characteristic peaks of six animal’ s whole blood |
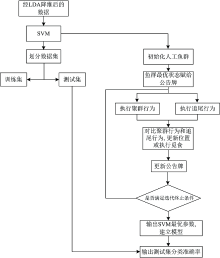
通过算法进行物种识别的时候, 主要分为两个步骤: 降维和分类。 总体的算法流程如图3所示。
2.2.1 降维
光谱数据中含有大量的冗余信息, 需要对预处理后的光谱数据进行降维处理, 为下一步分类做准备。 采用线性判别分析(LDA)的方法, 考虑数据的标签, 是一种有监督的降维方法[13, 14], 目标是使投影的方向类内方差小, 类间方差大, 被广泛地用于降维和特征提取。
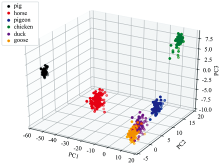
预处理后的光谱数据为406维(拉曼频移在500~1 800 cm-1范围内有406个像素点), 将其降至3维, 前三个主成分的贡献率分别为86.4%, 7.3%, 3.7%, 总计达到97.4%。 猪和马作为哺乳动物, 血液成分和禽类有很大差别, 而四种家禽中, 鸡和鸽与其他两种区分度较大, 鸭和鹅同属雁形目动物, 光谱相似性较高, 难以完全区分。 图4为使用LDA对光谱数据降维后的可视化结果。
2.2.2 分类
采用支持向量机[15](SVM)对降维后的血液数据进行分类, 以准确率最高为目标, 采用人工鱼群算法(AFSA)对支持向量机(SVM)的关键参数进行优化。
SVM的核函数选用高斯核(RBF), 把样本数据映射到高维空间, RBF核应用范围最广且对数据有良好的适应性, 相比于多项式核函数, 需要更少的参数。 RBF核有两个重要的参数, 惩罚因子C和γ。 C表示对分类误差的宽容度, γ做为支持向量影响半径的倒数, 调节这两个参数。
人工鱼群算法(AFSA)是Li提出的一种仿生优化算法[16]。 模仿人工鱼的聚群、 追尾和觅食行为, 通过多条人工鱼同时进行寻优, 达到鱼群中各个体的局部寻优, 从而在群体中凸显全局最优解。 (C, γ)为每条人工鱼Xi, 支持向量机准确率为F, 目标为找到F(C, γ)的最大值, 以测试集的分类准确性作为鱼群食物浓度的好坏评价, 建立如式(1)和式(2)模型
具体的寻优过程为图5(a)所示, 相比于传统的网格搜索法, 人工鱼群算法可以在连续的超参数空间进行搜索, 不局限于离散的网格点, 可以更好地发现超参数C和γ之间的非线性关系, 提高搜索效率, 更快收敛。 同时, AFSA算法还具有较好的全局搜索能力, 能够在参数空间中广泛搜索, 避免陷入全局最优解。 初始化鱼群时, 设置一些固定参数值, 鱼群规模为50, 最多试探次数try_number=200, 感知距离visiual=0.6, delta=0.618, 最大迭代次数MAXGEN=80, 步长step=0.5。 使用AFSA优化支持向量机参数的过程如图7所示, 最终得到最优的C为0.20, g为0.134, 得到的测试集的准确率为97.2%。 参数寻优的结果如图5(b)所示。
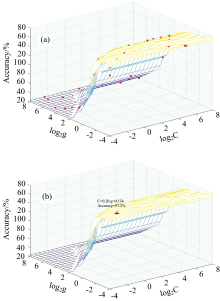 | 图5 (a) AFSA-SVM模型收敛过程, (b) AFSA-SVM寻优结果Fig.5 (a) convergence process of AFSA-SVM model, (b) optimum results of AFSA-SVM |
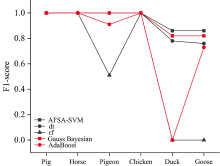
为了表明AFSA-SVM算法的优越性, 与决策树dt[17]、 随机森林rf[18]、 高斯朴素贝叶斯bnb、 Adaboost[19]四种算法进行比较, 几种算法均按照训练集0.7, 测试集0.3的比例划分。 分别从测试集整体准确率和F1分数两个维度来考量。 在准确率方面, 五种算法的准确率分别为97.2%, 93.7%, 93.7%, 84.6%和68.5%, 如表2所示。 AFSA-SVM具有显著优势。 F1分数[20]是精确率precision与召回率recall的调和平均数, 如式(3), 只有当精确率和召回率都大的时候, F1才有较大取值。 可以看出, AFSA-SVM, 决策树和随机森林的F1都达到了1, 表示对猪、 马、 鸽、 鸡的分类准确性达到了100%, 鸭和鹅的准确率AFSA-SVM略高于决策树和随机森林, 高斯贝叶斯和Adaboost对鸽、 鸭、 鹅的分类效果都比AFSA-SVM逊色很多。
| 表2 五种算法的实验结果 Table 2 Accuracy of five algorithms |
| 表3 五种算法的F1-score实验结果 Table 3 F1-scores of five algorithms |
由算法的分类结果, 对哺乳动物的分类效果优于禽类, 鹅和鸭同属雁形目下的鸭科, 亲缘关系非常相近, 反映在光谱上的差异也不明显, 故这两个物种的分类效果不如其他四种。 可从表2和图6直观地看出5种算法的分类结果。
采用实验室自主研发的生物特征光谱识别仪, 检测动态血液的拉曼光谱, 创新了检测方式, 解决了传统检测方式(比如涂片法)样品易变质, 不明来源样本具有传播疾病的风险等问题, 最大程度降低了激光对血液的损伤。
为了证明该仪器检测的可行性和有效性, 选用六种常见的家禽家畜新鲜血液, 样品处理后放进仪器观测其光谱。 对光谱进行去基线、 归一化等预处理后对比六个物种的平均光谱, 发现六个物种的全血光谱在很多相同位置存在特征峰。 可能因为血液成分相差不大, 单从特征峰位置难以准确分类, 需要借助算法的手段, 在分类前通过有监督的降维方法LDA对数据进行降维。
提出AFSA-SVM的方法, 通过人工鱼群算法AFSA找到最优的支持向量机参数, 即C=0.20, g=0.134, 并与决策树dt、 随机森林rf、 高斯朴素贝叶斯、 adaboost四种算法进行比较, 无论是从整体准确率还是F1-score, 都优于其他算法。 说明本课题组提出的这种检测方法, 结合提出的AFSA-SVM算法可以对不同物种的血液进行有效鉴别。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|