作者简介: 于 水, 1998年生, 长春理工大学物理学院硕士研究生 e-mail: yushui1206@163.com
近红外光谱分析已成为工农业生产过程质量监控领域中不可或缺的重要分析手段之一, 在食品、 农业、 医药等定性定量分析领域被广泛应用。 预测精度高、 运行速度快、 泛化能力强的近红外光谱预测模型可用于不同物质的定性定量分析。 但由于近红外光谱数据量的激增, 传统的近红外光谱建模方法已经出现明显的不足。 随着人工智能技术的不断发展, 深度学习算法在近红外光谱分析领域得到了广泛应用。 提出了一种基于并联卷积神经网络的近红外光谱定量分析模型(PaBATunNet)。 该模型由1个一维卷积层、 1个并联卷积模块(Module)、 1个展平层、 4个全连接层和1个参数调节器(PR)组成, Module模块包括5个子模块分别对光谱数据进行线性及非线性多维特征提取, 并通过Concatenate函数将提取后的光谱特征数据进行拼接, PR模块通过调节优化PaBATunNet模型参数, 提高模型预测精度。 基于Gard-CAM思想给出了PaBATunNet模型高贡献度特征波长, 增加了PaBATunNet模型的可解释性。 以谷物、 柴油、 啤酒、 牛奶四组公开的近红外光谱数据为例, 将PaBATunNet模型的预测结果与偏最小二乘(PLS)、 主成分回归(PCR)、 支持向量机(SVM)和BP神经网络(BP)模型的预测结果进行比较。 结果表明, 与PLS相比, PaBATunNet模型在谷物、 柴油、 啤酒、 牛奶数据集的预测精度上分别提高了30.0%、 40.7%、 43.0%、 52.8%; 与PCR相比, PaBATunNet模型的预测精度分别提高了28.8%、 35.9%、 40.8%、 52.2%; 与SVM相比, PaBATunNet模型的预测精度分别提高了45.5%、 37.4%、 45.3%、 54.7%; 与BP相比, PaBATunNet模型的预测精度分别提高了7.9%、 32.4%、 90.1%、 62.0%。 基于并联卷积神经网络的近红外光谱建模方法相比于传统建模方法解决了模型预测精度低、 运行时间长、 泛化能力差以及可解释性不强等问题, 可有效应用于工农业生产中不同物质的定量分析, 为建立快速、 无损、 高精度的近红外光谱定量分析模型提供了科学基础。
Near-infrared spectroscopy has become an indispensable analysis method in industrial and agricultural production process quality monitoring. It has been widely used in the qualitative and quantitative analysis of food, agriculture, medicine and others.-A near-infrared spectroscopy prediction model with high prediction accuracy, high-speed running,and strong generalization ability plays an essential role in the qualitative and quantitative analysis of different substances. However, due to the increase innear-infrared spectroscopy data, the disadvantages of traditional near-infrared spectroscopy modeling methods are obvious. With the development of artificial intelligence technology, deep learning algorithms have been widely used in the field of near-infrared spectroscopy. The quantitative analysis model of near-infrared spectroscopy based on a parallel convolution neural network (PaBATunNet) was proposed. PaBATunNet comprisedone1-D convolutional layer, one parallel convolution module (Module), one flattening layer, four fully connected layers and one parameter regulator (PR).The Module included five submodules and one Concatenate function, which was used to extract the linear and nonlinear multidimensional features of the spectral data, respectively and concatenate them. The prediction accuracy of PaBATunNet was improved by PR, which optimized the model parameters. The high contribution characteristic wavelengths of PaBATunNet were given based on Gard-CAM, which improved the interpretability of PaBATunNet. By taking public near-infrared spectroscopy datasets of grain, diesel fuel, beer and milk as examples, the prediction results of PaBATunNet were compared with partial least squares (PLS),principal component regression (PCR), support vector machine (SVM) and back propagation neural network (BP). The results showed that the prediction accuracies of PaBATunNet tograin, diesel fuel, beer and milk datasets were respectively increased by 30.0%, 40.7%, 43.0% and 52.8% in comparison with PLS, 28.8%, 35.9%, 40.8% and 52.2% in comparison with PCR, 45.5%, 37.4%, 45.3% and 54.7% in comparison with SVM, and 7.9%, 32.4%, 90.1% and 62.0% in comparison with BP. Compared with the traditional near-infrared spectroscopy modeling methods, the PaBATunNet based on the parallel convolutional neural network, has solved the problems of low prediction accuracy, long running time, poor generalization ability and poor interpretability. It can be effectively applied to quantitative analysis in industrial and agricultural production. It provides a theoretical basis for establishing the rapid, nondestructive and high-precision near-infrared spectroscopy quantitative analysis model.
近红外光谱分析(near infrared spectroscopy, NIRS)技术在大型流程工业的应用日趋成熟和广泛, NIRS技术正在以产业链的方式应用于诸多领域, 如农业[1]、 石化[2]、 医药[3]、 食品[4]和饲料[5]等。 NIRS技术根据样本含氢基团(主要是CH、 OH、 NH等)对近红外光的吸收差异, 结合化学计量学方法, 可以快速高效地测定样本中的化学组成和物化性质, 从而进行物质含量的定性定量分析[6, 7]。 目前, 如何构建预测精度高、 泛化能力强的近红外光谱预测模型是NIRS技术的重点和难点。 常用的传统近红外光谱建模方法有偏最小二乘(partial least squares, PLS)[8]、 主成分回归(principal component regression, PCR)[9]、 支持向量机(support vector machine, SVM)[10]、 BP神经网络(back propagation neural network, BP)[11]等。 但随着近红外光谱数据量的激增, 传统的近红外光谱建模方法在预测精度上难以达到预期效果, 如PLS和PCR表现为对线性特征拟合好, 但是对于非线性特征拟合较差, 使得模型无法取得较好的预测精度; SVM模型在处理大数据时容易出现模型过拟合; BP模型在建模过程中的运行时间长, 预测精度低等。 近年来, 随着人工智能技术的不断发展, 以卷积神经网络(convolutional neural network, CNN)为代表的深度学习算法在近红外光谱分析领域得到了广泛应用。 Aulia[12]等利用深度置信网络和CNN建立婴儿食品中营养元素含量的近红外光谱定量分析模型; Mishra[13]等利用一维卷积神经网络和光谱预处理算法对芒果果实中干物质进行了近红外光谱定量分析; Zhang[14]等利用端到端卷积神经网络对玉米蛋白质、 小麦蛋白质、 土壤有机碳等进行了近红外光谱定量分析; Ma[15]等利用时频分析结合一维卷积神经网络对烟草样本中的总糖、 还原糖、 尼古丁等化学成分含量进行了近红外光谱定量分析。 然而, 基于CNN的近红外光谱模型仍然存在着模型运行速度慢、 预测精度低、 稳健性差和可解释性不强等问题。
为此, 提出了一种基于并联加宽自适应调参的卷积神经网络近红外光谱定量分析模型(parallel broadening adaptive tuning network, PaBATunNet)。 该模型基于Inception网络块[16]思想, 利用并联卷积模块(Module)增加了预测模型宽度, 降低了预测模型深度, 提升了预测模型运行速度及预测精度。 通过设置PaBATunNet模型卷积层、 池化层的卷积核大小、 特征向量数量, 实现对近红外光谱数据的线性及非线性多维特征提取, 并学习多维特征信息, 减少模型运算量, 防止模型过拟合; 根据预测值与真实值之间偏差, 通过参数调节器(parameter regulator, PR)不断优化参数, 通过全连接层的线性变换, 提升模型的预测精度及稳健性。 基于梯度加权类激活映射(gradient-weighted class activation mapping, Gard-CAM)[17]思想, 选取不同数据集的近红外光谱高贡献度特征波长, 增加PaBATunNet模型的可解释性。 应用在谷物、 柴油、 啤酒、 牛奶四组公开近红外光谱数据集, PaBATunNet模型相比PLS、 PCR、 SVM和BP神经网络模型, 表现出预测精度更高、 稳健性更好。
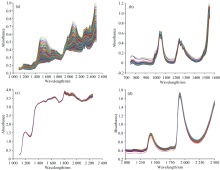
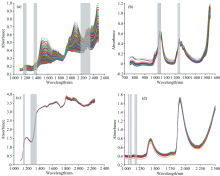
谷物数据集包含231个谷物样本的近红外光谱数据以及对应的酪蛋白含量, 主要目的是预测酪蛋白含量。 谷物近红外光谱是Tormod Naes和Tomas Isaakson测量得到, 波长范围为1 104~2 495 nm, 波长间隔为12 nm, 共117个波长。 根据Kennard-Stone(K-S)方法[18]对谷物数据集进行划分, 将谷物样本划分为153个校正集样本和78个预测集样本。 谷物样本的近红外光谱如图1(a)所示, 从谷物光谱图中可以看出, 1 490~1 540 nm的吸收峰为C=O键和N— H键伸缩振动的第二倍频区, 与酪蛋白中的氨基酸等物质相关[19]; 在2 054 nm处的光谱特征反映了蛋白质N— H键的吸收; 在2 120 nm处的光谱吸收与氨基酸的C=O伸缩倍频有关[20]。 谷物样本的酪蛋白含量分布如表1所示。 谷物数据集可从https://eigenvector.com/resources/data-sets/下载。
| 表1 校正集和预测集中物质含量统计分布 Table 1 Statistical distribution of substance contents in calibration set and prediction set |
柴油数据集包含394个柴油样本的近红外光谱数据以及对应的总芳烃含量, 主要目的是预测总芳烃含量。 柴油近红外光谱是美国西南研究所测量得到, 波长范围为750~1 550 nm, 波长间隔为2 nm, 共401个波长。 根据K-S方法对柴油数据集进行划分, 将柴油样本划分为318个校正集样本和76个预测集样本。 柴油样本的近红外光谱如图1(b)所示, 从柴油光谱图中可以看出, 1 050~1 070和1 220 nm的吸收峰为结合O— H键和C— H键的伸缩振动的第二倍频区, 与芳烃中苯环类物质吸收相关[21]。 柴油样本的总芳烃含量分布如表1所示。 柴油数据集可从https://eigenvector.com/resource/data-set/下载。
啤酒数据集包含60个啤酒样本的近红外数据以及对应的酵母含量, 主要目的是预测酵母含量。 啤酒近红外光谱波长范围为1 100~2 250 nm, 波长范围为2 nm, 共576个波长。 根据K-S方法对啤酒数据集进行划分, 将啤酒样本划分为40个校正集样本和20个预测集样本。 啤酒样本的近红外光谱如图1(c)所示, 从啤酒光谱图中可以看出, 1 200~1 220 nm的吸收峰与O— H键伸缩振动的第一倍频区相关, 与酵母等物质成分吸收相关[22]。 啤酒样本的酵母含量分布如表1所示。 啤酒数据集来源于参考文献[23]。
牛奶数据集包含67个牛奶样本的近红外数据以及对应的蛋白质含量, 主要目的是预测蛋白质含量。 牛奶近红外光谱的每个光谱扫描32次, 取平均值, 扫描间隔为4 cm-1, 波数范围为4 000~10 000 cm-1。 根据K-S方法对牛奶数据集进行划分, 将牛奶样本划分为50个校正集样本和17个预测集样本。 牛奶样本的近红外光谱如图1(d)所示, 从牛奶光谱图中可以看出, 1 400~1 430 nm的吸收峰与O— H键伸缩振动的第一倍频区和C— H键伸缩振动有关; 1 910~1 940 nm的吸收峰与O— H键伸缩振动的第二和第三倍频区有关, 与蛋白质中氨基酸等物质吸收相关[24]。 牛奶样本的蛋白质含量分布如表1所示。 牛奶数据集来源于参考文献[25]。
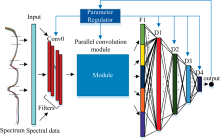
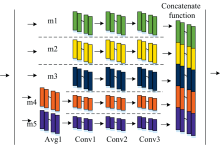
PaBATunNet模型由1个一维卷积层(Conv0)、 1个并联卷积模块(Module)、 1个展平层(F1)、 4个全连接层(D1、 D2、 D3、 D4)和1个参数调节器(PR)组成, 其网络结构如图2所示。 其中, Module模块由5个子模块m1、 m2、 m3、 m4、 m5和Concatenate函数构成, 其网络结构如图3所示。 m1、 m2、 m3子模块均由3个一维卷积层Conv1、 Conv2、 Conv3构成, m4、 m5子模块均由1个一维平均池化层Avg1和3个一维卷积层Conv1、 Conv2、 Conv3构成, Concatenate函数对五个子模块处理后的光谱特征向量在第一维度(即特征向量维度)进行顺序拼接, 重构一个包含多重特征的新光谱。 PaBATunNet模型中一维卷积层使用tanh激活函数, 不使用填充; 平均池化层和全连接层均不使用激活函数, 不使用填充。 PaBATunNet模型采用RMSprop优化器, 初始学习率设置为0.001, 学习衰减速率设置为0.9, 使用均方误差(mean squared error, MSE)损失函数对模型进行训练, 使用PR进行模型参数寻优, 不同数据集的最优模型参数如表2所示。 PaBATunNet模型详细代码见https://github.com/YuShui18/PaBATunNet.git。
| 表2 PaBATunNet在四个数据集上的参数 Table 2 PaBATunNet parameters for four datasets |
PaBATunNet模型是基于一维卷积神经网络搭建, 采用了并联卷积神经网络模式, 引入了Inception网络块思想, 增加了网络模型宽度, 降低了网络模型深度。 原始近红外光谱数据经过卷积层、 池化层和全连接层的特征提取、 筛选和回归, 形成光谱数据与待测样本化学值之间的函数关联。 PaBATunNet模型的具体建立步骤如下:
(1)测量样本的近红外光谱数据X和化学值数据Y, 并运用K-S算法将样本划分为校正集和预测集;
(2)模型迭代次数设置为100, 批量归一化参数设置为1, 学习率设置为0.001, 学习率衰减速率设置为0.9;
(3)使用PR生成一组初始参数, 若所生成的参数均被选择, 则跳转至第11步;
(4)原始光谱数据进入Conv0层, 通过卷积运算得到filter0个通道的特征向量;
(5)经过Conv0层卷积运算后的数据进入Module模块, 并同时进入m1、 m2、 m3、 m4、 m5;
① m1、 m2、 m3: filter0个通道的特征向量经过Conv1卷积运算得到filter1个通道的特征向量, filter1个通道的特征向量经过Conv2卷积运算得到filter2个通道的特征向量, filter2个通道的特征向量经过Conv3卷积运算得到filter3个通道的特征向量。
② m4、 m5: filter0个通道的特征向量经过Avg1层平均池化, 计算每个子区域的平均值, 平均池化处理后的数据再经过Conv1、 Conv2、 Conv3卷积运算得到filter3个通道的特征向量。
(6)经过m1、 m2、 m3、 m4、 m5层处理后的数据通过Concatenate函数在第一维度进行光谱数据顺序拼接, 重构一个新的特征光谱;
(7)F1层将新特征光谱展平为一维向量;
(8)展平后的数据经过D1、 D2、 D3、 D4层后, 得到一个预测值;
(9)通过MSE计算真实值与预测值偏差, 根据偏差值使用RMSprop优化器对模型参数进行优化;
(10)如果没有达到设定的迭代次数, 则跳转到第3步;
(11)训练结束后保存最优模型参数, 保存PaBATunNet预测模型。
采用的模型评价参数分别为校正均方根误差(root-mean-square error of calibration, RMSEC)、 校正集决定系数(calibration determination coefficient,
式中,
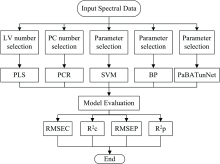
对谷物、 柴油、 啤酒、 牛奶四个公共近红外光谱数据集分别建立了PLS、 PCR、 SVM、 BP和PaBATunNet模型, 具体实验设计如图4所示。 PaBATunNet模型使用Keras深度学习库搭建; PLS、 PCR、 SVM和BP模型使用Scikit-learn机器学习库搭建。 对于谷物、 柴油、 啤酒、 牛奶四个公共近红外光谱数据集, PLS选取最优的潜在变量(LVs)分别为7、 6、 7、 10, 最大迭代次数为500; PCR选取的最优主成分数分别为6、 7、 9、 8; SVM使用GridSearchCV自动搜索来获得最佳超参数; BP隐含层设置为6层, 第一层神经元个数为350, 第二层神经元个数为300, 其余四层用循环迭代的方法来确定。 最终统计PaBATunNet、 PLS、 PCR、 SVM和BP神经网络模型的预测结果。
基于Gard-CAM思想处理得出了PaBATunNet模型的高贡献度特征波长。 Gard-CAM原理是对于一个训练好的分类网络, 使用梯度的全局平均来计算得到最后一层特征图, 此特征图代表了不同类别的贡献程度, 即权重。 利用权重将特征图合在一起, 得到对于某一类别的贡献度波长图[17]。
基于不同数据集的PaBATunNet高贡献度特征波长分布如图5所示。 谷物数据集的PaBATunNet高贡献度特征波长分布如图5(a)所示, 其高贡献度特征波长为1 164~1 224、 1 344~1 404和2 184~2 364 nm。 其中, 波长1 164~1 224 nm与C— H键伸缩振动的第二倍频区相一致, 1 344~1 404 nm与C— H合频(— CH3键伸缩振动的第一倍频区和— CH2键伸缩振动的第一倍频区)相一致, 2 184~2 364 nm与C— H合频(— CH3键伸缩振动的基频区和— CH2键伸缩振动的基频区)相一致[26]。 柴油数据集的PaBATunNet高贡献度特征波长分布如图5(b)所示, 其高贡献度特征波长为1 030~1 060和1 230~1 250 nm。 其中, 波长1 030~1 060 nm与O— H键伸缩振动的第二倍频区相一致, 1 230~1 250 nm与C— H键伸缩振动的第二倍频区相一致[27]。 啤酒数据集的PaBATunNet高贡献度特征波长分布如图5(c)所示, 其高贡献度特征波长为1 150~1 170和1 260~1 350 nm。 其中, 波长1 150~1 170和1 260~1 350 nm与C— H键伸缩振动的第二倍频区相一致[28]。 牛奶数据集的PaBATunNet高贡献度特征波长分布如图5(d)所示, 其高贡献度特征波长为1 065~1 070、 1 080~1 095和1 175~1 200 nm。 其中, 1 065~1 070和1 080~1 095 nm与O— H键伸缩振动的第二倍频区相一致, 1 175~1 200 nm与C— H键伸缩振动的第二倍频区相一致[29]。 综上, PaBATunNet模型的高贡献度特征波长均与被测物质的化学官能团相关, 进一步表征了谷物、 柴油、 啤酒和牛奶的近红外光谱与其被测化学值之间的内在关联, 增加了PaBATunNet模型的可解释性。
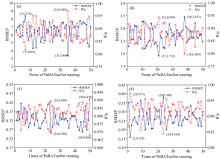
将谷物、 柴油、 啤酒、 牛奶的近红外光谱数据采用PaBATunNet建模方法均运行50次, 不同数据集PaBATunNet模型的RMSEP值和
使用PLS、 PCR、 SVM、 BP和PaBATunNet五种建模方法分别对谷物、 柴油、 啤酒、 牛奶的近红外光谱数据进行建模, 每种建模方法均运行50次。 PLS、 PCR、 SVM、 BP和PaBATunNet模型在谷物中酪蛋白含量、 柴油中总芳烃含量、 啤酒中酵母含量、 牛奶中蛋白质含量的建模统计结果如表3所示。
| 表3 不同数据集的不同建模方法统计结果 Table 3 Statistical results of different modeling methods for different datasets |
由表3可以看出, 在谷物、 柴油、 啤酒、 牛奶四组公开近红外光谱数据集上, PaBATunNet模型与PLS、 PCR、 SVM和BP模型相比, 其预测精度最高、 稳健性最好。 在谷物酪蛋白含量的预测中, PaBATunNet模型与PLS、 PCR、 SVM、 BP模型相比, RMSEP值分别由9.248、 9.090、 11.870、 7.026下降到6.471, 预测精度分别提升了30.0%、 28.8%、 45.5%、 7.9%; 对柴油中总芳烃含量的预测, PaBATunNet模型与PLS、 PCR、 SVM、 BP模型相比, RMSEP值分别由4.223、 3.909、 3.999、 3.706下降到2.504, 预测精度分别提升了40.7%、 35.9%、 37.4%、 32.4%; 对啤酒中酵母含量的预测, PaBATunNet模型与PLS、 PCR、 SVM、 BP模型相比, RMSEP值分别由0.581、 0.559、 0.605、 3.345下降到0.331, 预测精度分别提升了43.0%、 40.8%、 45.3%、 90.1%; 对牛奶中蛋白质含量的预测, PaBATunNet模型与PLS、 PCR、 SVM、 BP模型相比, RMSEP值分别由0.568、 0.561、 0.592、 0.705下降到0.268, 预测精度分别提升了52.8%、 52.2%、 54.7%、 62.0%。
在谷物、 柴油、 啤酒、 牛奶数据集的预测结果中, PLS、 PCR、 SVM、 BP模型预测结果的RMSEC值均表现较好, 但其RMSEP值均表现较差, 说明这些方法在建模过程中均产生了严重的过拟合现象, 而PaBATunNet模型预测结果的RMSEC值和RMSEP值均表现稳定, 说明该模型不存在过拟合现象; PaBATunNet模型在不同数据集上的RPD平均值均优于PLS、 PCR、 SVM、 BP预测模型, 说明PaBATunNet模型具有较强的泛化能力。
综上, PaBATunNet模型采用并联卷积网络模块更多的提取了原始光谱的多维特征信息, 通过Concatenate函数在第一维度对五个子层生成的多维特征信息进行了顺序融合, 重构了包含多重特征的新光谱, 实现了光谱特性的全面表征, 通过池化和正则化方法避免了模型过拟合, 提高了模型运行速度, 通过参数调节器和全连接层的线性变换, 提升了模型的预测精度及稳健性。 相比于传统的PLS、 PCR、 SVM和BP建模方法, PaBATunNet建模方法更好地解决了预测模型的非线性表征问题以及预测模型的过拟合问题。
PaBATunNet模型是一种基于并联卷积神经网络的自适应调参近红外光谱定量分析模型。 该模型引入了Inception网络块思想, 将多个卷积层、 池化层并行连接在一起, 增加了网络宽度, 降低了网络深度, 使模型运行速度更快, 并通过多个卷积层、 池化层对原始光谱数据进行多维特征提取, 极大程度提升了模型的预测精度。 基于Gard-CAM思想, 选取不同数据集的近红外光谱高贡献度特征波长, 表征了近红外光谱与被测化学值之间的内在关联, 增加PaBATunNet模型的可解释性。 利用谷物、 柴油、 啤酒和牛奶的公共近红外光谱数据库建立了PaBATunNet预测模型, 结果表明, 与PLS、 PCR、 SVM和BP模型相比, PaBATunNet模型具有更好的预测精度和泛化能力。 PaBATunNet建模方法有效简化了模型, 提升了模型运行速度, 提高了模型预测精度, 解决了传统近红外光谱建模过程中的非线性表征问题及模型过拟合问题。 利用PaBATunNet对近红外光谱数据进行建模具备可行性, PaBATunNet模型具有良好的应用前景。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|