作者简介: 陈凤霞, 1994年生, 云南农业大学资源与环境学院硕士研究生 e-mail: cfx0909@126.com
牛肝菌作为一种著名的野生食用菌, 具有较高的食用价值和经济价值。 牛肝菌种类繁多, 不易区分, 建立一种有效、 快速、 可信的种类鉴别技术, 可为牛肝菌提高品质提供一种方法。 本研究采集云南不同地区7种野生牛肝菌共计683株, 获取样品中红外光谱和紫外光谱, 分析不同种类牛肝菌平均光谱图特征。 基于多种预处理组合(SNV+SG, 2D+MSC+SNV, 1D+MSC+SNV+SG, MSC+2D)的单一光谱数据结合两种特征值提取法(PCA, LVs)构建了偏最小二乘法判别分析与随机森林算法并结合数据融合策略对牛肝菌进行种类鉴别, 有一定的创新性。 结果表明: (1)中红外光谱和紫外光谱的不同种类牛肝菌平均光谱吸收峰差异较小, 吸光度具有细微差异。 (2)合适的预处理可提高光谱数据信息, 偏最小二乘法判别分析和随机森林算法模型的中红外光谱数据和紫外光谱数据最佳预处理组合为2D+MSC+SNV, SNV+SG, 2D+MSC+SNV, 1D+MSC+SNV+SG。 (3)单一光谱模型中, 中红外光谱模型优于紫外光谱模型, 中红外光谱最佳预处理组合2D+MSC+SNV的偏最小二乘法判别分析模型正确率训练集99.78%, 验证集99.12%; 随机森林模型正确率训练集93.20%, 验证集99%。 (4)数据融合策略提高了分类正确率, 低级融合的偏最小二乘法判别分析模型训练集和验证集正确率为100%, 99.12%。 随机森林模型训练集和验证集正确率为92.32%, 99.14%。 (5)随机森林算法中级数据融合Latent variable(LVs)正确率为训练集92.76%, 验证集96.04%; 中级数据融合Principal components analysis(CPA)正确率为训练集97.15%, 验证集100%。 (6)偏最小二乘法判别分析中级数据融合(LVs)正确率为训练集100%, 验证集99.56%; 中级数据融合(CPA)训练集和验证集正确率均能达到100%。 基于偏最小二乘法判别分析和随机森林算法结合数据融合策略对牛肝菌进行种类鉴别, 鉴别效果理想。 偏最小二乘法判别分析中级数据融合(CPA)可作为一种低成本高效率的牛肝菌种类鉴别技术。
As a famous wild edible mushroom, boletus has great edible and economic value. There are many kinds of boletus, and it is not easy to distinguish. An effective, rapid and credible species identification technology can be established to improve the quality of boletus.In this study, a total of 683 strains of 7 species of wild bolete from different regions of Yunnan were collected, the infrared and ultraviolet spectra of the samples were obtained, and the average spectral characteristics of different kinds of bolete were analyzed. Based on the single spectral data of multiple preprocessing combinations (SNV+SG, 2D+MSC+SNV, 1D+MSC+SNV+SG, MSC+2D) combined with two feature value extraction methods (PCA, LVs), the partial least squares discrimination analysis and random forest algorithm combined with data fusion strategy to identify the species of boletus.There is a certain degree of innovation. The results show: (1) The average spectral absorption peaks of different types of boletus in the mid-infrared spectrum and the ultraviolet spectrum have small differences, and the absorbance has subtle differences. (2) Appropriate preprocessing can improve spectral data information. The best preprocessing combination of mid-infrared spectral data and ultraviolet spectral data for partial least square discriminant analysis and random forest algorithm model is 2D+MSC+SNV, SNV+SG, 2D +MSC+SNV, 1D+MSC+SNV+SG. (3) The mid-infrared spectroscopy model is better than the ultraviolet spectroscopy model in the single spectrum model. The partial least squares discriminant analysis model of the best preprocessing combination of mid-infrared spectroscopy 2D+MSC+SNV has a correct rate of 99.78% in the training set and 99.12% in the validation set. The accuracy of the random forest model is 93.20% on the training set and 99% on the validation set. (4) The data fusion strategy improves classification accuracy. The accuracy of the low-level fusion partial least squares discriminant analysis model training set and validation set is 100%, 99.12%. The accuracy of the random forest model's training set and validation set are 92.32% and 99.14%. (5) Random Forest Algorithm Intermediate Data Fusion latent variable (LVs) training set 92.76%, validation set 96%; Intermediate Data Fusion principal components analysis (CPA) training set 97.15%, validation set 100%. (6) Partial Least Squares Discriminant Analysis Intermediate Data Fusion (LVs) training set is 100%, and validation set is 99.56%; the accuracy of intermediate data fusion (CPA) training set and validation set can reach 100%. Based on the discriminant analysis of the partial least squares method and random forest algorithm combined with data fusion strategy, the species identification of boletus is satisfactory. Partial Least Squares Discriminant Analysis Intermediate Data Fusion (CPA) can be used as a low-cost and high-efficiency technology for identifying boletus species.
牛肝菌(Boletus)属担子菌亚门, 伞菌目牛肝菌科(Boletaceae)和松塔牛肝菌科(Strobilomycetaceae)统称[1]。 中国已知牛肝菌种类390种, 其中3/5可食用。 牛肝菌是著名的野生食用菌, 具有较高的食用价值和药用价值[2], 我国凭借得天独厚的地理条件成为食用菌主要生产和出口大国[3]。 野生牛肝菌种类混杂, 难以分辨, 建立一种快速有效的牛肝菌鉴别方法, 可提高牛肝菌品质, 保障消费者健康。
数据融合将多源数据进行联合以便获得更多信息[4]。 近年来, 光谱数据融合结合机器学习进行食用菌鉴别研究较为普遍[5]。 胡翼然等[6]采用随机森林结合红外光谱数据融合法成功找到绒柄牛肝菌产地鉴别技术。 Yao等[7]的研究表明红外光谱和紫外光谱结合数据融合用于牛肝菌产地及种类区分具有可靠性。 光谱指纹图谱技术具有低成本、 快捷、 易获取等特点, 常用于质量检测及食用菌研究[8]。 光谱指纹图谱结合数据融合策略是将同一研究物的多个不同来源数据信息用化学计量学方法进行优化重组。 现目前, 数据融合策略在药品、 食品等领域研究较为广泛[9]。 Li等[10]利用偏最小二乘法判别分析和随机森林进行光谱数据融合技术, 寻找到灵芝种类鉴别分析法。 Yao等[11]研究了232株食用菌, 采用支持向量机和随机森林结合数据融合分析鉴别野生食用菌和栽培食用菌, 为食用菌分类提供了准确有效的方法。 偏最小二乘法判别分析与随机森林基于样本数据建立数学模型, 在菌类研究中使用广泛且效果显著。
光谱数据准确性受各种背景影响, 如: 噪音、 基线漂移、 随机误差等, 进行光谱数据预处理, 可提高分辨率和灵敏度。 本工作采用预处理后的中红外光谱和紫外光谱结合偏最小二乘法判别分析与随机森林算法对牛肝菌种类进行单一光谱和数据融合数据模型分析, 寻找出最优牛肝菌种类鉴别方法, 为牛肝菌质量控制和保障消费者食用安全提供借鉴。
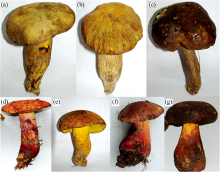
采集云南各地7种成熟牛肝菌共计683份, 其中灰褐牛肝菌98株、 美味牛肝菌221株、 栗色牛肝菌110株、 小美牛肝菌32株、 皱盖疣柄牛肝菌132株、 双色牛肝菌30株、 绒柄牛肝菌60株, 详见图1和表1, 均由云南农业大学刘鸿高教授鉴定。 样品初始处理用陶瓷刀刮去泥土, 自来水清洗干净后用超纯水润洗三遍; 晾干表面水分后置于恒温烘箱50 ℃烘干至恒重。 不同种类牛肝菌分类粉碎后使用100目标准筛过筛保存。
| 表1 牛肝菌样品信息 Table 1 Boletus samples information |
Frontier型傅里叶变换红外光谱仪, 美国Perkin Elmer公司; TU-1901紫外-可见分光光度计, 日本岛津公司; AR1140型电子分析天平, 上海升隆电子科技有限公司; 100目标准筛盘, 浙江上虞市道墟五四仪器厂; FW-100型高速粉碎机, 天津市华鑫仪器厂; UPT-I-10超纯水机, 优谱科技有限公司。 分析纯溴化钾, 天津风船化工科技有限公司; 氯仿, 西陇化工股份有限公司。
中红外光谱: 采用电子分析天平称取牛肝菌样品粉末(1.2± 0.2) mg, 溴化钾粉末(150± 0.5) mg置于玛瑙研钵中混合研磨成均匀细粉于压片机制成薄片, 光谱仪用溴化钾片进行空白扫描, 扣除背景影响, 每个样本重复扫描三次, 信号累计扫描16次, 范围在4 000~400 cm-1, 分辨率为4 cm-1。
紫外光谱: 称取牛肝菌样品粉末(150± 0.5) mg, 将称取好的样品置于25 mL试管加入10 mL氯仿封口, 超声提取40 min, 三层滤纸过滤取清液, 光谱仪预热1 h, 扣除背景后进行光谱扫描, 采样间隔0.3 min, 每个样本重复三次, 取平均光谱。
将整理好的光谱数据进行数据预处理, 使用OMNIC软件进行自动基线校正和吸光度转换, SIMCA软件进行Savitzky-Golay(SG)、 Standard normal variables(SNV)、 Multiple scattering correction(MSC)、 First derivative(1D)、 Sec-ond derivatives(2D)联合预处理, 不同的光谱预处理具有不同的作用和优势, 结合实际合理选择最佳的预处理方法, 将不同预处理组合后的数据用Kennard-Stone算法分为2/3(456)的训练集合1/3(227)的验证集, 用于模型建立。 先进行单一光谱数据建模, 寻找到最佳预处理建模结果后, 将最佳数据集组合用于建立数据融合模型。 将两种单一光谱数据进行联合分析, 以增加信息量。 研究中使用两种算法(偏最小二乘法判别分析和随机森林)进行牛肝菌样品模型建立, 寻找最佳种类分类模型, 达到牛肝菌种类鉴别目的。
图2为中红外和紫外光谱图, 图中为7种牛肝菌样品的平均光谱。 由图2(a)可知, 波数范围在3 700~2 750 cm-1之间为主要强吸收峰波段, 不同种类牛肝菌吸收峰差异较小但具有细微差异, 表明化学成分大致相同。 波数在3 385 cm-1附近与糖O— H; 纤维素N— H伸缩振动有关, 波数在2 994~2 927 cm-1范围表征炔烃类基团CH伸振动。 1 637~1 695 cm-1与烯烃类C=C伸缩振动有关。 1 079~1 059 cm-1与醚类、 酯类等含氧化合物C— O— C伸缩振动, 主要成分为蛋白质, 糖类。 846~532 cm-1为醇类、 酚类O-H伸缩振动, 表征多糖结构。 由图2(b)可知, 牛肝菌紫外光谱有效吸收峰范围在225~350 cm-1, 不同种类牛肝菌紫外光谱存在指纹差异性, 较强吸收峰在296~263 cm-1, 主要组成为蛋白质、 氨基酸类。
偏最小二乘法判别分析(partial least squares discriminantion analysis), 是一种与主成分有关的统计学方法, 将数据降维后建立回归模型并对结果进行判别分析[12]。 模型通过Y轴累积解释率(
| 表2 偏最小二乘法判别分析模型主要参数与正确率 Table 2 The main parameters and accuracy of the discriminant analysis model of partial least squares |
低级融合通过单一光谱数据模型, 选取最佳预处理(红外: 2D+MSC+SNV, 紫外: SNV+SG)数据进行合并建模。 模型选择特征值大于1的成分作为提取主成分的个数。 根据表2所示, 低级融合选取主成分19个,
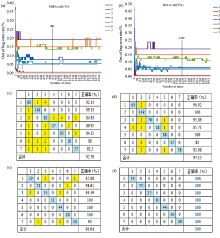
随机森林(random forest, RF)属于机器学习中集成学习方法的一种, 包含了多个Bagging集成学习技术的决策树, 通过集成学习思维将多棵决策树作为分类器的集成算法[13]。 对于一个输入样本, 将输出N个分类结果的原理, 在多个分类器输出的分类结果中筛选投票次数最多的类别作为结果。 模型建立中参数Ntree和Mtry的选择决定模型质量。 Ntree指定随机森林所包含的决策树树目, Ntree越大, 决策树棵数越多, 模型训练工作量越大。 Mtry确定了每次迭代变量抽样数值, 用于二叉树变量个数。 为避免过拟合风险增加, 模型建立分为两步, 先进行初始筛选, 计算出袋外错误率(out-of-bag error, OOB), 再进行Ntree和Mtry精确筛选, 以达到高质量模型建立的目的。 如表3所示, 在单一光谱数据矩阵中, 中红外光谱采用了三种预处理组合方法建立随机森林模型, 分别是SNV+SG, 2D+MSC+SNV, 1D+MSC+SNV+SG。 初始筛选Ntree: 2 000, 1 800和2 100, Mtry: 40, 50和42; 精确筛选Ntree: 1 412, 1 149和1 537, Mtry: 3, 5和5。 三种不同预处理得出不同的正确率, 其中2D+MSC+SNV预处理方法结果最佳, 训练集和验证集分别为93.20%和99%。 紫外光谱采用了4种预处理方法, 由表3可知, 紫外光谱模型结果正确率均低于中红外光谱, 其中1D+MSC+SNV+SG精确筛选后的Ntree和Mtry值为1 562和4, 训练集和验证集分别为62.28%与76.14%, 模型较佳。
| 表3 随机森林模型主要参数与正确率 Table 3 The main parameters and accuracy of the random forest model |
低级融合是将最佳的单一光谱预处理(红外: 2D+MSC+SNV, 紫外: 1D+MSC+SNV+SG)矩阵进行简单的数据合并, 组合成新的数据集, Kennard-Stone分类后再进行模型建立, 低级融合效果相比单一光谱, 正确率有所提高, 其中训练集和验证集分别为92.32%和99.14%。 结果表明数据融合策略用于随机森林算法具有可行性, 中级融合方法为两个单一光谱(中红外, 紫外)数据特征提取值组合矩阵。 本工作使用了两种特征值提取方法, 潜在变量和主成分提取法。 不同的特征值提取法结合OOB选择最佳决策树树目。 图3为两种特征值提取法模型的Ntree选择信息; 虽决策树选取越多越好, 但为避免过拟合风险, 在图3(a), 图3(b)预测结果中均选用平稳段Ntree(865, 1 249), 和Mtry随时抽样变量个数5; 图3(c)— (f)分别为模型正确率矩阵, 其中蓝色部分为正确数, 黄色部分为错误数, 通过矩阵可确定7类牛肝菌训练集和验证集模型的正确率。 由图3(c)和(e)可知, 中级融合(LVs)训练集和验证集正确率总个数为423与218; 由图3(d)和(f)可知, 中级融合(CPA)训练集和验证集正确率总个数为443与227。 中级融合LVs预测集与验证集为92.76%和96.04%, 中级融合CPA预测集与验证集为97.15%和100%。
研究了7种牛肝菌两种光谱谱数据融合结合偏最小二乘法判别分析和随机森林算法建立数学模型分析牛肝菌种类的准确性。 结果表明: 7种牛肝菌红外平均光谱和紫外平均光谱吸收峰大致相同, 吸光度具有细微差异, 不同种类牛肝菌化学成分相同但含量具有差异性。 偏最小二乘法判别分析和随机森林算法模型的中红外光谱数据和紫外光谱数据最佳预处理组合为2D+MSC+SNV和SNV+SG, 2D+MSC+SNV和1D+MSC+SNV+SG。 单一光谱模型结合偏最小二乘法判别分析, 单一光谱模型结合随机森林效果不佳, 中红外光谱数据模型正确率大于紫外光谱数据模型。 数据融合策略可提高牛肝菌种类鉴别模型的正确率, 随机森林、 偏最小二乘法判别分析结合数据融合分类效果明显。 其中随机森林算法中级数据融合(CPA)训练集为97.15%, 验证集为100%; 偏最小二乘法判别分析中级数据融合(CPA)训练集和验证集正确率均能达到100%, 可作为牛肝菌种类分类方法。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|