作者简介: 李 玲, 1995年生, 重庆大学化学化工学院硕士研究生 e-mail: 3257292748@qq.com;何薪宇, 1996年生, 重庆大学化学化工学院博士研究生 e-mail: 646746893@qq.com;李 玲, 何薪宇: 并列第一作者
真菌是一种广泛存在于自然界的病原微生物, 具有细胞核、 细胞壁等结构, 可以引起动、 植物和人类的多种疾病。 真菌感染是临床上常见的感染性疾病之一, 使得近年来针对真菌的高效检测及真菌相关领域的研究备受关注。 目前真菌的传统检测方法主要有培养、 镜检与分子生物学检测法等, 均具有操作复杂、 耗时等缺点。 表面增强拉曼散射(SERS)技术以其不受水分子干扰、 能反应分子指纹信息、 检测迅速等特点在真菌的检测与鉴别领域逐渐发挥出明显的优势。 在简要介绍真菌的结构特点及真菌常用的检测方法基础之上, 主要针对拉曼光谱(Raman spectrum)/SRES技术在真菌检测和鉴别中的应用进行调研和讨论。 首先通过对Raman/SERS技术的特点以及真菌的结构特征进行解析, 根据调研Raman/SERS技术用于真菌检测的相关文献, 分析了SERS技术用于真菌检测的可行性, 提出SERS技术在真菌检测时会面临检测灵敏度低、 信号复杂、 选择性和特异性差以及信号重现性和稳定性不佳等难点。 为解决以上难题, 分析了SERS的增强模式, 重点针对SERS的纳米增强介质材料、 SERS标签(SERS tag)的信号放大效应以及SERS光谱分析技术与微流控芯片分析技术结合等SERS分析新进展, 予以了系统地综述和讨论。 通过纳米材料选择和纳米微结构的构建, SERS增强介质所产生的SERS增强效应在真菌鉴别以及临床疾病快速诊断中显示出巨大的发展潜力; 基于SERS tag产生的信号放大机制, 可以有效提高真菌SERS检测的灵敏度、 特异性和重现性; 在微流控芯片中设计和集成SERS增强纳米微结构, 构建基于SERS tag 的信号放大策略, 开展针对真菌的快速高效测试方法研究, 更有望实现真菌样本的高通量及高内涵SERS检测, 其在真菌的鉴别和检测上显示出巨大的研究价值和应用前景。
LI Ling and HE Xin-yu: joint first authors
Fungi are a group of pathogenic microorganisms with nuclei and cell walls. They are widespread and cause a variety of diseases in animals, plants, and humans. Fungal infection is one of the most common clinical infectious diseases, making the efficient detection method and related research of fungi attract more attention in recent years. At present, fungi traditional detection methods mainly include culture, microscopy and molecular biology detection, which have the disadvantages of complex operation and time-consuming. Surface-enhanced Raman scattering (SERS) technology has gradually played an important role in fungal detection and identification due to its advantages such as no interference from water, providing molecular fingerprint information and rapid detection. In this paper, based on the brief introduction of the structural characteristics and the common detection methods of fungi, Raman/SERS technology in the identification and detection of fungi was investigated and discussed. Firstly, through the analysis of the characteristics of Raman/SERS and the structure of fungi, according to the related references, the feasibility of SERS technology for fungal detection was analyzed. It was found that there are some problems in the detection of fungi by SERS, such as low sensitivity, complex signal, poor selectivity and specificity, poor signal reproducibility and stability. To solve these problems, the enhancement mechanism of SERS was analyzed. In particular, the review and prospect of the new advances in SERS analysis focused on the nano-enhanced medium materials of SERS, the signal amplification effect of SERS tag and the combination of SERS spectral microfluidic chip analysis technology. The selection of nanomaterials and the construction of nanostructures showed that the SERS enhancement effect produced by the SERS enhancement substrate has great potential in fungal identification and rapid diagnosis of clinical disease. Based on the SERS tag’s signal amplification mechanism, the sensitivity, specificity and reproducibility of fungal SERS detection can be greatly improved. More importantly, the design and integration of SERS nano-enhanced substrates on microfluidic chips and the construction of signal amplification strategy based on SERS tags are more likely to achieve high-throughput and high-content SERS detection of fungal samples, which shows great research value and application prospects in the identification and detection of fungi.
真菌(fungus; eumycetes)是具有细胞核和细胞壁的异养生物, 是生物界中很大的类群, 通常分为酵母菌、 霉菌和蕈菌(大型真菌)三大类, 世界上已被描述的真菌属达1万以上, 种超过10万个。 在多数真菌的细胞壁中最具特征性的是甲壳质(chitin), 其次是纤维素。 常见的真菌细胞器有: 线粒体, 微体, 核糖体, 液泡, 溶酶体, 泡囊, 内质网, 微管, 鞭毛等, 常见的内含物还有肝糖, 晶体, 脂体等。 近年来, 随着广谱抗生素、 糖皮质激素、 免疫抑制剂的广泛使用, 以及各种介入性医疗技术的使用, 临床上真菌感染的数量及种类逐年增加, 已成为医院内感染的主要原因之一。 例如, 临床分析发现绝大多数医院临床真菌感染的源头是念珠菌, 其中白色念珠菌占全部感染的60%左右, 导致死亡率接近40%[1], 这种感染甚至能引起器官移植患者、 癌症患者和艾滋病患者的死亡。 因此, 高效辨识和监测真菌的方法和技术备受关注。
传统的真菌检测方法是培养与镜检, 培养是真菌学检查的“ 金标准” , 但其具有耗时长、 易污染、 灵敏度低、 无法区分定植菌及感染菌等缺点[2]; 直接镜检更简便、 快速, 但其准确率普遍不高, 难以为真菌早期诊断提供准确判断。 近年来, 分子生物学检测法是目前国内外研究发展热点, 具有特异性强、 灵敏度高的特点[3], 具体包括: 聚合酶链式反应法、 G试验法和GM试验法(galactomannan, GM test)等, 但这类方法成本较高, 操作复杂且未标准化, 临床上易产生假阳性结果。 随着现代仪器科学与技术的发展, 基质辅助激光解析电离飞行时间质谱(matrix-assisted laser desorption/ionization time of flight mass spectrometry, MALDI-TOF-MS)[4]和环介导等温扩增(loop-mediated isothermal amplification, LAM)等技术开始应用于真菌鉴定和检测, 但其对于检验人员要求较高, 涉及的实验仪器也比较昂贵, 在临床快速诊断中的应用受到限制。 因此, 研发并建立真菌快速、 高效的检测方法、 技术和系统具有重要的研究意义和迫切的应用需求。 基于前期研究和调研, 我们发现拉曼(Raman)光谱尤其是表面增强拉曼散射(surface-enhanced Raman scattering, SERS)光谱分析技术在真菌鉴别和检测领域大有可为。
据此, 本文以临床诊断和环境空间中真菌为检测对象, 重点对Raman/SERS在真菌鉴别和检测中的应用进行探讨, 尤其针对纳米SERS增强介质材料、 SERS标签(SERS tag)信号放大效应以及集成SERS的微流控芯片分析方法等研究新进展进行综述, 分析探讨Raman /SERS应用于真菌检测的优势及其发展趋势。
Raman光谱分析技术因其具有能提供分子基团结构信息、 无需对样品进行前处理、 无水分干扰、 易于实现原位实时检测等优点, 特别适用于生物样本及生化体系的检测。 早在1995年, Edwards等[5]报道了利用拉曼光谱技术对三种不同属的真菌样本进行检测, 并对拉曼谱峰进行了归属指认。 10年后, Gussem等[6]确认收集到的拉曼信号主要来源于真菌细胞壁上的多糖、 几丁质和支链淀粉以及细胞膜上的脂质和磷脂。 但是, 由于Raman光谱是基于散射光信号建立的光谱分析技术, 散射信号强度弱且重现性较差, 大大限制了其在复杂生命体中的应用。
随着材料学、 光学、 仪器科学等相关领域的迅猛发展, 通过引入纳米技术而建立的SERS光谱分析技术, 利用粗糙的金属表面等离子体, 在纳米级间隙(通常是1 nm)的区域内产生热点, 极大地增强待测物分子的Raman信号, 可实现目标物的更高效检测。 基于SERS光谱分析理论研究的不断深入, 人们发现其除具备Raman光谱分析所具有的基本特性外, 更在检测灵敏度、 抗光漂白、 易于实现原位无损检测等方面显示出强大优势。 据此, SERS光谱分析技术被广泛用于多种生化样本的检测, 如: 磷脂、 葡萄糖、 谷胱甘肽、 凝血酶等[7], 近年来更是被拓展到了细菌和细胞等生命体中[8], 也使得真菌的高效SERS检测和临床诊断成为可能。 本课题组前期已开展了将SERS光谱技术应用于生物分子、 细菌和细胞及其相关生化过程研究[9, 10, 11]。 Gussem[6]等应用Raman光谱分析乳杆菌(Lactarius)的真菌孢子, 解析其组成成分, 将得到的真菌光谱与已知存在于大型真菌中的参考物质的拉曼光谱进行比较, 包括糖类, 脂质和一些可用作特异性生物标记物(腺嘌呤, 麦角甾醇和甘氨酸)的次要化合物。
与传统生化小分子Raman/SERS检测大量的文献报道相比, 以真菌为样本的SERS检测研究报道相对较少。 这是由于对于真菌这种真核细胞生命体, 其成分复杂且本身具有一定的不均一性, 检测特性与细菌和细胞的检测有一定的相似性, 面临检测灵敏度低、 信号复杂、 选择性和特异性差以及信号重现性和稳定性不佳等难题。 尽管如此, 当针对真菌SERS检测中的这些难点有所突破时, 就有理由相信SERS对真菌的检测和相关研究是可行的, 并具有极大地发展潜力。
SERS的增强模式主要有电磁场增强以及化学增强[12], 其是依托金属、 半导体及多种复合纳米微结构来实现的。 这些活性纳米基底的结构和效能对于提高SERS检测灵敏度、 重复性尤为重要。 最常应用于SERS检测的是金纳米粒子(Au NPs)和银纳米粒子(Ag NPs), 通过更改金属纳米粒子的形貌, 制备成纳米棒[13]、 纳米簇[14]、 纳米星[15]等, 可形成热点效应, 显著增强待测物Raman信号。 采用单一金属纳米材料其增强效能往往也不能达到测试的需求, 于是, 过渡金属、 TiO2、 CuO、 ZnO等半导体材料以及复合纳米材料进入人们视线[16], 目前, 多种复合纳米结构更是显示出优异的SERS效能, 如壳核纳米颗粒结构、 二维Au纳米伞结构、 多面体Ag颗粒和原子层沉积纳米颗粒等, 更有金属纳米颗粒与磁性半导体材料、 有机聚合物等多种纳米材料复合[17], 这些新型的纳米微结构具有更好的生物兼容性、 更强的吸附能力和更强的增强效果, 可实现更高效的SERS检测[18]。 近年来, 随着纳米材料及其制备技术的迅猛发展, 结构有序可控的SERS基底纳米微结构设计与制备日趋成熟, 许多合成制备技术, 微机电系统(MEMS, Micro-Electro-Mechanical System)加工工艺等能够用于制备具有良好重复性能的SERS基底[19], 如化学还原法、 磁控溅射、 电子束光刻技术和自组装等。
基于多种类型的纳米微结构SERS基底的发展和应用, 近年来人们开始将SERS光谱技术应用于病毒、 真菌、 细菌和细胞及其相关生化过程的监测和研究。 Sivanesan等[20]在粗糙化的纳米银基板上通过恒电位电沉积薄金层, 获得银-金双金属复合SERS基底, 涂覆抗生素后, 从血液样本中选择性鉴定出大肠杆菌、 肠炎链球菌和表皮葡萄球菌。 Luo等[21]通过疏水相互作用将油溶性Ag NPs与多孔碳膜自组装获得SERS基底, 结合主成分分析(principal component analysis, PCA), 有效区分了猪圆环病毒2型(PCV2)、 猪细小病毒(PPV)和猪伪狂犬病病毒(PRV)。 Witkowska等[22]以纳米Ag作为SERS增强基底, 测得这三种不同属真菌: 毛癣菌, 小孢子菌和表皮癣菌的SERS光谱, 利用主成分分析, 实现了对常见皮肤真菌毛癣菌不同亚种的有效鉴别, 使同属不同亚种真菌的鉴别成为可能。 Dina等[23]采用化学还原法合成Ag NPs, 辅以主成分分析法及线性判别法(principal component analysis-linear discriminant analysis, PCA-LDA), 成功用SERS技术对烟曲霉、 隐孢曲霉和粉状根霉三种真菌进行了鉴别(图1), 整个过程耗时仅5分钟, 大大缩短了侵袭性真菌感染的确诊时间。 Prusinkiewicz等[24]通过Au NPs与构巢曲霉共孵育后, 采用SERS光谱对真菌细胞内外进行检测, 利用获取的SERS图谱还开展了真菌细胞环境分析。 Tripathi等[25]通过拉曼光谱对水悬浮液中不同浓度的大肠杆菌和枯草芽孢杆菌孢子的混合物进行研究和区分。 王科兵等[26]基于纳米银胶建立了SERS法快速判别失活白色念珠菌的方法, 白色念珠菌灭活后的SERS光谱与失活前的光谱有明显差异。
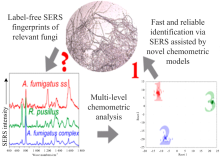 | 图1 烟曲霉、 隐孢曲霉和粉状根霉三种真菌的SERS光谱及主成分分析中PC1和PC2得分[23]Fig.1 SERS spectra of aspergillus fumigata, aspergillus cryptosporus and rhizopus powdery combined with principal component analysis[23] |
可以看到, 融合纳米技术的SERS光谱检测技术在真菌类菌种的鉴别以及临床疾病的快速诊断方面显示出非常好的发展潜力和应用前景。 但目前SERS在真菌检测中的研究和应用相对较少, 其面临检测灵敏度不高、 信号稳定性不佳、 信号识别有难度等局限和不足。 研发更为高效的纳米SERS基底和纳米微结构, 改进检测模式, 可以使SERS在真菌检测中发挥更大的优势。
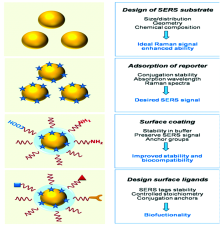
将SERS技术应用于真菌检测时, 面临的核心问题就是灵敏度、 重现性和特异性。 真菌样本自身的特性, 制约了SERS光谱对其进行高效定量测试。 为了解决这个问题, 人们提出采用SERS标签(SERS tag)的检测策略和方法。 SERS tag的设计思想主要包括: SERS活性金属纳米粒子制备, SERS报告分子连接, 表面修饰, 探针分子(抗体、 适配体等)结合, 由此实现目标组分的信号放大和高效检测(图2)[27]。 SERS tag由金属纳米基底材料与能够提供特征性SERS指纹图谱的信号分子组成, 目前常用的信号分子包括: 4-巯基苯甲酸(MBA)、 苯-4, 4’ -二硫醇(DBDT)、 内消旋-四(4-羧基苯基)卟啉(TCPP)、 Ru(bpy)(三(2, 2’ -联吡啶基)氯化钌(Ⅱ ))、 罗丹明6G(R6G)、 孔雀石绿异硫氰酸酯(MGITC)等[28]。 近年来, 多种新型SERS tag被设计和制备出来, 成功应用于生物分子、 病原菌、 细胞等的特异性检测, 甚至被用于体内组织或者器官的活体成像, 其在提高SERS检测灵敏度的同时, 更是对特定样本实现了高选择性甚至是特异性检测。
Zhao等[29]制备了一种由AgNPs和5, 5’ -二硫双(2-硝基苯甲酸)(DTNB)报告分子组成的SERS tag, 与磁性纳米颗粒通过抗体抗原反应进行偶联后, 用于未经处理的全血样本中基质金属蛋白酶9(MMP-9)的免疫检测, 检测限为1 pg· mL-1[图3(A)]。 Bamrungsap等[30]采用层层组装的方法在金银纳米棒(Au-Ag NRs)表面组装了拉曼报告分子4-氨基硫酚和荧光标记的适配子, 以此合成双功能SERS tag, 可实现宫颈癌细胞上表达的人蛋白酪氨酸激酶-7(PTK-7)的特异性靶向与荧光成像。 Madiyar等[31]利用氧化铁-金(IO-Au)核壳纳米卵形颗粒(NOV)包被QSY21拉曼报告分子制成SERS tag, 并通过特异性免疫化学连接到大肠杆菌菌株DHα 5上, SERS tag的拉曼信号增强效果与介电泳富集效果相结合, 捕获时间仅为50 s, 检测下限可达210 cfu· mL-1[图3(B)]。
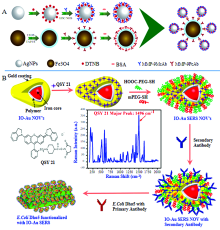 | 图3 不同SERS tag的生化检测策略示例[29, 31]Fig.3 Examples of biochemical detection strategies based on different SERS tags[29, 31] |
Mabbott等[32]以单链硫代DNA修饰的银羟胺纳米粒子作为SERS tag, 结合主成分分析法, 成功对白色念珠菌、 光滑念珠菌、 克鲁斯念珠菌和烟曲霉进行鉴别和归类(图4)。 Yuan等[33]利用抗菌肽(AMP)功能化磁性纳米颗粒作为细菌分离探针, 4-巯基苯基硼酸(4-MPBA)修饰的镀金银氧化石墨烯(Au@Ag-GO)纳米复合材料作为SERS tag, 分离并检测了大肠杆菌、 金黄色葡萄球菌和铜绿假单胞菌, 每种细菌的最低检测浓度仅为10 cfu· mL-1。 Zhang等[34]利用万古霉素修饰Fe3O4@Au磁性纳米颗粒, 开展细菌的广谱识别和高效富集, 使用适配体功能化的SERS tag, 对金黄色葡萄球菌和大肠杆菌的检测限分别达到20和50 cells· mL-1。 Pang等[35]采用适配体-Fe3O4@Au磁性纳米颗粒作为磁性底物和SERS激活底物, 基于金壳的双SERS增强和适配体/万古霉素的双重识别能力, 用于目标细菌的富集和定量检测, 可以在没有其他非靶标细菌干扰的情况下, 在50 min内对金黄色葡萄球菌的检测达到3 cells· mL-1的检测限。 这些研究显示, SERS tag所带来的高特异性和高灵敏度在临床应用方面极具前景。
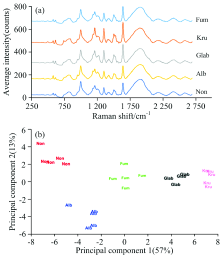 | 图4 肉眼难以分辨的不同SERS tag与不同真菌结合后的SERS光谱(a)及利用PCA对不同的SERS谱图进行识别(b)[32]Fig.4 Shows the spectral signal arising from a multiplex, consisting of all the fungal probes together with the individual targets (a); Discrimination of these spectra is difficult by eye; Discrimination of the multiplexed targets is possible when PCA is applied to the dataset (b)[32] |
为了进一步提高SERS tag在检测中的稳定性, 人们发展并研制出核-壳纳米粒子结构的SERS tag。 He等[36]将拉曼信号分子3, 3’ -二乙基噻三碳菁碘(DTTC)嵌入共轭金银纳米壳, 对耐甲氧西林金黄色葡萄球菌(MRSA)检测灵敏度可降至300 cfu· mL-1。 Ye等[37]采用种子介导法和分步纳米加工法, 制备了双金属双壳体(Au@BDT@Au@BDT@Ag), 极大提高了纳米粒子的SERS性能, 并建立了新的表面增强拉曼散射的侧向流动免疫分析方法, 对人绒毛膜促性腺激素(Human chorionic gonadotrophin, hCG)的检测限低至0.7 mIU· mL-1(0.077 ng· mL-1)。
另外, SERS tag在拉曼成像方面也显示出独特的优势。 Aberasturi等[38]设计并制备了不同的十二胺改性的聚异丁烯-α -马来酸酐(PMA)包覆金纳米星的SERS tag, 分别以不同的小分子芳香族硫醇作为报告分子, 来自五个不同细胞系的单个乳腺癌细胞可以在共同培养中被检测、 区分和成像, 且细胞培养的SERS信号和图像在24 h以上保持稳定。 Bohndiek等[39]设计制备了4种修饰了不同报告分子的SERS tag, 经裸鼠皮下注射后, 利用开发的小动物拉曼成像(small animal Raman imaging, SARI)仪, 可实现在较大区域(> 6 cm2)上以高空间分辨率的快速成像, 如图5所示, 观测到SERS tag在裸鼠肝脏的蓄积, 实现了活体内的器官成像。
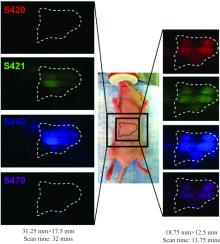 | 图5 活体SERS成像: 注射四种SERS tag (S420, S421, S440, S470)后小鼠肝脏的SERS成像图[39] 注射后1 h(左); 注射后2 h(右)Fig.5 In vivo SERS imaging: Map of mouse liver after injection of four SERS tags (S420, S421, S440, S470)[39] 1 hour after injection (left) and 2 hours after injection (right) |
可以看到, 基于SERS tag设计与制备技术的快速发展, 新型SERS tag在满足特异性检测目标分析物的同时, 可进一步提高了检测的灵敏度与重现性。 SERS tag在细胞和细菌检测中显示出成效, 也有少量针对真菌的研究, 而将SERS tag用于真菌的高效定量检测必将受到关注和发展。
微流控芯片分析技术不仅可将样品预处理、 分离、 检测等多个基本操作单元集成, 而且可与多种检测技术进行结合, 实现生化样本的高通量及高内涵检测, 在细菌、 真菌和细胞等研究中展现出突出的优势和潜力。 在微流控芯片上集成SERS分析模块, 可将含真菌的生化样本引入到微流控芯片的设定集成纳米微结构SERS检测微区, 直接进行原位检测并获取其生化信息, 为生化体系的高效检测提供了新机遇和新途径。
众所周知, 细菌、 真菌和细胞类生化样本在检测中通常存在基底复杂、 背景信号高、 水分干扰极大、 样本暴露于空气而受到环境中各种因素的影响等难题, 而集成SERS 基底的微流控芯片系统及相应的测试方法, 对上述难题的有效解决显示出明显的优势[40]。 首先, 由于拉曼激光斑点小, 能够在微流控芯片的通道上直接聚焦测试; 微流控芯片通道内反应试剂较少, 符合拉曼灵敏度高的条件; 由于反应试剂没有直接接触, 对反应体系不造成干扰; 同时, 由于SERS光谱具有特征指纹性, 能够对反应体系中的混合物进行分析和识别。 因此, 集成SERS增强基底的微流控芯片(简称: 微流控SERS芯片)可提供一个生化样本高效测试环境空间, 有效提高检测的灵敏度、 重复性和可靠性; 同时, 将纳米技术与微流控技术融合, 将推动微流控芯片的功能拓展, 在临床诊断、 生物学等领域具有广阔的应用前景。
Li等[41]研制的适体-表面增强拉曼散射(Aptamer-SERS)传感芯片, 可以预先完成芯片功能化、 SERS标签制备、 适体互补DNA杂交等复杂操作, 成功实现了真菌毒素— 黄曲霉毒素B1(AFB1)的检测, 线性范围为1 fg· mL-1~1 ng· mL-1, 检出限可达0.4 fg· mL-1[图6(a)]。 Wang[42]等设计的CD式样微流控芯片, 通过使用具有径向微流体通道的聚二甲基硅氧烷(PDMS)聚合物板将寡核苷酸探针线阵列“ 印刷” 在玻璃芯片上, 通过在螺旋通道内进行样品杂交, 实现对浓度低至3 ng· mL-1的真菌PCR产物的检测。 杨宁等[43]根据微尺度下孢子富集动力学特征设计了病害孢子高效富集微流控芯片, 结合光电检测系统, 进行了水稻的稻曲病菌孢子的检测。 Wang等[44]通过在微流控SERS芯片[图6(b)]上同时部署针对同一病原菌不同表位的3个不同SERS探针, 实现了单细胞水平上对病原菌的不同亚种特异性检测。 Yang等[45]研制的便携式带正电荷的SERS芯片, 成功用于直接从培养基中捕获并鉴定大肠杆菌CFT 073、 铜绿假单胞菌PAO1和奇异变形杆菌PRM1等三种泌尿系统病原菌。 本课题组Su等[11]设计了集成血液分离、 试剂混合、 SERS检测一体化的微流控芯片, 在检测区原位集成Ag膜-纳米金SERS基底[图6(c)], 针对水中肌酐的检出限低至4.42× 10-3 μ mol· mL-1, 且可在2 min内完成血液样本中肌酐的测试。 Wang等[46]采用自组装-化学镀复合方法, 在微通道内的TiO2纳米管的孔口表面和内壁上覆盖Au@Ag纳米颗粒作为SERS基底, 对R6G的检测限低至10-10 mol· L-1, 利用Au@Ag/TiO2纳米管的光催化性能, 制备了一种新型可回收微流控SERS芯片, 可用作多种生化分子的高效SERS检测器。
 | 图6 基于不同微流控SERS芯片的SERS检测示例及得到的SERS光谱图[41, 44, 11]Fig.6 Examples of SERS detection based on different microfluidic SERS chips and the obtained spectra[41, 44, 11] |
显然, 将多种纳米结构在微流控芯片上集成制备是可行的, SERS光谱采集对试剂和样本是非接触式的, 对生化体系和过程不会造成干扰, 拉曼激光斑点小能够在微流控芯片的通道上直接聚焦, 同时SERS光谱具有的指纹性能够获得更多的生化信息。 据此, 我们认为结合微流控技术, 有望解决并突破目前SERS分析技术存在检测效率低、 信号稳定性差、 应用范围窄等瓶颈问题, 对真菌检测提供更高精度、 更可靠测试手段。
针对近年来真菌感染带来的对真菌高效检测的需求, SERS光谱分析以其信息量丰富、 无标、 无损、 原位检测的优势, 在真菌的高效检测与鉴别方面展示出良好的研究意义和应用潜力。 真菌作为一种复杂的生命体, 在其SERS检测中涉及到许多物质、 能量以及信号的转换, 其Raman/SERS效应也相对复杂, 在该领域仍然存在一些问题和挑战。 提高检测灵敏度、 重现性和选择性是实现有效鉴别和测试的关键, 通过设计与制备结构可控、 增强效果佳的SERS活性增强基底, 研制特异性识别真菌的SERS tag, 在提高检测灵敏度和选择性的同时, 借助微流控芯片自身优势, 改善SERS检测的环境, 提升检测的重现性和稳定性, 有望实现对复杂样本中真菌的快速鉴别与检测。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|