作者简介: 张娇, 女, 1994年生, 云南中医药大学中药学院硕士研究生 e-mail: jzhang2019@126.com
黄精药材品质优劣与基原植物产地环境因子密切相关, 建立简单、 快速且能够准确鉴别药材产地的方法对保证其质量可控及用药安全具有重要的理论意义和应用前景。 研究中以云南、 四川和广西9个产地的133份滇黄精 Polygonatum kingianum coll. et Hemsl根茎为试验材料, 采集衰减全反射-傅里叶变换红外光谱(ATR-FTIR)和紫外-可见光光谱(UV-Vis)数据预处理后分别建立单一光谱随机森林(Random forest, RF)模型; 将ATR-FTIR与UV-Vis数据直接串联完成低级融合, 提取两种光谱的主成分数(PCs)和潜在变量(LVs)以实现中级(中级融合PCs和中级融合LVs)和高级数据融合(高级融合PCs和高级融合LVs), 基于不同数据融合策略分别建立RF模型; 比较不同模型的正确率(ACC)、 灵敏度(SEN)和特异性(SPE), 筛选产地鉴别最佳模型。 结果显示, 不同产地滇黄精ATR-FTIR和UV-Vis峰型相似, 吸光度略有差异, ATR-FTIR显示14个共有峰, 与糖类、 甾体皂苷、 黄酮类和生物碱类物质有关, 其UV-Vis共有峰主要位于272及327 nm处, 与黄酮类物质有关; ATR-FTIR、 UV-Vis和低级融合的RF模型, 训练集和预测集ACC分别为(76.34%, 95.00%), (80.65%, 95.00%)和(83.87%, 100.00%), 但SEN和SPE值较低, 故不宜采用; 中级融合PCs和中级融合LVs的RF模型的SEN和SPE分别为大于0.91和0.98, 训练集ACC分别为91.40%和97.85%, 预测集ACC均为97.50%; 高级融合PCs和高级融合LVs的RF训练集ACC分别为77.42%和97.85%, 预测集ACC均为95.00%, 高级融合PCs的RF模型鉴别效果较差, 高级融合LVs的RF模型存在过拟合现象; 模型鉴别能力为中级融合LVs>中级融合PCs>低级融合> UV-Vis>ATR-FTIR>高级融合PCs; 提取LVs对产地鉴别的方法优于PCs; 中级融合LVs建立的RF模型鉴别ACC最高, SEN和SPE大于0.98, 模型性能最佳。 该方法可为黄精药用资源的科学评价提供理论依据和技术支撑。
The quality of Polygonati Rhizoma medicinal materials is closely related to the original plants’ origin environment. It is necessary to ensure their quality control and drug safety by establishing a simple, rapid and accurate origin identification method for the medicinal materials.In this study, the attenuated total Reflection-Fourier transform infrared (ATR-FTIR) spectra and ultraviolet visible (UV-Vis) spectra of 133 Polygonatum kingianum rhizomes from 9 geographic origins in Yunnan, Sichuan and Guangxi Provinces were collected to establish random forest (RF) modelafter data pretreatment, respectively. ATR-FTIR and UV-Vis spectra data were directly connected in series to complete the RF model of low-level data fusion. Principal components (PCs) and latent variables (LVs) of the two spectra were extracted to achieve RF model ofmid-level (mid-PCs and mid-LVs) and high-level (high-PCs and high-LVs) data fusion. The accuracy (ACC), sensitivity (SEN) and specificity (SPE) of different models were compared to select the best model for origin identification. The results showed that the peaks of ATR-FTIR and UV-Visspectrain P. kingianum were similar, and their absorbance were different. There were 14 common peaks in ATR-FTIR spectra of P. kingianum, which were related to carbohydrate, steroidal saponins, flavonoids and alkaloids. The common peaks of UV-Visspectra in P. kingianum were mainly at 272 and 327 nm, which were related to flavonoids. For the RF models of ATR-FTIR, UV-Vis and low-level fusion, the ACC of the training set and prediction set were respectively (76.34%, 95.00%), (80.65%, 95.00%) and (83.87%, 100.00%), however, the SEN and SPE values were so low that they were not suitable to use. The SEN and SPE of mid-PCs and mid-LVs RF models were greater than 0.91 and 0.98, respectively. The ACC of the training set was 91.40% and 97.85%, respectively, and that of the prediction set both were 97.50%. The ACC of RF training set with high-PCs and high-LVs was 77.42% and 97.85%, respectively, and the prediction set ACC both were 95.00%. The RF model with high-PCs has poor identification effect, and the RF model with high-LVs was over-fitted. In summary, the identification of model from high to low was: mid-LVs>mid-PCs>low fusion>UV-Vis>ATR-FTIR>high-PCs. LVs extraction method is better than PCs for origin identification. RF model of mid-LVs established has the highest ACC with the best model performance, and the SEN and SPE greater than 0.98, and, which can provide a theoretical basis for the scientific evaluation of medicinal resources of Polygonati Rhizoma.
滇黄精Polygonatum kingianum coll. et Hemsl是百合科(Liliaceae)黄精属药食同源植物, 主要分布于我国云南、 贵州、 四川等西南地区和越南、 缅甸、 日本等国家[1]。 其干燥根茎具有补气养阴、 健脾、 润肺和益肾之功效, 是中药黄精的主要来源之一[2]。 滇黄精主要药效成分是多糖和甾体皂苷, 此外还含有黄酮、 生物碱、 氨基酸等成分, 现代药理学研究表明其具有降血糖、 抗衰老、 抗肿瘤等作用[3]。 调查发现, 滇黄精栽培范围逐年扩大, 不同产地的气候、 土壤条件等均影响其药材质量。 李婧等[4]以4种黄酮类成分含量为指标筛选影响其含量的环境因子, 结果表明降水量、 年平均温度、 黏土量等环境因子对黄酮类成分影响最大。 研究发现不同产地黄精中的多糖、 薯蓣皂苷元[5]和挥发性成分[6]等均存在显著差异。 为保证黄精质量的有效性和一致性, 产地鉴别研究是其中的关键环节和重要前提条件。 目前, 可进行准确定量的色谱技术、 液(气)质联用技术及电化学指纹图谱技术广泛应用于其产地溯源研究[7], 但这些技术存在操作复杂、 成本高和耗时长等缺点, 因此找到一种快速、 简便且可靠的方法显得尤为重要。
衰减全反射-傅里叶变换红外-光谱(attenuated total reflection-Fourier transform infrared spectra, ATR-FTIR)和紫外-可见光光谱(ultraviolet-visible spectra, UV-Vis)技术具有方便、 快速、 无损等特点, 已被广泛应用于食品与中药的产地鉴别。 Zhao等[8]利用FTIR技术和化学计量学方法鉴别滇龙胆产地, 结果显示正确率达到97.22%, 为滇黄精产地鉴别提供参考。 但单一指纹图谱通常不能全面反映样品化学信息, 采用数据融合策略能够弥补此方面的不足。 Yao等[9]采用FTIR和UV-Vis技术对7个产地牛肝菌进行鉴别, 训练集和预测集正确率(accuracy, ACC)分别为80.18%和94.14%, 使用中级数据融合策略后达到99%。 Wu等[10]采集ATR-FTIR和UV-Vis信息结合高级数据融合策略鉴别6个产地野生滇重楼, 分类正确率为98.88%。 由以上研究结果可知, 数据融合策略可有效提高产地鉴别正确率, 能够实现中药产地的快速、 方便和无损鉴别。
本研究拟采集9个产地共133份滇黄精根茎样品的ATR-FTIR和UV-Vis光谱信息, 经预处理及特征变量筛选后, 建立单一(ATR-FTIR, UV-Vis)和数据融合(低级、 中级和高级)随机森林(random forest, RF)模型, 通过比较其灵敏度、 特异性和分类正确率参数, 最终确定快速鉴别滇黄精产地的最佳模型和方法, 为其药用资源评价提供理论依据。
采集于云南、 四川和广西9个产地的133份样品, 由云南省农业科学院药用植物研究所张金渝研究员鉴定为滇黄精Polygonatum kingianum coll. et Hemsl的根茎[图1(a, b, c)和表1]。 样品去除须根, 用去离子水将附着的杂质和泥土清洗干净, 切片, 于55 ℃烘箱中干燥至恒重。 粉碎过筛(100目)后保存于自封袋中备用。
 | 图1 滇黄精样品和采集地图片 (a): 全株植物; (b): 根茎; (c): 样品采集地Fig.1 Collectionorigins of P. kingianum samples (a): The whole plant; (b): Rhizome; (c): Samples collection area |
| 表1 滇黄精样品信息 Table 1 Information of P. kingianum samples |
1.2.1 ATR-FTIR采集
ATR-FTIR光谱通过配备ZnSe衰减全反射附件及氘化硫酸三甘氨酸(DTGS)检测器的FTIR光谱仪(frontier perkin elmer, USA)采集。 扫描范围为4 000~550 cm-1, 扫描信号累加16次, 分辨率为4 cm-1。 每个样品重复3次, 取平均光谱。
1.2.2 UV-Vis采集
UV-Vis光谱通过配有积分球检测器的UV2700紫外-可见分光光度计(Shimadzu, Japan)采集。 使用石英容器压片, 压制成1 mm薄片进行光谱采集。 样品测试前使用BaSO4进行背景扫描。 扫描范围220~850 nm, 采样间隔为1, 狭缝宽度为5.0 nm。 每个样品重复3次, 取平均光谱。
RF是以决策树为基础的有监督学习算法[11]。 建模前使用Kennard-Stone算法[12]将每个产地样品的2/3划分为为训练集, 1/3划分为预测集。 采用训练集样品建立模型, 预测集样品用来验证模型的性能。 建模时从原始训练集中使用自助法随机且有放回地取出m个样品, 共进行n次取样, 得到n个训练集并对每一个训练集训练, 根据袋外-误差率(out-of-bag error, OOB)最小来选择最优的ntree棵决策树(classification and regression tree, CART)。 在CART分类过程中没有进行剪枝处理。 每个样品有M个变量, 随机变量数(mtry)决定每棵树的分类性能, 在建模过程中使用± 10来寻找最优mtry。 最后根据找到的最优参数ntree和mtry建立最终鉴别模型。 通过集成多个CART的分类结果进行投票获得最后的分类结果, 即使数据分布不平衡或有多个缺失值, 也能提供稳定、 准确度高的分类模型[13]。 采用灵敏度(sensitivity, SEN)、 特异性(specificity, SPE)和正确率ACC来衡量模型是否稳定。 ACC, SEN和SPE值越接近于1, 模型的性能越好。 计算公式见式(1)和式(2)
数据融合属于化学计量学方法之一, 是将不同来源数据有效结合后再建立分类模型的一种策略[13], 通常分为低级、 中级和高级融合。 低级融合是指将不同来源的数据合并成一个新数据矩阵再建立分类模型。 中级融合是分别提取单一光谱、 色谱或者波谱的特征变量串联形成一个新数据矩阵来建立分类模型。 主成分数(principal components, PCs)、 潜在变量(latent variables, LVs)、 变量投影重要性等, 是数据融合中常用的特征变量提取方法。 高级融合是在中级融合的基础上, 用特征变量分别建立单一模型, 融合单一模型结果, 根据模糊集合论的最大值(maximum, Max)、 最小值(minimum, Min)、 乘积(product, Pro)和平均值(average, Ave)进行投票得到最终结果。 为了使数据处理方便在数据融合前进行归一化处理。
通过OMNIC 9软件将ATR-FTIR透光率转化为吸光度。 使用SIMCA 14.1软件对光谱数据进行预处理。 用ORIGIN 9.1软件作图。 R studio软件用于建立RF模型。 光谱易受噪音和样品性质的影响, 对其进行适当的预处理是必要的。 采用一阶导数(first derivative, FD)、 二阶导数(second derivative, SD)和标准正态变量(standard normal variable, SNV)对光谱进行预处理, 根据决定系数(determination coefficient, R2)、 交叉验证均方根误差(root mean square error of cross validation, RMSEcv)和校正均方根误差(root mean square error of estimation, RMSEE)及ACC来选择最佳预处理方式。 UV-Vis波长在700~850 nm范围内受噪音影响较大, 在建立RF模型时去除这一波段。 ATR-FTIR在建模分析时去除4 000~3 700 cm-1的基线区、 682~653 cm-1的CO2光谱区和2 500~1 799 cm-1的ZnSe晶体光谱区[14]。 当Q2(模型预测能力)第一次达到最大值时, 提取相应特征变量[15]。 从ATR-FTIR的133× 1 775个变量中分别提取133× 17个PCs和133× 13个LVs, 从UV-Vis的133× 467个变量中分别提取133× 9个PCs和133× 5个LVs用于建立模型。
2.1.1 ATR-FTIR分析
图2(a)是9个产地滇黄精原始ATR-FTIR平均图。 不同产地的ATR-FTIR有14个共有峰, 波数分别为3 338, 2 924, 2 845, 1 735, 1 625, 1 416, 1 362, 1 316, 1 262, 1 115, 1 024, 928, 873和824 cm-1。 共有峰的峰形和峰位相似, 其差异较大的是吸光度值。 云南镇雄县和四川会东县两个产地样品的吸光度明显高于其他7个产地。 3 338 cm-1表征— OH或者— NH— 的伸缩振动; 2 924和2 825 cm-1由吡喃糖环中C— H的伸缩振动引起; 1 735 cm-1表征C=O的伸缩振动; 1 625 cm-1表征C=C和C=N的伸缩振动, 与黄酮类、 甾体皂苷和生物碱有关; 1 416, 1 362和1 316 cm-1表征C— H或者— OH的弯曲振动; 1 262, 1 115和1 024 cm-1表征C— O的伸缩振动; 928, 873和824 cm-1表征=CH的弯曲振动[16, 17]。
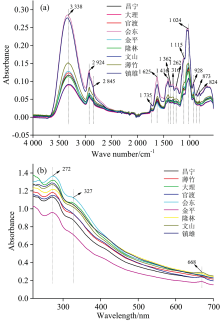 | 图2 9个产地滇黄精的平均光谱图 (a): ATR-FTIR; (b): UV-VisFig.2 Average spectra of 9 origins in P. kingianum (a): ATR-FTIR; (b): UV-Vis |
2.1.2 UV-Vis分析
图2(b)为9个产地滇黄精原始UV-Vis平均图。 其特征峰波长为272和327 nm, 推测与滇黄精中黄酮类物质有关[18]。 部分样品在668 nm处的可见光区存在吸收峰。 整体而言, UV-Vis吸收峰较少, 主要反映芳香族和含有共轭体系的黄酮类物质信息。 此外, 产自大理的滇黄精UV-Vis吸光度次之, 与ATR-FTIR显示产自镇雄的滇黄精吸光度结果不一致, 因此使用不同性质和原理的指纹图谱来评价或鉴别滇黄精产地是必要的。
两种光谱经FD, SD, SNV预处理(表2), UV-Vis在预处理(除SD)后建模不成功, SD为两种光谱的最佳预处理方式, 鉴别能力较差, 选择非线性的RF算法对滇黄精产地进行鉴别分析。 ATR-FTIR的RF模型最优ntree为1 140, mtry为35。 UV-Vis的RF模型最优ntree为1 041, mtry为42。 结果如表3所示, ATR-FTIR光谱结果显示训练集ACC=76.34%, 预测集ACC=95.00%, 训练集的SEN为0.77(< 0.8), 训练模型时对样品识别能力较差, 模型存在不稳健现象; UV-Vis的RF模型SEN和SPC值分别为0.8和0.98, 训练集ACC=80.65%, 预测集ACC=95.00%, 对产地鉴别效果较差。 采用数据融合策略建立RF模型对这9个产地的滇黄精进行鉴别。
| 表2 单一光谱预处理结果 Table 2 Single spectral pretreatment results |
| 表3 数据融合的结果 Table 3 The results of data fusion |
2.3.1 低级融合
将ATR-FTIR的133× 1775个变量和UV-Vis的133× 467个变量串联起来形成一个新的数据矩阵建立RF模型, 其最优ntree和mtry值如图3(a)所示。 模型的灵敏度和特异性大于0.83, 训练集和预测集的正确率(表3)分别为83.87%和100.00%, 训练模型时对样品的识别能力较弱, 表明光谱含有一些对产地鉴别冗余的波段, 需要挖掘对产地鉴别有用的信息。
2.3.2 中级融合
在中级融合中, 使用PCs和LVs来建立RF产地鉴别模型, 比较两种特征变量融合对产地鉴别的能力。 提取特征变量结果如图4所示, ATR-FTIR的133× 17个PCs和UV-Vis的133× 9个PCs被提取建立RF模型, ATR-FTIR的133× 13个LVs和UV-Vis的133× 5个LVs提取建立RF模型。 结果如表3所示, LVs建立中级融合(中级融合LVs)的RF模型中3个样品被分类错误; PCs建立中级融合(中级融合PCs)的RF模型中8个样品被分类错误。 基于LVs建立的中级融合RF模型训练集和预测集的灵敏度、 特异性和ACC均高于PCs结果, 其分类正确率均大于97.85%, 因此在中级融合中LVs选择为产地鉴别的特征变量。 从原始的133× 2 242个降到133× 17个变量, 明显缩短模型拟合时间, 提高了产地鉴别能力。 LVs建立的RF模型的参数优化如图3(b1, b2)所示, 最优的ntree为267, mtry为2。
2.3.3 高级融合
高级融合的结果如表4所示, PCs的高级融合RF模型(高级融合PCs)训练集和预测集的SEN, SPC和ACC分别为0.80, 0.97, 77.42%和0.95, 0.89和95.00%, 其鉴别能力较差。 LVs的高级融合(高级融合LVs)RF模型对9个产地鉴别进行鉴别, 其RF模型最优ntree和mtry结果如图3(c)所示, 图3(c1)和(c2)是ATR-FTIR的RF模型最优ntree和mtry结果, 图3(c3)和(c4)是UV-Vis的RF模型最优ntree和mtry结果。 在高级融合中的133个样品中有42个样品需根据CART进行投票。 42个样品中有37个样品经投票后分类正确, 有1个样品分类出现分歧(No. 74, 文山薄竹), 其余4个样品分类错误。 分类错误及分歧样品的投票结果如表4所示。 No. 74样品被UV-Vis分类到Class4, 被ATR-FTIR分类到Class8, 最后获得相同票数。 4个被误分的样品中有2个(No. 21、 106)是ATR-FTIR投票结果正确, 而UV-Vis投票错误导致分类错误, 1个(No. 51)是ATR-FTIR投票错误, UV-Vis投票正确, 最终被分类错误, 剩余1个(No. 122)样品的ATR-FTIR和UV-Vis投票结果均错误。 高级融合LVs和中级融合LVs鉴别能力较好, 训练集和预测集的SEN和SPC均高于0.93, 其鉴别能力比低级融合和单光谱的鉴别能力增强, 但高级融合LVs模型存在过拟合现象。 中级和高级融合结果表明: 中级融合LVs建立不同产地滇黄精鉴别模型, 其训练集ACC为97.85%, 预测集ACC为97.50%, 鉴别能力最好。
| 表4 高级数据融合分类错误的样品投票结果 Table 4 Voting results of misclassified samples in high-level data fusion |
滇黄精的UV-Vis光谱在紫外-可见光区吸收峰是芳香族和含有共轭体系黄酮类成分的化学信息, 其ATR-FTIR光谱的吸收峰显示的是官能团和化学键信息。 两种指纹图谱反映不同的化学成分信息, 融合两种光谱的化学信息可以更加全面的反映其化学信息, 可对滇黄精实现更加全面的质量评价。
探讨了ATR-FTIR和UV-Vis及数据融合策略结合RF算法对9个产地滇黄精鉴别的可行性。 通过两种光谱对滇黄精产地鉴别分析表明, 单一光谱对产地评价不够全面, 可以利用数据融合策略来弥补不足, 提取光谱的两种特征值结合RF方法提高了对产地的鉴别效果。 采用SEN和SPE和模型分类正确率筛选出最佳模型, 其鉴别能力为中级融合LVs> 中级融合PCs> 低级融合> UV-Vis > ATR-FTIR> 高级融合PCs; 提取LVs对产地鉴别的方法优于PCs; 中级融合LVs建立的RF模型分类正确率最高, SEN和SPE大于0.98, 模型性能最佳, 为黄精药用资源的科学评价提供理论依据和技术支撑, 同时为其它中药材鉴别新方法的建立有借鉴作用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|




