作者简介: 段 龙, 1998年生, 石河子大学信息科学与技术学院硕士研究生 e-mail: duanlong@stu.shzu.edu.cn
棉花精量播种技术目前已经在新疆兵团全面推广, 该技术能精确实现一穴一粒的农艺技术指标, 但是也对高质量棉种的筛选提出了更高的要求。 为了避免播种往年活力不足的棉种而导致发芽率降低的问题, 结合机器学习和近红外(NIR)高光谱成像技术(HSI)进行棉种年份精确鉴别, 实现棉种的快速无损筛选。 采集2016年—2019年近四年外观无明显差异的棉种各360粒, 共1 440粒棉种(按照3:1:1划分训练集、 验证集和测试集)作为样本, 按照每批60粒采集915~1 698 nm范围的棉种高光谱图像, 去除首尾两端噪声大的光谱, 保留1 002~1 602 nm范围的光谱为原始数据。 利用Savitzky-Golay(SG)平滑算法对光谱进行预处理, 采用主成分载荷方法(PCA-loading)选取13个特征波段, 基于全部光谱数据和特征波段(±10 nm)数据建立逻辑回归(LR)、 偏最小二乘判别分析(PLS-DA)、 支持向量机(SVM)、 循环神经网络(RNN)、 长短记忆网络(LSTM)和卷积神经网络(CNN)六种分类模型。 使用全光谱数据建模时, 六种分类模型在测试集上的鉴别准确率分别为96.27%, 98.98%, 99.32%, 96.95%, 97.63%和100%, 其中CNN和SVM模型取得了较好的结果; 使用特征光谱数据建模时, 六种分类模型在测试集上的鉴别精度分别为93.56%, 97.29%, 98.30%, 95.25%, 94.24%和99.66%, 其中CNN和SVM模型仍有较好的分类结果。 结果表明, 使用全光谱数据建模时, 六种分类模型都可以实现较高精度的棉种年份鉴别, 使用特征光谱数据建模时CNN和SVM模型的鉴别精度仍可达到98%; 其中深度学习方法优于传统机器学习方法, 但是传统机器学习方法仍能保持较好的鉴别准确率。 因此, 结合近红外高光谱成像技术和机器学习方法能够实现棉种年份的高精度鉴别, 为棉花精量播种过程中的优质棉种选种技术提供理论依据和方法。
At present, the technology of precision cotton seeding has been promoted comprehensively in Xinjiang Corps, which can accurately achieve the agronomic technical standards of one grain per hole, but it also sets higher demands for the screening of high-quality cotton seeds. To avoid the decrease of germination rate caused by the cotton seeds with lack of vitality in previous years, machine learning and near-infrared (NIR) hyperspectral imaging (HSI) technology can be used to identify cotton seed years with high precision and to screen cotton seeds quickly and nondestructively. A total of 1 440 cotton seeds with no difference in appearance were collected in 2016, 2017, 2018, and 2019, and 360 seeds per year (According to 3:1:1, it is divided into the training set, validation set, and test set.) as samples. Hyperspectral images of cotton seeds in the range of 915~1 698 nm were collected according to each batch of 60 seeds, and average spectra (1 002~1 602 nm) for removing obvious noise at the beginning and the end were extracted as the raw data. SavitzkyGolay (SG) smoothing algorithm was used to preprocess the spectra. The principal component analysis loading (PCA-loading) method was used to select 13 effective wavelengths. Six classification models, including logistic regression (LR), partial least squares discriminant analysis (PLS-DA), support vector machine (SVM), recurrent neural network (RNN), long-short memory network (LSTM), and convolution neural network (CNN), were established based on full spectra and effective wavelengths. When using full spectra to build models, the identification accuracy of the six classification models on the test set was 96.27%, 98.98%, 99.32%, 96.95%, 97.63%, and 100%, respectively, among which CNN and SVM models had achieved good results. When using effective wavelengths to build models, the identification accuracy of the six classification models on the test set was 93.56%, 97.29%, 98.30%, 95.25%, 94.24%, and 99.66%, respectively, among which CNN and SVM models still had excellent classification results. The results showed that the six classification models could achieve high precision cotton seed years identification when the full spectra were used, and the identification accuracy of CNN and SVM models was still up to 98% when the effective wavelengths were used. The deep learning methods are generally better than the traditional machine learning methods, but traditional machine learning methods can still maintain good identification accuracy. Therefore, the combination of near-infrared hyperspectral imaging technology and machine learning methods can achieve high-precision identification of cotton seed years. It provides theories foundation and methods for selecting high-quality cotton seeds in the process of precision sowing.
棉花种植面积不断扩大, 为深入推进棉花全程机械化战略目标, 棉花精量播种技术受到了充分重视。 该技术要求精确实现一穴一粒的农艺技术指标, 因此对棉种质量提出了严格的要求。 棉种破损程度和储存的年限是棉种质量的两个重要评价指标。 高攀等[1]利用近红外高光谱成像技术实现了微破损棉种的无损检测; 但对外观无明显差异, 年份却不同的棉种鉴别尚未见相关研究。 储藏时间较长的棉种, 其种子活力和发芽率会严重降低[2], 混入正常棉种中并进行精量播种会影响棉花出苗率, 最终影响棉花产量。 因此, 准确有效鉴别棉种年份尤为重要。
种子年份鉴别的传统方法包括经验鉴别和化学分析, 采用经验鉴别时, 对鉴别经验要求较高, 鉴别准确率不稳定; 化学分析因其破坏性采样和技术要求高等因素, 无法大规模检测。 高光谱成像技术(hyperspectral imaging, HSI)有着近似连续的光谱信息、 探测识别能力极大提高, 其在果蔬、 谷物的品质检测、 成分分析等方面广泛应用[3, 4]。 Feng等[5]利用HSI成功寻求到一种鉴别不同加速老化时间下玉米籽粒活力的方法, 不同老化时间下的鉴别准确率从61%到100%不等。 在年份鉴别方面, Huang等[6]基于HSI开展了不同年份玉米种子分类研究, 使用最小二乘支持向量机方法得到了精度为94.4%的分类结果。 研究表明, HSI应用在分类检测、 成分分析、 年份鉴别等方面是可行性。
HSI具有波段多、 光谱分辨率高、 信息量大、 信息相关性强等特点[7], 因此, 选取合适高效的数据分析处理方式对充分利用HSI尤为重要。 深度学习作为机器学习的一个新的研究方向, 具有特征学习、 特征提取和海量数据处理能力[8], 并且在光谱分析和高光谱图像处理领域有所应用, Gao等[9]将HSI与深度学习相结合, 成功鉴别了沙枣的不同产地, 鉴别精度达到97.79%, Feng等[10]基于HSI和深度学习方法成功检测了冬枣中的细微损伤, 不同产地的冬枣检测精度均在90%~100%之间。 研究表明, 机器学习和深度学习方法可用于HSI处理, 并实现种类鉴别。
本文旨在通过采集棉种的高光谱图像, 并结合机器学习和深度学习进行特征选择与特征提取, 对不同年份棉种进行快速无损鉴别, 为棉花精量播种过程中优质棉种选种技术提供理论依据和方法。
实验样品为新疆石河子市棉花研究所2016年— 2019年四年的新陆早71号棉种。 取4个年份棉种各360粒, 共采集1 440份无破损、 外观无明显差异的棉种样品, 样品如图1所示。
在图1中, (a)— (d)四子图中分别为2019年, 2018年 2017年和2016年的样本。 为了建立分类模型, 将样本随机分成训练集、 验证集和测试集(3: 1: 1)。 具体划分如表1所示。
| 表1 样本划分 Table 1 Sample classification |
采用近红外(near infrared, NIR)高光谱成像系统拍摄了近四年不同年份的棉种表面。 NIR高光谱成像系统由四个模块组成, 包括成像模块、 照明模块、 升降模块和软件模块, 其具体信息如表2所示。
| 表2 仪器信息 Table 2 Instrument information |
NIR高光谱成像系统采集的图像尺寸为288× 512× 640(波段数× 像素宽× 像素长), 在拍摄过程中将60个同一年份的棉种样品放置在黑色面板上并调整SOC与样本的距离为79 cm, 使成像模块能够捕捉到所有样本。 HSI采集后, 根据式(1)将原始HSI进行校正[11]。
式(1)中, Ir是校正后的值, Io对应原始图像, Ig是灰色(50%黑色和50%白色组合)参考图像(使用SOC提供的灰色参考板拍摄)。
考虑到拍摄过程中存在明显的电信号噪声, 采用SG平滑滤波器对图像进行预处理(核大小为5× 5× 5, 多项式阶数为3), 以减少随机噪声。 用掩模法逐个提取单粒棉种的HSI, 计算并平均反射光谱。 利用SOC获得的1 100 nm波段的图像来计算掩模, 统计含有单粒棉种的掩膜的坐标。 根据坐标从HSI中提取含有单粒棉种的子图像, 并提取出计算的平均光谱。 光谱提取过程如图2所示。
1.4.1 逻辑回归
逻辑回归(logistic regression, LR)在机器学习和统计领域是最经典的分类模型之一, 可以获得良好的鉴别结果[12]。 对于LR模型, 通过网格搜索算法选择合适的正则化参数、 损失函数优化算法和正则化系数C来优化模型, 正则化参数选择L2, 损失函数优化算法和正则化系数C的调参范围分别为{newton-cg, lbfgs, liblinear, sag}和10-5-50。
1.4.2 偏最小二乘判别分析
偏最小二乘法判别分析(partial least squares discriminant analysis, PLS-DA), 是多变量数据分析中常用的判别分析法, 是最常用的分类模型之一[1]。 PLS-DA具有模型参数少、 解释样本观测数目少和可以减少变量间多重共线性产生的影响等优势。 在本实验中, 对于PLS-DA模型, 通过网格搜索算法选择合适的主成分数量来优化模型, 主成分数量的调优范围为2~20。
1.4.3 支持向量机
支持向量机(support vector machine, SVM)是常用的分类模型, 可用于定量和定性分析[13], 其泛化能力强, 通过最大化决策边界来获取稳定的分类结果。 在此, 对SVM模型, 通过网格搜索算法选择合适的核函数、 正则化系数C和核系数γ 来优化模型, 核函数、 正则化系数C和核系数γ 的调参范围分别为{poly, rbf, sigmoid}、 10-5-50、 10-5-50。
1.4.4 循环神经网络
循环神经网络(recurrent neural network, RNN)常用于处理序列化数据, 它能够有效获取序列变化数据的特征信息[14]。 实验所采集的光谱数据也为序列数据, RNN可用作探索不同波段之间的关联关系, 理论上会有良好的分类效果。 在此, 设计包含两个部分的RNN结构, 如图3所示。
在图3中, 第一部分为输入层和三个RNN层, 输入层的特征数为220, RNN层的隐藏层大小分别为110, 55, 28, RNN每一层包含层归一化以及激活函数, 使用LayerNorm进行层归一化, 选取tanh为激活函数; 第二部分由全连接层和输出层组成, 输出的类别包含4类。
1.4.5 长短记忆网络
长短记忆网络(long short-term memory, LSTM)是一种特殊的RNN结构, 能够更好地解决长序列训练过程中的梯度消失和梯度爆炸问题[15], 能够在处理更长的序列化数据中有更好的表现。 由于所采集的高光谱数据有大量波段, 所以采用LSTM在面对长序列光谱数据会有更好的分类效果。 设计包含两个部分的LSTM结构, 如图4所示。
在图4中, 第一部分为输入层和三个LSTM层, 输入层的特征数为220, LSTM层的隐藏层特征数分别为110, 55, 28, LSTM每一层都包含层归一化以及激活函数, 使用LayerNorm进行层归一化, 选取tanh为激活函数; 第二部分由全连接层和输出层组成, 输出的类别包含4类。
1.4.6 卷积神经网络
卷积神经网络(convolutional neural networks, CNN)在特征选择与提取、 识别与分类等方面的表现优于浅层网络, 具有两点优势: (1) 局部感知, 即CNN仅对数据的局部元素进行感知, 然后在更高层的网络将这些局部的信息进行融合, 从而得到数据的全部表征信息; (2) 权值共享的网络结构使网络模型的复杂度降低, 使权值的数量减少。 CNN现已广泛应用于多个领域[8, 9, 10], 但尚未发现被应用于棉种的高精度年份鉴别。 常见的CNN结构由以下六层组成: 输入层、 卷积层、 激活层、 池化层、 全连接层和输出层, 在本实验中, 所构建的CNN结构如图5所示。
在图5中, CNN结构包括输入层、 输出层、 全连接层, 以及3个卷积层和3个池化层; 每个卷积层与池化层之间包含层归一化和激活函数, 使用BatchNorm进行层归一化, 选取ReLU为激活函数; 其中输入层为训练集数据输入, 共220个波段点, 输出层为类别的预测值, 共四类。 卷积层的卷积核大小为1× 3, 每个卷积层卷积核的个数分别为100, 50和20。
采集的近红外光谱在915~1 699 nm范围内共288各波段。 在拍摄的过程中, 由于机器的启动和结束, 所获取的高光谱数据起止波段部分有较为明显的噪声, 为了保证数据的可靠性, 截去前后两端的波段, 选取1 002~1 600 nm, 其光谱数据如图6所示。
在图6全光谱曲线图中可以看到棉种的光谱反射率曲线的变化趋势具有明显的相似性, 在平均光谱曲线图中不同年份棉种的光谱虽然有少许差异, 但这些差异并不足以用作棉种年份的高精度鉴别。
主成分分析(principal components analysis, PCA)是一种通过数据降维将多变量转化为几个能够描述数据信息的主成分的统计方法。 利用PCA对棉种的光谱数据进行分析, 选取贡献率分别为96.8%, 2.7%和0.2%的主成分PC1, PC2和PC3, 贡献率累计达到99.7%, 涵盖了绝大多数的数据信息。 PC1, PC2和PC3的三维得分图如图7所示。
图7是PCA的3维得分图, 使用PCA方法降低了数据维度, 但是图中大部分数据仍然交错在一起, 加入PCA方法仍无法实现棉种年份的高精度鉴别。 因此, 需要对棉种光谱数据进一步的分析与处理。
基于全部光谱数据建立分类模型时, 常常因数据量过大而造成数据冗余和数据的共线性, 不利于有效提取光谱信息, 影响模型的分类效果。 为了减少数据计算量, 降低模型复杂度, 需要找到与棉种年份相关性较强的特征波段。 在本实验中, 采取了PCA-loading方法提取特征波段。
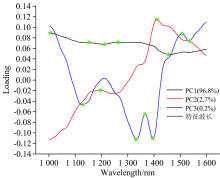
PCA-loading法可以反映主成分与原始光谱波段之间的相关性, 波段所对应的loading值越大, 表明其对主成分的贡献率越大[16]。 首先明确不同主成分的贡献率, 然后基于累积贡献率选取主成分, 最后设定阈值, 并以波段载荷图为依据筛选出波峰或波谷作为特征波段, 选择特征波段的过程如图8所示。
经筛选, 选取了1 005, 1 128, 1 152, 1 196, 1 212, 1 264, 1 332, 1 365, 1 395, 1 411, 1 455, 1 507和1 534 nm共13个特征波段, 将特征波段(± 10 nm)用作模型训练。
建立了六种分类模型, 包括传统机器学习方法LR, PLS-DA和SVM, 深度学习方法RNN, LSTM和CNN。 为了便于分类模型的建立, 按照划分好的数据集将去除噪音的数据作为输入数据, 所使用的python版本为3.7.0, 使用scikit-learn0.2.1构建LR、 PLS-DA和SVM模型, 使用pytorch1.5.0构建CNN、 RNN和LSTM模型, 模型的迭代次数设置为200次。 六种不同的分类模型的分类结果如表3所示。
| 表3 不同模型鉴别结果 Table 3 Identification results of different models |
在表3中, 对于棉种的全光谱近红外光谱数据, 六种模型均具有良好的性能, 训练集、 验证集和测试集的分类准确率均超过96%。 SVM和CNN模型的结果接近, 均超过99%, 而LR, PLS-DA, RNN和LSTM模型的结果相对较低。 LR模型采用L2范式作为损失函数, 模型参数(C, solver)经调优为(48.7, lbfgs), 其在训练集、 验证集和测试集中的分类准确率超过96%。 在PLS-DA模型中, 保留的主成分数为7个, 其在训练集、 验证集和测试集中的分类准确率均超过98%。 对于SVM模型, 最终模型参数(kernel, gamma, C)经调优为(rbf, 3.2, 20.4), 训练集, 验证集和测试集的分类准确率均超过98%。 对于RNN, LSTM和CNN三个深度学习模型, CNN的分类精度在划分的三个数据集中均达到100%, 而RNN和LSTM仅达到96%。 由此可见, 在处理全光谱数据时, CNN相较于其他深度学习模型有更好的表现, PLS-DA和SVM相较于LR有更好的表现。
对于棉种的特征光谱数据, 六种模型的分类效果都有不同程度的降低, 但仍具有较好的性能。 SVM和CNN模型的结果接近, 测试集精度均超过98%, 相较于全光谱数据, 精度降低1%。 PLS-DA模型精度降低2%, RNN精度降低了1%, LSTM精度降低2%, 但是仍保持在94%以上, 但是LR模型的分类精度仅有93%, 精度降低4%。 在特征光谱数据的训练中, LR模型采用L2范式作为损失函数, 模型参数(C, solver)经调优为(48.1, newton-cg); PLS-DA模型经调优, 保留15个主成分; 对于SVM模型, 最终模型参数(kernel, gamma, C)经调优为(rbf, 22.0, 24.4)。 对于RNN, LSTM和CNN三个深度学习模型, CNN的分类精度最高, 所降低的精度也最小, 而RNN和LSTM仅达到94%和95%。 由此可见, 在处理特征光谱数据时, CNN相较于其他深度学习模型有更好的表现, PLS-DA和SVM优于LR。
结合机器学习和近红外高光谱成像技术, 实现了棉种的年份精确鉴别。 采用SG方法进行光谱平滑, 使用PCA-loading方法进行特征波段选取, 使用六种分类模型(LR, PLS-DA, SVM, RNN, LSTM和CNN)分别对棉种的全谱和特征光谱数据进行建模分析。 结果表明, CNN和SVM分类模型的效果优于其他模型, 深度学习分类模型优于传统机器学习。 由于选取特征光谱, 丢失了部分数据信息, 用特征光谱数据建模, 分类效果会有不同程度的降低, 其中CNN和SVM两个分类模型仅降低1%, 测试集精度仍达到98%, PLS-DA在测试集上的分类精度达到97%, 其他三种模型的分类效果仅达到94%左右。 因此, 在采用近红外高光谱数据进行棉种年份鉴别时, CNN和SVM相较于其他四种分类模型是更好的选择。 结合整体的研究结果, 将高光谱成像技术与机器学习相结合可用于棉种年份精确鉴别, 为完善棉花精量播种技术提供了理论依据与方法。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|