作者简介: 许毓婷, 女, 1996年生, 长春理工大学理学院硕士研究生 e-mail: 2742592493@qq.com
近年来, 激光诱导击穿光谱(LIBS)与算法相结合分类、 识别生物组织逐渐兴起。 由于猪肉各部位组织光谱特性相似, 仅通过分析光谱信息很难达到准确识别的效果, 采用来自于同一个体、 四个不同部位的猪肉进行研究, 并将其进行切片、 压平, 应用LIBS技术对4种部位的组织(里脊, 梅花, 前腿, 五花)进行了样品光谱的采集, 每种样品采集100幅光谱进行分析, 选取Ca, Na, K等6条谱线进行了初步光谱分析, 观测谱线发现除脂肪含量较多的五花组织C—N以及C含量较其他组织高以外, 其他组织很难区分, 进一步对这6个成分进行主成分分析(PCA), 得到PC1, PC2, PC3累计贡献率达到95%, 通过特征分数作为支持向量机(SVM)模型输入源, 建立SVM分类模型, 得到几种部位样品的混淆矩阵图, 通过观察混淆矩阵可以清楚分辨出每个种类样品的分类整准确率, 发现四种样品准确率分别为96%, 98%, 97%, 100%, 平均准确率达到了97%以上。 研究证明LIBS结合PCA-SVM可作为一种快速鉴别猪肉不同组织部位的检测方法。
In recent years, laser-induced breakdown spectroscopy (LIBS) is gradually emerging to classify and identify biological organizations by combining them with algorithms. Due to each part of the pork similar spectral characteristics, it is difficult to achieve accurate identifications only through the analysis of the effect of spectral information, so in this paper studied pork from four different parts of the same individual and sliced and planished them, then applied LIBS technology on four parts, i.e., the organization, fillet, plum flower, and front legs. 100 specimens of each sample were collected and the spectrum analysis was conducted. A preliminary analysis of the spectrum was performed on Ca, Na, K and 6 lines. It was found that other tissues were difficult to distinguish except for the C—N tissue of plum flower with more fat content and higher C content than other tissues, so the Principal Component Analysis (PCA) on these 6 principal components was carried out. The cumulative contribution rate of PC1, PC2 and PC3 reached 95%. The Support Vector Machines (SVM) classification model was established by employing feature scores as the input source of SVM model, and the confusion matrix diagram of these samples got obtained. Through observation of the confusion matrix, the classification accuracy of each type of samples could be clearly distinguished. The results showed that the accuracy of the four samples was 96%, 98%, 97% and 100%, respectively, with an average accuracy surpassing 97%. The study proved that LIBS combined with PCA-SVM can be used as a fast identification method for different parts of pork tissues.
猪肉制品营养成分丰富, 在人类饮食中占有重要地位, 由于近年来猪肉制品价格水涨船高。 不同部位的猪肉组织较为类似, 在某些猪肉产品销售市场上, 一些不良商家将廉价部位的猪肉以次充好高价售卖, 让消费者无法分辨。 猪肉部位的传统检测方法主要有物理学方法(显微检测法)[1]、 免疫学方法(蛋白质检测)[2]和分子生物学方法(检测DNA分子)[3], 但这些方法操作繁琐, 检测周期长, 需专业技术人员操作。 基于分子光谱学的猪肉部位检测方法如近红外光谱技术和拉曼光谱技术[4], 方法虽便捷, 由于各部分猪肉成分组成接近且猪肉组织接近, 造成猪肉部位分类识别的识别率有待提高。
激光诱导击穿光谱技术(laser-induced breakdown spectroscopy, LIBS)[5]是一种利用高能激光脉冲激发待测样品并产生等离子体, 通过分析样品的等离子体发射光谱, 最终识别待测物质成分的分析方法。 近年来, LIBS作为一种新兴的光化学元素分析技术, 具有快速、 绿色、 无损检测等优点[6], 因此LIBS技术在冶金、 航天、 土壤污染、 水体污染、 食品安全等多个领域进行推广和应用[7, 8]。 在食品安全领域, 王彩虹[9]等基于LIBS技术和线性回归方法对猪肉中的Cr元素进行定量分析, 分析了单变量和多变量特征谱线对猪肉中Cr元素的定量分析影响。 Gonca Bilge[10]等将猪肉、 牛肉、 鸡肉碾碎混合, 基于LIBS技术并结合主成分分析(principal component analysis, PCA)机器算法, 对肉类的分类和识别准确率达到83.37%。 汪海燕[11]等提出了LIBS技术结合KNN和支持向量机(support vector machines, SVM)机器算法对猪油、 猪皮、 里脊等软组织进行分类识别, 识别准确率和灵敏度分别达到了99.83%和99.5%。
研究上述表明LIBS技术结合机器学习对肉类组织以及肌肉组织的分类和识别是可行的, 但在猪肉部位分类和识别方面的研究还未见报道。 本工作通过LIBS技术与PCA-SVM机器学习相结合, 对同一头猪的不同猪肉部位开展研究, 实现猪肉部位的分类和识别。
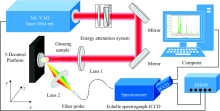
猪肉部位的分类和识别LIBS实验装置示意图如图1所示, 实验均在一个大气压、 环境温度23 ℃、 相对湿度40%的洁净空间下完成。 激光光源为输出波长1 064 nm的Nd:YAG激光器(SLII-10, Continuun), 光束模式TEM00, 脉宽为10 ns, 重复频率为10 Hz, 光束直径约为6 mm, 最大输出脉冲能量为250 mJ。 激光光束经二分之一玻片和格兰棱镜组成的能量衰减系统, 经1 064 nm HR镜和焦距为120 mm的石英透镜聚焦猪肉组织表面, 焦点位于猪肉样品表面内0.1 mm, 诱导击穿猪肉的激光脉冲能量为60 mJ。 诱导击穿产生的等离子体由45° 放置、 焦距为75 mm的透镜收集经光纤耦合传输到配有ICCD探测器的中阶梯光谱仪(Mechelle 5000, Andor, 波长范围200~850 nm, 光谱分辨率为0.05 nm)进行探测。 ICCD探测器和激光输出同步触发由数字脉冲延时发生器DG645控制, ICCD探测器相对激光脉冲延时为1 μs, 积分时间1 μs。 由于猪肉组织样品表面不均匀, 为使猪肉样品不被击穿打在载玻片上产生误差, 在实验中同时保证PI平移台处于以0.1 mm· s-1的速度移动, 采集100个脉冲的LIBS光谱并进行平均, 降低脉冲能量抖动对LIBS光谱的稳定性影响, 且在同一实验条件下每个猪肉部位样品采集100幅光谱。
实验样品购自长春市莲花山猪肉市场的同一头肉猪的前腿肉、 梅花肉、 里脊肉、 五花肉。 由于猪肉组织成分的表面分布不同于固体金属表面均匀稳定。 本实验为了减小样品的随机性, 对同一种类的猪肉采集多个切片; 样品形态、 表面温度、 干燥程度均会对实验结果产生影响, 为减小外界因素对实验结果产生影响, 实验开始前通过人工切片的方式将猪肉切成7 mm×2 mm×1 mm片状样品, 并放置于零下20℃的保鲜柜中, 为反映实验结果真实, 不做干燥处理。 实验开始前将样品取出, 将样品放置在三维平移台上, 确保每次激光作用于新的激光烧蚀点。
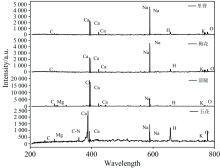
四种猪肉部位降噪处理后的LIBS光谱如图2所示。 由图2可知, 猪肉组织中观察到H 656.2 nm, O 777.1 nm等非金属元素谱线, 由于实验在空气环境中进行, 空气等离子体发射光谱可能会对猪肉组织LIBS光谱中的H和O非金属元素造成一定的干扰, 因此H和O元素不能作为分析谱线。 由于猪肉组织有大量的碳水化合物脂肪组织, 因此实验中观察到了C—N谱线如385.4和387.1 nm。 猪肉组织LIBS光谱中还存在Mg Ⅱ 279.8 nm, Ca Ⅱ (393.3, 396.8, 422.7, 430.2和445.4 nm等), Na Ⅰ 588.9 nm, Na Ⅰ 589.0 nm, K Ⅱ 766.4 nm和K Ⅰ 769.9 nm等金属元素谱线。 一般LIBS的特征谱线的选取, 要选择干扰较少, 信背比较高, 峰值明显, 易激发的波段, 如图3所示。 通过实验所得的光谱信息, 参照美国NIST原子光谱数据库, 选取Ca Ⅱ 393.3和422.7nm, Na Ⅰ 588.9和589.0 nm, K Ⅱ 766.4 nm, K Ⅰ 769.9 nm作为猪肉组织分类和识别的LIBS特征分析光谱线。
脂肪含量较多的五花肉LIBS光谱中存在大量C—N及C元素谱线峰, 且谱线强度明显比其他三种部位组织样品高。 由于实验选择的前腿肉、 梅花肉、 里脊肉、 五花肉四类组织都来自同一种生物个体, 获得的LIBS特征谱线都较为相似。 若从LIBS光谱图中分辨猪肉部位, 脂肪含量较高的五花肉和含钙较高的前腿肉可以直接区分, 而梅花肉、 里脊肉两种猪肉组织元素LIBS光谱强度差异较小, 不能进行直接分辨。 因此仅通过4种部位组织的LIBS光谱无法对猪肉部位组织进行分类鉴别, 需结合机器学习算法对猪肉部位进行分类识别。
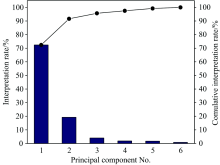
分析猪肉组织的LIBS光谱, 元素线状光谱和分子光谱有十多条之多, 因此对猪肉部位进行分类识别的过程中, 需要减少LIBS谱线的分析数据量。 主成分分析(PCA)是一种常用的线性变换技术, 能够降低噪声和维度[12], 使用PCA对猪肉部位LIBS光谱数据进行降维处理, 同时能够保留大部分原始光谱数据信息。 将选取的Ca Ⅱ 393.3和422.7 nm, Na Ⅰ 588.9和589.0 nm, K Ⅱ 766.4 nm, K Ⅰ 769.9 nm六个特征谱线强度作为输入变量, 得到PC1, PC2, PC3, PC4, PC5和PC6的主成分贡献率及累计贡献率如图3所示。
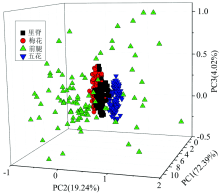
由图3可知, PC1, PC2和PC3的累计贡献率达到了95%以上, 说明由PC1, PC2和PC3三个主成分基本能够表征猪肉部位的LIBS光谱全部信息, PC1, PC2和PC3三个主成分的三维散点图如图4所示。
由图4可知, 经PCA降维后发现, 里脊、 梅花、 五花肉数据点呈现出明显的聚类, 但里脊和梅花肉部位信息存在部分重叠。 前腿的光谱数据点较为分散, 没有呈现出聚类, 表明前腿猪肉组织部位包括了里脊、 梅花和五花肉类组织的生物特性, 因此前腿部位的光谱数据和里脊、 梅花、 五花等部位光谱数据重叠区域较大。
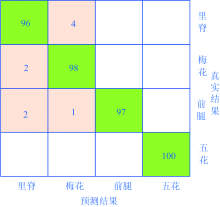
由于生物组织光谱数据所固有的高维性, 需结合一种使用大数据统计的分类算法, 支持向量机(support vector machine, SVM)[13]是一种基于统计学习理论的新型学习机。 本文将采用SVM结合PCA算法对4种猪肉部位进行分类识别, 以PC1, PC2和PC3主成分代替猪肉的特征光谱, 构建猪肉样品的特征空间向量, 特征向量构成的数据矩阵作为SVM识别算法的输入量。 为保证猪肉部位识别模型的稳定性, 采用十倍交叉验证的方式对模型进行评估, 得到分类识别结果混淆矩阵如图5所示。
由图5的猪肉部位PCA-SVM分类识别混淆矩阵可知, 里脊组织、 梅花组织、 前腿组织、 五花组织的分类识别率分别为96%, 98%, 97%和100%, 说明基于LIBS技术结合PCA-SVM分类模型可以有效地实现猪肉部位的快速识别。 猪肉组织相邻较近的组织成分接近, 因此LIBS光谱数据的信息冗余性较高且噪声干扰较强, 通过PCA实现LIBS光谱分析变量减少, 克服了生物组织光谱数据信息重叠性及噪声干扰。 在PCA基础上, 构造核函数的方式将两条相近不可分的谱线映射到高维空间中, 再从高维空间得到线性区分, 采用SVM算法克服了猪肉组织光谱数据维度较高的缺点, 因此, PCA-SVM机器学习算法可有效提高了猪肉部位的分类准确率。
通过LIBS技术结合PCA-SVM机器学习对猪肉部位识别进行了研究。 通过对比分析同一头肉猪的前腿肉、 梅花肉、 里脊肉、 五花肉的4个部位组织的LIBS特征光谱, 发现4个部位组织的LIBS特征光谱具有较高的相似性。 通过PCA算法对猪肉部位LIBS光谱数据进行降维处理, 里脊、 梅花、 五花呈现出明显的聚类, 但里脊和梅花部位信息存在部分重叠, 前腿的光谱数据点较为分散, 没有呈现出聚类。 通过十倍交叉验证的方式, 采用PCA-SVM分类模型得到里脊组织、 梅花组织、 前腿组织、 五花组织的分类识别率分别为96%, 98%, 97%和100%, 表明LIBS与机器学习相结合的方法可以作为一种绿色、 快速的生物组织检测技术手段。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|