作者简介: 宋 楠, 1985年生, 中国科学院长春光学精密机械与物理研究所副研究员 e-mail: 158507394@qq.com
高光谱成像(HSI)是一种新兴的快速、 安全的现代医学检测技术。 将自行研制的高光谱成像仪与显微镜相结合, 对人源脑胶质瘤小鼠和H&E染色病理切片进行成像光谱研究。 通过对脑胶质瘤与正常脑组织的高光谱数据进行分析和处理, 获取二者之间的光谱差异以实现对脑胶质瘤与正常脑组织的区分。 实验结果表明, 无论是病理切片还是在体脑组织数据, 脑胶质瘤与正常脑组织反射光谱中特定光谱范围内的反射率均存在一定差异, 可据此对二者进行区分, 为今后临床实验与脑胶质瘤术中辅助诊断提供依据和参考。
Hyperspectral imaging (HSI) is a new rapid and safe modern medical detection technology. In this paper, a self-developed hyperspectral imager was combined with a microscope to study the imaging spectrum of glioma tissue in human glioma mice and H&E staining pathology slides. The hyperspectral data of glioma and normal tissues were analyzed and processed to find the difference between them so as to realize the distinction of them. The experimental results show that there are some differences in the reflectivity within a specific spectral range between glioma and normal tissues, whether in pathology slides or in vivo data. We can distinguish glioma and normal tissues and provide reference and gist for clinical trials and auxiliary diagnosis in glioma surgery base on these differences.
脑胶质瘤是神经系统疾病中发病率较高的一种恶性肿瘤, 发病位置的特殊性使其致残、 致死率高, 患者预后极差[1, 2]。 目前最成熟的治疗方法为手术切除。 但由于脑胶质瘤呈浸润性侵袭性生长, 与周围的正常脑组织不存在明显的边界, 故最大程度的切除肿瘤区域的同时保护正常脑组织是手术中的一个难题[3]。 目前常用的术中辅助技术主要包括超声、 CT、 核磁共振等。 但这些方法有检测时间长、 不能实时定位、 存在毒副作用和设备价格昂贵等缺点, 且往往依赖医生经验判断[4]。 因此, 本工作尝试采用高光谱成像技术作为术中辅助诊断的一种新手段。
目前, 高光谱成像技术在地质勘测、 土壤植被生态监测、 海洋检测等方面取得了丰富的研究成果。 随着技术的不断发展, 其在医学领域的应用逐渐成为研究热点。 高光谱成像技术将光谱和图像合并, 形成包含二维空间图像数据与一维光谱数据的三维数据立方体, 不仅能获取物体的空间信息, 还能获得图像上每个像素点的光谱信息以分析物体的内部属性。 高光谱成像技术具备精确、 快速、 高灵敏度、 非接触、 无损伤、 高安全性等特点, 符合现代医学检测技术的发展要求[5]。 目前, 高光谱成像技术已经应用于视网膜情况检测和癌症检测等领域, 西班牙拉斯帕尔马斯大学(ULPGC)对脑胶质瘤病理切片的光谱特征展开了相关研究[6], 国内的第三军医大学开展了小鼠植入脑胶质瘤的光谱特征研究。
本文采用将自行研制的高光谱成像仪, 将其与显微镜结合, 采集脑胶质瘤小鼠脑部的高光谱成像数据, 对正常组织区域和肿瘤组织区域的光谱特征进行分析, 为高光谱成像技术在脑外科手术领域的临床应用提供依据。
将自行研制的高光谱成像仪与吉林大学中日联谊医院提供的奥林巴斯BX51显微镜和蔡司S5手术显微镜相结合, 如图1所示。 采集脑胶质瘤小鼠脑部的高光谱成像数据以及相关H& E染色病理切片数据, 并利用自主编写的光谱数据分析软件对采集结果进行分析。 该系统包含显微镜、 棱镜-光栅(PG)分光模块、 高分辨率CCD相机、 推扫机构、 控制计算机等组成。 光谱采集范围为400~1 000 nm, 光谱分辨率优于2.8 nm, 可采集270个波段的光谱数据, 图像尺寸为7.8 mm× 14.2 mm[7]。
 | 图1 高光谱成像系统 (a): 高光谱成像仪与显微镜结合; (b): 高光谱成像仪与手术显微镜结合Fig.1 Hyperspectral imaging system (a): Hyperspectral imager combined with a microscope; (b): Hyperspectral imager combined with a operating microscope |
为了实现图像数据和光谱数据的同时采集, 本系统采用面阵探测器接收的推扫方式进行数据采集。 CCD每帧采集的图像为: 沿狭缝方向一维成景物的空间像, 垂直于狭缝方向一维为空间各像元的光谱数据。 令电机垂直于狭缝方向运动, CCD同时进行采集。 将CCD采集的单帧图像融合得到一副完整的物质图像数据, 同时获得图像中每个像元的光谱数据。 这样就得到了一个完整的包含图像数据和光谱数据的物质数据立方体[8]。
所使用的胶质瘤细胞悬液、 无胸腺裸小鼠和标准H& E染色病理切片均由吉林大学中日联谊医院提供, 其中无胸腺裸小鼠体重15~20 g, 鼠龄5~7周, 饲养于无菌净化室内。 实验经吉林大学中日联谊医院审批, 并符合科技部《关于善待实验动物的指导性意见》要求。
共取8只无胸腺裸小鼠, 建立人脑胶质瘤裸小鼠脑内原位移植动物模型, 分别经20%水合氯醛吸入麻醉后, 俯卧位固定头颅, 酒精消毒皮肤, 在裸小鼠额部距颅中线右侧2.5 mm, 冠状缝前1 mm处先用直径1 mm的牙科钻小心钻穿颅骨, 然后用吸有瘤细胞悬液的微量进样器经孔垂直进入脑实质中, 进针深度为针尖距颅骨表面3.5 mm, 注射前将针稍微回退0.5~1 mm, 缓慢注入, 除针前停留2 min。 接种部位为裸小鼠右侧大脑尾状核。 肿瘤接种后19 d对裸小鼠进行MRI检查, 确认其颅内肿瘤形成。
每次进行高光谱成像数据采集前要采集环境暗噪声和标准漫反射率板的成像光谱数据, 作为高光谱成像数据的校正依据[9]。 整个数据采集过程保持在室温22 ℃、 恒定暗光照条件进行, 依次对每只脑胶质瘤小鼠进行数据采集。 首先, 将脑胶质瘤小鼠麻醉后, 俯卧位固定于立体定向仪底板上, 在手术显微镜下沿中线剪开约2 cm, 颅骨钻孔形成圆形骨窗, 暴露肿瘤表面。 其次, 调节搭载高光谱成像仪的手术显微镜, 使得肿瘤位于显微镜视野中心, 利用高光谱成像仪采集肿瘤的高光谱成像数据, 每隔1分钟采集一组, 共采集5组数据。 并将数据存储于计算机中。
数据采集完成后, 立即完整取出脑组织及肿瘤组织, 固定于4%多聚甲醛中, 24 h后进行石蜡包埋处理, 之后制作标准H& E染色切片。 利用搭载高光谱成像仪的显微镜进行脑胶质瘤染色病理切片的高光谱成像数据采集, 并将数据存储于计算机中。
光源辐射能量穿透样本或在样本表面反射后形成样本的特征光谱, 并为高光谱成像仪所记录, 对该特征光谱进行分析以寻找正常组织与肿瘤组织的光谱差异并以此实现二者之间的区分。 为降低噪声影响, 使光谱特征差异显著化以便于区分, 需要对所获取的光谱信息进行一定的数据处理。
2.4.1 非均匀性校正
对于本研究使用的高光谱成像仪, 探测器平面的每一行都有一定数量的探测单元, 对应一个波段, 随着推扫运动以行为单位获取成像光谱数据。 对于一个波段来说, 探测单元之间存在响应不一致的问题, 进而产生严重的条带噪声, 即图像列之间的不均匀, 影响成像光谱数据的后续处理和应用。 不同探测单元的响应函数会随着入射强度变化而不同, 因而导致条带噪声。 需要对数据进行非均匀性校正, 方法如下:
逐波段对原始成像光谱数据进行标准矩匹配校正, 得到标准矩匹配校正图像。 计算方法为
其中, D'i, j为矩匹配校正后像元值; Di, j为原始像元值; i和j分别为列和行号; M和N分别为列数和行数; μ i和μ r分别为第i列和参考列的均值; σ i和σ r分别为第i列和参考列的标准方差。
以标准矩匹配校正图像为基础, 选择相邻两列像元中相同像元, 对于某一波段来说, 选择方法为
其中, Φ i为所选择相邻两列像元中相同像元集; D'i, j , D'i-1, j为标准矩匹配校正像元值; T为阈值; abs()为取绝对值。
对于相邻两列原始数据, 以前一列为基准, 用两列中相同像元, 通过线性回归得到后一列的校正系数, 计算方法为
其中, Ai和Bi为校正系数; regress(· , · )为线性回归。 根据校正系数对后一列进行校正, 顺次遍历一个波段的所有列, 完成一个波段图像非均匀性校正。 计算方法为
其中,
2.4.2 光谱去噪
对于高光谱成像数据而言, 去噪需要尽可能保证光谱的形状。 SG滤波器是一种在时域内基于滑动滤波窗口多项式拟合的滤波方法, 具有计算量小, 计算速度快, 可以很好地保持波形的相对极大值、 极小值和脉冲宽度等特性, 比较适用于光谱去噪。 SG滤波过程如下:
确定滤波窗口和多项式次数。 根据高光谱成像仪的采样特点确定成像光谱数据滤波窗口大小及变化范围, 多项式次数及其变化范围。
用最小二乘法对原始成像光谱数据进行滑动拟合, 并求出卷积系数。 用卷积系数对原始成像光谱数据进行卷积计算, 完成SG滤波, 得到去噪后的成像光谱数据。 在2M+1成像光谱数据滤波窗口内, 可以用多项式来拟合波形数据, 多项式表达为
其中, p(n)为多项式; n为2M+1成像光谱数据滤波窗口内波形数据点的位置, -M≤ n≤ M; k为多项式次数; ak为多项式系数。 最小二乘法求出系数ak就可计算拟合后的成像光谱数据滤波窗口内某点的拟合数值, 该数值作为去噪后的数值。 按照上述方式, 遍历显微成像光谱数据所有像元。 窗口选择与仪器噪声水平有很大关系, 需要结合数据噪声大小进行调整, 噪声越大窗口越大。 因此, 对于特定仪器窗口相对较稳定, 所选择的去噪窗口大小5。 处理效果如图2所示。
 | 图2 光谱去噪处理 (a): 常规方法去噪处理结果; (b): SG滤波法去噪处理结果Fig.2 Spectral denoising (a): Noise removal by conventional method; (b): Noise removal by SG filtering method |
2.4.3 极差标准化处理
无论是反射光谱, 还是透射光谱, 其特征主要反映在光谱曲线的形状上。 形状的差异更有利于区分不同物质的特征光谱。 对光谱信息进行极差标准化处理, 可以更加直观地反映光谱曲线的形状特征。 光谱极差标准化的计算公式为
其中, S(λ )为样本的反射率或透射率; Smin(λ )为光谱的最小值; Smax(λ )为光谱的最大值。 极差标准化处理可以有效减弱因光照、 暗电流偏移等因素造成的光谱信息系统性误差。 极差标准化处理效果如图3所示。
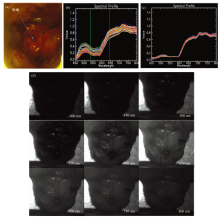
利用搭载高光谱成像仪的手术显微镜采集脑胶质瘤小鼠脑部成像光谱数据, 并对数据进行处理和分析。 图4为脑胶质瘤小鼠在体脑部成像光谱数据。
经过逐像元的反射率计算、 非均匀性校正和光谱去噪之后, 数据的条带噪声和光谱曲线噪声基本被消除。 观察图4(b)和(c)的光谱曲线, 在540~600 nm范围内, 非肿瘤脑组织反射光谱呈现“ W” 状, 而脑胶质瘤反射光谱较为平滑, 两种反射光谱存在一定区别。 这主要是因为血液中存在两种血红蛋白, 一种为含氧血红蛋白, 吸收峰位于554 nm左右, 含氧血红蛋白含量占总血红蛋白含量的百分比称为氧饱和度[10]。 由于肿瘤组织存在快速生长及血管异常的特点, 缺氧是肿瘤细胞普遍存在的状态, 肿瘤组织处的脱氧血红蛋白的含量高于非肿瘤脑组织, 导致氧饱和度低于非肿瘤脑组织。 可见, 在体状态下, 脑胶质瘤与非肿瘤脑组织在氧饱和度方面存在一定区别。
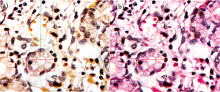
利用搭载高光谱成像仪的显微镜采集脑胶质瘤小鼠脑部H& E染色病理切片成像光谱数据, 并对数据进行处理和分析。 图5为脑胶质瘤小鼠脑部H& E染色病理切片成像光谱数据。
经过逐像元的反射率计算、 非均匀性校正和光谱去噪之后, 数据的条带噪声和光谱曲线噪声基本被消除。 从图5(c)中可见, H& E染色病理切片中的脑胶质瘤与非肿瘤脑组织反射光谱存在一定差别, 在530~540 nm范围内反射率有所不同。 H& E染色病理切片中的脑胶质瘤与非肿瘤脑组织反射光谱与在体状态下的脑胶质瘤与非肿瘤脑组织反射光谱相比存在差异, 主要原因可能是因为H& E染色病理切片中染色剂的存在、 染色不均性以及组织离体后细胞产生变化等, 还需要进行进一步研究与实验。
将高光谱成像技术应用于脑胶质瘤检测具有可行性。 研制了一台高光谱成像仪, 在VNIR范围内(400~1 000 nm)共获取8组脑胶质瘤小鼠的高光谱数据, 每组包含1组脑胶质瘤小鼠在体脑部成像光谱数据及1组脑胶质瘤小鼠脑部H& E染色病理切片成像光谱数据, 并通过多种数据处理手段对数据进行了分析和处理。 通过实验数据和理论分析可知, 在体状态下, 脑胶质瘤与非肿瘤脑组织在氧饱和度方面存在一定区别, 而H& E染色病理切片中的脑胶质瘤与非肿瘤脑组织反射光谱在特定波长范围内的反射率上存在一定差别。 初步表明高光谱成像技术可区分脑胶质瘤和非肿瘤脑组织, 进而实现脑胶质瘤的检测, 为今后临床实验与脑胶质瘤术中辅助诊断提供依据和参考。
综上所述, 高光谱成像技术已被证明可实现脑胶质瘤的检测, 但考虑到人体的复杂性和个体的差异性, 仍须进行进一步的研究, 如进行更多样本量的成像光谱数据采集, 建立丰富的脑胶质瘤高光谱数据库, 将高光谱成像技术与人工智能相结合, 形成基于高光谱成像的快速、 准确、 安全的脑胶质瘤检测技术, 最终实现对脑胶质瘤的术中辅助诊断。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|