作者简介: 刘金明, 1981年生, 东北农业大学工程学院博士研究生, 黑龙江八一农垦大学电气与信息学院副教授e-mail: jinmingliu2008@126.com
预处理是提高玉米秸秆生物转化利用效率的有效途径。 玉米秸秆经生物炼制转化为生物燃料时, 转化率与其原料内的纤维素和半纤维素含量直接相关。 为了实现对预处理后玉米秸秆的生物炼制过程的有效调控, 提出使用近红外光谱(NIRS)对玉米秸秆的纤维素和半纤维素含量进行快速检测, 解决传统化学方法测试速度慢、 成本高的问题。 为了提高NIRS检测的效率和精度, 将遗传算法与模拟退火算法相结合构建遗传模拟退火算法(GSA)用于预处理后玉米秸秆纤维素和半纤维素含量NIRS特征波长优选。 GSA算法以NIRS波长点数为码长进行二进制编码, 以偏最小二乘法(PLS)回归模型的交叉验证均方根误差为目标函数, 结合温度参数设计适应度函数, 基于Metropolis判别准则实现扰动解的选择复制, 能够在避免早熟的同时有效提高进化后期的搜索效率。 采用碱预处理、 生物预处理及其相结合的方法对采集的玉米秸秆进行预处理后制备样品120个, 并测定其纤维素和半纤维素含量及NIRS。 使用7点Savitzky-Golay平滑结合多元散射校正和标准正则变换对光谱进行预处理后, 利用Kennard-Stone法按3∶1比例划分校正集和验证集。 然后, 使用GSA算法对NIRS全谱进行特征波长优选(记为Full-GSA)、 对协同区间偏最小二乘法(SiPLS)优选后谱区进行特征波长优选(记为SiPLS-GSA)、 对反向区间偏最小二乘法(BiPLS)优选后谱区进行特征波长优选(记为BiPLS-GSA), 并使用PLS回归模型和验证集对特征波长优选结果进行评测。 Full-GSA以全谱1 557个波长点为基因, 执行16次算法, 优选出118个纤维素特征波长点和164个半纤维素特征波长点。 SiPLS-GSA经SiPLS优选的纤维素和半纤维素谱区波长点数分别为388个和160个, 再经GSA进一步优选后得到157个纤维素特征波长点和148个半纤维素特征波长点。 BiPLS-GSA经BiPLS优选的纤维素和半纤维素谱区波长点数分别为358个和180个, 再经GSA进一步优选后得到130个纤维素特征波长点和153个半纤维素特征波长点。 结果表明, 通过波长优选, 不仅参与建模的波长点数量显著减少, 而且回归模型的性能显著优于全谱建模。 其中, 采用Full-GSA优选的纤维素特征光谱回归性能最佳, 采用SiPLS-GSA优选的半纤维素特征光谱回归性能最佳。 回归模型验证集的平均相对误差(MRE)分别为1.752 4%和2.020 8%, 较全谱建模分别降低了13.636 6%和25.368 4%。 基于结合温度参数设计适应度函数的策略构建的GSA具有良好的全局搜索性能, 适用于玉米秸秆纤维素和半纤维素含量NIRS特征波长优选。 GSA以全谱每个波长点为染色体基因的编码方案适用于NIRS全谱的特征波长优选。 GSA同样适用于SiPLS和BiPLS优选后谱区的特征波长优选, 能够有效实现优选后谱区的波长点优选。
Pretreatment is an effective way to improve the utilization efficiency of the corn stover biotransformation. The conversion rate is directly related to contents of the cellulose and hemicellulose in corn stover during the bio-refinery conversion to biofuels. To achieve an effective control for the corn stover bio-refining process after the pretreatment, the near infrared spectroscopy (NIRS) was used to quickly detect contents of the cellulose and hemicellulose, solving the problems of being time consuming and high-cost in the traditional chemical analysis method. To improve the efficiency and precision of the NIRS detection, the genetic simulated annealing algorithm (GSA) based on genetic algorithm (GA) combined with simulated annealing algorithm (SA) was presented for optimizing the characteristic wavelength variables of NIRS. In the GSA, firstly, the number of the NIRS wavelengths was used as the code length for binary coding; secondly, the root mean square error of cross-validation (RMSECV) of the partial least squares (PLS) regression model was used as the objective function; thirdly, the fitness function was designed combining with the temperature parameter; and last, the selective replication of the perturbation solution was realized based on the Metropolis criterion. Therefore, GSA can effectively improve the search efficiency at the later stage of evolution while avoiding premature convergence. 120 samples of corn stover were prepared by using the pretreatments of alkaline, biology, and the combination of alkaline and biology. The contents of cellulose and hemicellulose were measured using the wet chemistry methods. The NIRS were collected using the Nicolet Antaris Ⅱ Fourier near infrared spectrometer. The spectrum was pretreated by 7 points Savitzky-Golay smoothing combining with multivariate scattering correction and standard normal variate transformation. The samples were divided into correction set and validation set by using Kennard-Stone algorithm at a ratio of 3∶1. The GSA is used for the characteristic wavelength variables optimizations of the NIRS whole wavelengths (Full-GSA), the synergy interval partial least squares selected spectral region (SiPLS-GSA), and the backward interval partial least squares selected spectral region (BiPLS-GSA), respectively. And then, the optimized results of the characteristic wavelength variables were evaluated by the PLS regressive model with the validation set. In Full-GSA, 1 557 wavelength points were used as chromosome genes in whole wavelengths, 118 cellulose characteristic wavelength points and 164 hemicellulose characteristic wavelength points were selected after 16 executions. In SiPLS-GSA, the cellulose and hemicellulose wavelength points of spectral region optimized by SiPLS were 388 and 160, respectively, and 157 cellulose characteristic wavelength points and 148 hemicellulose characteristic wavelength points were gotten after the further optimization by GSA. In BiPLS-GSA, the cellulose and hemicellulose wavelength points of spectral region optimized by BiPLS were 358 and 180, respectively, and 130 cellulose characteristic wavelength points and 153 hemicellulose characteristic wavelength points were selected after the further optimization by GSA. It was shown that not only the number of wavelengths was significantly decreased after the optimization, but also the performance of regressive model was obviously better than that of the whole wavelengths. The best performance of regressive model for cellulose characteristic wavelengths was obtained by Full-GSA, and the best performance for hemicellulose characteristic wavelengths was obtained by SiPLS-GSA. The mean relative error (MRE) values of validation set for cellulose and hemicellulose in the best model were 1.752 4% and 2.020 8%, which were decreased by 13.636 6% and 25.368 4% compared with the whole wavelengths, respectively. The GSA combining with temperature parameters to design the fitness function is suitable for the NIRS characteristic wavelength selection of the cellulose and hemicellulose contents in corn stover, and has a good global search capability. The encoding scheme of GSA using each wavelength point in whole wavelengths as chromosome gene is suitable for the characteristic wavelength selection of NIRS whole spectrum. GSA is also suitable for the characteristic wavelength selection of the spectral region optimized by SiPLS and BiPLS, and the selection of wavelength points in the optimized spectral region can also be achieved effectively.
玉米秸秆是我国主要的农作物秸秆之一, 是一种亟待处理和利用的可再生资源。 厌氧发酵产沼气和酶解糖化产乙醇等生物转化技术是将作物秸秆变废为宝的有效手段[1]。 但玉米秸秆因其自身紧密的木质纤维素结构而具有强大的耐酶解特性, 导致其生物转化效率偏低[2]。 玉米秸秆主要由纤维素、 半纤维素和木质素三种木质纤维素成分组成。 通过预处理打破这三种物质在细胞壁中紧密结合在一起形成的坚硬、 稳定的木质纤维素结构, 是提高玉米秸秆生物转化利用率的有效途径[3]。 玉米秸秆经生物炼制转化为生物燃料时, 转化率与其原料内的纤维素和半纤维素两种碳水化合物含量直接相关[4]。 因此, 为了对预处理后玉米秸秆的生物炼制过程进行有效调控, 有必要对秸秆中纤维素和半纤维素含量进行快速、 准确测定[5], 但采用传统化学方法测定其含量时存在测试速度慢、 成本高的问题[6]。
近红外光谱(near infrared spectroscopy, NIRS)分析技术具有简便、 快速、 无损、 低成本等众多优点[7], 已广泛用于农产品及农牧业废弃物的定性分析[8]和定量检测[9]。 近年来, 相关学者开始研究应用NIRS对植物细胞壁中的纤维素和半纤维素含量进行测定[10]。 Niu等[11]提出使用NIRS技术对不同成熟期的小麦秸秆进行纤维素和半纤维素等碳水化合物的测定。 Xue等[5]提出了一种NIRS在线检测方法用于玉米秸秆木质纤维素成分的实时检测。 上述研究主要以采集的秸秆为研究对象, 尚未见专门以预处理后的玉米秸秆为研究对象, 使用NIRS对其进行纤维素和半纤维素测定。
随着近红外光谱仪采集精度的提高, 采集的数据量越来越大。 以全谱波长点建模时, 计算量大, 波长冗余严重。 通过特征波长优选, 可以有效消除光谱中的不相干和非线性波长点对模型预测精度的影响。 相关学者提出应用遗传算法(genetic algorithm, GA)[12]、 粒子群优化算法[13]、 蚁群算法[14]等智能优化算法进行NIRS变量组合优化, 从而筛选出有效的波长变量。 其中GA具有较强的鲁棒性和全局搜索能力, 其随机搜索特性能够有效解决光谱波长点之间的共线性问题[12], 还可以与其他光谱谱区优化算法相结合进行特征波长点的优选[12, 14]。 但GA存在早熟问题, 且进化后期搜索效率较低。
因此, 将GA与模拟退火算法(simulated annealing algorithm, SA)相结合构建遗传模拟退火算法(genetic simulated annealing algorithm, GSA), 通过结合温度参数设计适应度函数, 在避免算法早熟的同时有效提高算法进化后期的搜索效率。 然后, 以偏最小二乘(partial least squares, PLS)回归模型的交叉验证均方根误差(root mean square error of cross-validation, RMSECV)为目标函数, 使用构建的GSA算法对预处理后玉米秸秆纤维素和半纤维素含量NIRS的全谱及协同区间偏最小二乘法(synergy interval partial least squares, SiPLS)和反向区间偏最小二乘(backward interval partial least squares, BiPLS)优选后的谱区进行特征波长优选, 从而有效提高纤维素和半纤维素含量NIRS回归模型的性能。
实验用玉米秸秆取自东北农业大学向阳农场, 自然风干后粉碎成10 mm的秸秆段备用。 依据秸秆在生物炼制过程中碱性预处理[15]效果好、 工艺简单, 以及生物预处理方法在环境友好性方面的优势, 采用地衣芽孢杆菌(生物方法)、 NaOH溶液(碱性试剂)、 猪粪沼液(富含微生物的弱碱性溶液)、 沼液加NaOH(富含微生物的强碱性溶液)共四种方法对玉米秸秆进行了预处理, 并按不同处理试剂浓度、 不同处理时间采样, 共计采样120个。 将预处理后玉米秸秆样品进行烘干、 粉碎, 过40目筛后装袋备用。
纤维素和半纤维素的测定按照Van Soest法的原理, 采用Ankom 200i半自动纤维分析仪依次对秸秆粉末进行中性洗涤纤维、 酸性洗涤纤维和酸性洗涤木质素的测定, 通过计算得到样品的纤维素含量(酸性洗涤纤维含量-酸性洗涤木质素含量)和半纤维素含量(中性洗涤纤维含量-酸性洗涤纤维含量)。 每个样品测试3次, 取3次的平均值作为待测含量值。
对预处理后的玉米秸秆样品粉末使用Nicolet公司的Antaris Ⅱ 型傅里叶近红外光谱仪进行积分球漫反射光谱扫描, 光谱采集范围10 000~4 000 cm-1, 分辨率为8.0 cm-1, 样品扫描32次, 装样方式为带透明塑料袋扫描, 扫描时去除透明塑料袋背景, 三次扫描取平均值作为样品的原始光谱。 原始光谱的波长点为1 557个, 数据点间距为3.86 cm-1, 起始波长点为10 001.03 cm-1, 结束波长点为3 999.64 cm-1。
2.1.1 算法初始化
算法的初始化包括编码、 种群初始化、 初温设定、 降温操作、 进化代数等。 编码方式采用二进制编码, 码长为待优选光谱波长点个数。 “ 1” 和“ 0” 分别表示该波长点对应的数据“ 是” 、 “ 否” 选中参与运算。 种群初式化时随机产生一个M× L的二元矩阵, 其中M为种群规模, L为码长。 初温的确定依据t0=K(f0_max-f0_min), 其中K是一个正整数, f0_max和f0_min为初始种群中的最大和最小目标函数值。 退温操作依据tn+1=α tn, 其中0< α < 1。
2.1.2 适应度函数设计
适应度函数对算法的进化方向起指导作用, 其设计的是否合理直接决定着算法的性能。 选择PLS回归模型的RMSECV作为目标函数, 结合温度参数设计适应度函数如下
式中, f(x)为当前染色体的目标函数值, fmin为当前代种群中的最小目标函数值, t为当前代温度值。
采用此适应度函数设计方法, 使得算法在初始阶段计算的适应度值差异较小, 能够有效避免个别优良染色体充斥整个种群导致算法收敛到局部最优解; 在进化后期优良染色体具有相对更大的适应度函数值, 更容易遗传给下一代, 进而加快算法的搜索速度。
2.1.3 进化过程设计
算法的进化过程分为GA的选择、 交叉、 变异操作和SA的Metropolis选择复制两部分。 GA的选择操作采用带最有保留策略的赌轮选择, 交叉操作采用离散重组交叉, 变异操作采用离散变异策略。 SA的Metropolis选择复制包括邻域解的构建和状态接受函数两部分。 邻域解的构建采用多位变异策略, 即在当前染色体中随机选择m位进行位变异。 状态接受函数基于Metropolis判别准则实现。
在进行NIRS波长优选前, 先使用Savitzky-Golay平滑、 一阶导数、 二阶导数、 多元散射校正(multivariate scattering correction, MSC)、 标准正则变换(standard normal variate, SNV)及其组合方法进行光谱预处理, 再使用Kennard-Stone法将预处理后光谱划分为90个校正集样本和30个验证集样本, 然后计算全谱下的PLS回归模型性能, 并基于预测均方根误差(root mean square error of prediction, RMSEP)来确定光谱预处理方法。 经计算比较后最终确定纤维素和半纤维素含量预测模型的光谱预处理方法为7点Savitzky-Golay平滑、 MSC和SNV。 在选定预处理方法后, 使用GSA算法分别对全谱(记为Full-GSA)及SiPLS优选后谱区(记为SiPLS-GA)和BiPLS优选后谱区(记为BiPLS-GSA)进行特征波长优选。 最后, 分别以三种优选后的特征波长为输入, 使用PLS建立秸秆纤维素和半纤维素含量定量分析模型, 并采用校正决定系数(
算法全部在Matlab R2012b软件平台中实现, 其中BiPLS和SiPLS基于itoolbox工具箱实现。
对120个样品的原始光谱经Savitzky-Golay平滑、 MSC和SNV预处理后, 使用Kennard-Stone法按3∶ 1的比例划分样本, 得到校正集样本90个、 验证集样本30个, 对应的纤维素和半纤维素含量如表1所示。
| 表1 纤维素和半纤维素含量 Table 1 Contents of cellulose and hemicellulose |
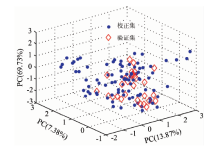
对预处理后的近红外光谱进行主成分分析, 第一、 第二和第三主成分的贡献率分别为69.73%、 13.87%和7.38%, 前三个主成分的累积贡献率达90.98%。 校正集和验证集的三维主成分空间分布情况如图1所示。
由表1和图1可知, 校正集和验证集样本的分布比较均匀, 校正集纤维素和半纤维素含量基本涵盖了验证集, 可以使用该样本划分方法进行NIRS分析。
3.2.1 Full-GSA特征波长优选
Full-GSA以全谱1 557个波长变量为基因, 随机生成300个码长为1557的染色体构建初始种群; 综合考虑搜索效率和性能, 选取初温确定系数K取100, 退温系数取0.8, 进化代数取50, 交叉概率取0.7, 变异概率取0.1, 邻域解扰动位数m取50。 为消除GSA算法的随机性, 分别执行算法10次(记为Full-GSA-10)、 12次(记为Full-GSA-12)和16次(记为Full-GSA-16)对纤维素和半纤维素含量特征波长进行优选。 多次执行时, 每次都选中的波长点代表了染色体的优良基因, 以这些特征波长点作为特征波长建立回归模型时, 可以有效消除GSA算法的随机性, 且能够得到较高的回归模型性能。 测试发现, 校正集和验证集回归性能参数
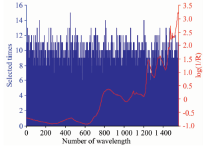
进行纤维素特征波长GSA优选时, 测试发现, 算法执行10次时, 选中9次以上的波长点(73个)回归性能最佳; 执行12次时, 选中10次以上的波长点(101个)回归性能最佳; 执行16次时, 选中13次以上的波长点(118个)回归性能最佳, 其优选结果与预处理后的平均光谱对比如图2所示。
由图2可知, 筛选出的特征波长中多数波长点在样本NIRS吸收峰附近, 能真实的反应纤维素对应的C— C, C— O, C— H, — OH和— CH2等官能团。 选中15次以上的波长点(37个)中, 258(9 009.80 cm-1), 400(8 462.11 cm-1), 406(8 438.97 cm-1), 470(8 192.13 cm-1), 630(7 575.02 cm-1), 665(7 440.02 cm-1), 667(7 432.31 cm-1), 785(6 977.19 cm-1)和822(6 834.49 cm-1)对应C— H, — CH2和— OH的二级倍频, 1 013(6 097.81 cm-1), 1 037(6 005.25 cm-1), 1 038(6 001.39 cm-1), 1 048(5 962.82 cm-1), 1 089(5 804.69 cm-1), 1 090(5 800.83 cm-1), 1 118(5 692.83 cm-1), 1 179(5 457.56 cm-1), 1 224(5 284.00 cm-1), 1 269(5 110.44 cm-1), 1 280(5 068.01 cm-1), 1 283(5 056.44 cm-1)和1 288(5 037.16 cm-1)对应着C— H, — CH2, C— O和— OH的一级倍频, 1 374(4 705.46 cm-1), 1 440(4 450.90 cm-1), 1 468(4 342.91 cm-1)和1 486(4 273.48 cm-1)对应C— C, C— H, — CH2和C— O的组合频。
在进行半纤维素特征波长优选时, 执行Full-GSA算法10次, 选中8次以上的波长点(83个)回归模型性能最佳; 执行12次时, 选中9次以上的波长点(119个)回归性能最佳; 执行16次时, 选中11次以上的波长点(164个)回归性能最佳, 其优选结果如图3所示。
由图3可知, 筛选出的多数特征波长点在样本的NIRS吸收峰附近, 能真实的反应半纤维素对应的C— C, C— O, C— H, C=O, — OH, — CH, — CH2, — CHO和— NH2等官能团。
3.2.2 SiPLS-GSA特征波长优选
SiPLS-GSA先使用SiPLS将全谱划分成多个均匀的子区间, 再组合不同个数的子区间进行建模, 选择模型RMSECV最小的组合区间作为SiPLS优选后谱区, 然后再使用GSA进行特征波长点优选。 为考察分割波长点个数对波长选择及模型预测性能的影响, 分别按约30, 40, 50, 60, 80, 100, 120个波长点划分子区间, 依次将光谱划分为52, 39, 31, 26, 20, 16和13个子区间, 并依据RMSECV选取2~4个子区间构建的组合区间作为SiPLS优选的特征谱区。 不同子区间个数下优选的纤维素和半纤维素特征谱区如表2所示。
| 表2 纤维素和半纤维维素SiPLS优选谱区结果 Table 2 Results of optimized spectral regions for cellulose and hemicellulose by SiPLS |
依据RMSECV选取划分16个子区间的最佳组合区间[11 12 13 15]作为SiPLS优选后的纤维素特征谱区。 SiPLS-GSA将该组合区间对应的976~1 266和1 364~1 460波长点(388个)作为GSA的输入波长进行再次寻优, GSA算法参数设置如下: 码长为388, 种群规模为80, 退温系数取0.9, 进化代数取100, 邻域解扰动位数m取15, 其他参数与Full-GSA一致。 算法连续执行10次后, 经计算后确定选中6次以上的波长点(157个)作为SiPLS-GSA优选的纤维素特征波长。
选取划分39个子区间的组合区间[17 21 27 33]作为SiPLS优选后的半纤维素特征谱区(波长点为641~680, 801~840, 1 041~1 080和1 281~1 320, 共计160个), 并使用GSA对其进行波长点优选, 算法码长为160, 种群规模为50, 邻域解扰动位数m取10, 其他参数与纤维素波长优选时一致。 算法连续执行10次后, 经计算后确定选中3次以上的波长点(148个)为SiPLS-GSA优选的半纤维素特征波长。
3.2.3 BiPLS-GSA特征波长优选
BiPLS-GSA采用与SiPLS-GSA相同的区间划分方案, 先将光谱划分为13, 16, 20, 26, 31, 39和52个子区间, 再使用BiPLS选取每种子区间数下RMSECV最小的组合区间作为BiPLS优选后谱区, 然后再使用GSA对优选后的谱区进行特征波长点优选。 不同子区间数下优选的纤维素和半纤维素特征谱区如表3所示。
| 表3 纤维素和半纤维素BiPLS优选谱区结果 Table 3 Results of optimized spectral regions for cellulose and hemicellulose by BiPLS |
选取划分26个子区间的最佳组合区间[16 18 19 20 24 25]作为BiPLS优选后的纤维素特征谱区。 对应的波长点为901~960, 1 021~1 200和1 381~1 498, 共计358个。 分析发现, SiPLS和BiPLS优选的纤维素特征谱区存在260个波长点的重复, 占BiPLS优选谱区的72.63%, 说明SiPLS和BiPLS谱区优选具有一致性。 将这358个波长点作为GSA的输入波长进行再次优选, 算法参数设置如下: 码长为358, 种群规模为70, 邻域解扰动位数m取10, 其他参数与SiPLS-GSA一致。 连续执行算法10次后, 经计算确定选中6次以上的波长点(130个)作为BiPLS-GSA优选的纤维素特征波长。
选取划分52个子区间的最佳组合区间[17 20 24 28 36 44]作为BiPLS优选后的半纤维素特征谱区, 对应波长点为481~510, 571~600, 691~720, 811~840, 1 051~1 080和1 291~1 320, 共计180个。 BiPLS和SiPLS优选的半纤维素特征谱区同样具有一致性。 使用GSA对BiPLS优选后谱区进行波长点优选时, 算法码长为160, 种群规模为50, 其他参数与纤维素波长优选时一致。 算法连续执行10次后, 计算确定选中3次以上的波长点(153个)作为BiPLS-GSA优选的半纤维素特征波长。
以Full-GSA, SiPLS-GSA和BiPLS-GSA优选后的特征波长点作为PLS回归模型的输入, 建立秸秆纤维素和半纤维素含量定量分析模型, 并与GA算法对全谱(记为Full-GA)、 SiPLS优选后谱区(记为SiPLS-GA)和BiPLS优选后谱区(记为BiPLS-GA)进行特征波长优选的结果进行对比, 其结果如表4所示。
| 表4 不同波长优选方法评价指标 Table 4 Evaluation indicators of wavelength optimization for different methods |
由表4可知, 各种波长优选方法下, GSA优选结果的预测性能都高于GA; GSA优选的纤维素回归模型的
由SiPLS和BiPLS回归模型的评价参数可知: SiPLS和BiPLS依据RMSECV选择多个组合区间作为优选谱区, 在一定程度上体现了纤维素和半纤维素特征波长点的分布特性, 两种方法的回归模型性能都高于PLS, 其中半纤维素采用SiPLS优选的谱区回归性能最佳, 而纤维素采用SiPLS和BiPLS优选的谱区回归性能相差不大。
由Full-GSA, SiPLS-GSA和BiPLS-GSA优选后的特征波长回归模型评价参数可知, 通过GSA可以在现有波长优选方法的基础上进行有效的再次优化, 去掉无关波长点, 提高回归模型的性能。 在使用GSA对全谱及SiPLS和BiPLS优选后谱区进行特征波长点优选时, 设置算法参数非常重要。 经反复评测发现, Full-GSA中全谱的码长为1 557, 种群规模设置为码长的1/5时, 能够有效兼顾搜索速度和寻优精度, 整体性能较好。 因种群规模较大, 为节省运行时间, 可适当减小遗传代数和降温系数。 而在执行SiPLS-GSA和BiPLS-GSA时, 因码长较小, 种群规模也较少, 可以通过增大遗传代数和降温系数的方式来提高算法的进化次数、 减缓适应度函数值的变化速度, 进而有效提高算法的寻优性能。
Full-GSA能够充分发挥GSA算法全局寻优的特性, 可以通过加大搜索次数的方式来解决随机性问题, 适用于NIRS特征波长的优选。 但以全谱波长点为码长执行GSA算法耗时较长, 在选定参数下, 采用10折交叉验证时执行一次算法约需8.27 h(硬件配置: CPU为AMD A6-7310 2.0 GHZ, 内存4 GB), 而且还需要考虑码长太长引起解空间发散的问题。 结合SiPLS和BiPLS在特征谱区优选方面的优势, 采用GSA对优选后谱区进行特征波长点优选, 能够在兼顾波长优选性能的同时有效减少搜索时间。 采用相同硬件配置下, 按本区间划分方法执行SiPLS和BiPLS优选特征谱区的时间分别为5.35和0.14 h; SiPLS和BiPLS优选的纤维素特征谱区波长点分别为388和358, 然后分别再执行一次GSA二次优选算法的时间约为0.91和0.69 h; SiPLS和BiPLS优选的半纤维素特征谱区波长点分别为160和180, 然后分别再执行一次GSA二次优选算法的时间约为0.39和0.48 h。 多次执行GSA优选算法时, SiPLS-GSA和BiPLS-GSA的搜索时间明显少于Full-GSA。 但SiPLS和BiPLS却限定了GSA搜索的波长点范围, 在一定程度上影响了GSA在光谱空间上的全局寻优能力。 因此, 在解决实际问题时, 需综合评定Full-GSA, SiPLS-GSA和BiPLS-GSA三种方法的性能, 以确定最佳特征波长优选方案。
以Full-GSA-16作为纤维素的波长优选方案, 以SiPLS-GSA作为半纤维素的波长优选方案, 以优选后的特征波长进行PLS回归模型性能评测, 其结果如图4所示。
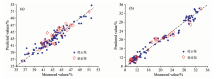 | 图4 纤维素和半纤维素实测值与预测值分布 (a): 纤维素; (b): 半纤维素Fig.4 Distribution of measured and predicted values for cellulose and hemicellulose (a): Cellulose; (b): Hemicellulose |
由图4可知, 纤维素和半纤维素含量的实测值与预测值点基本呈对角线分布, 经检验发现各参数的预测值与实测值无显著性差异。 纤维素和半纤维素回归模型的
基于结合温度参数设计适应度函数的策略构建的GSA具有良好的全局搜索性能, 适用于玉米秸秆纤维素和半纤维素含量NIRS特征波长优选。 GSA以光谱波长点为染色体基因的编码方案适用于NIRS全谱的特征波长优选, 更适用于SiPLS和BiPLS优选后谱区的特征波长优选, 能够有效实现优选后谱区的波长点优选。 通过波长优选, 不仅参与建模的波长点数量显著减少, 而且PLS回归模型的性能得到显著提升, 预测精度更高。 提出的NIRS波长优选方法, 能够有效实现预处理后玉米秸秆纤维素和半纤维素含量的快速、 准确检测。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|