作者简介: 刘思彤, 1998年生, 北京工商大学化学与材料工程学院硕士研究生 e-mail: Liusitong@st.btbu.edu.cn
簇状规则间隔短回文重复序列(CRISPR and CRISPR-associated system, CRISPR-Cas系统)是在大多数细菌和古菌中发现的一种获得性免疫系统, 由Cas酶和引导RNA (guide RNA, gRNA)组成, 根据gRNA序列的特异性识别并剪切靶标DNA或RNA。 近年来, 得益于CRISPR-Cas系统优异的酶切活性, 建立了多种生物传感技术(biosensor), 光学信号传感策略简单、 便携, 广泛应用于科学研究和实际应用中。 详细总结了近五年来基于CRISPR-Cas系统的各种光学传感策略的基本原理以及代表性成果和应用。 同时, 也对当前的应用前景和挑战进行展望。
The clustered regularly interspaced short palindromic repeats (CRISPR and CRISPR-associated system, CRISPR-Cas system) is an acquired immune system found in most bacteria and archaea, consisting of Cas enzyme and guide RNA (guide RNA, gRNA), which recognizes and shears target DNA or RNA based on the specificity of gRNA sequence. In recent years, thanks to the excellent enzymatic activity of the CRISPR-Cas system, a variety of biosensing technologies (biosensor) have been established, and optical signal sensing strategies are simple, portable, and widely used in scientific research and practical applications. We summarized in detail the basic principles and representative results and applications of various optical sensing strategies based on the CRISPR-Cas system in the last five years. Also, we provide an outlook on the current application prospects and challenges.
簇状规则间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)和CRISPR-associated (Cas)系统是在大多数细菌和古菌中发现的一种获得性免疫系统[1, 2, 3]。 按照Cas蛋白组成, CRISPR-Cas系统主要分为两类: Class 1和Class 2, 根据Cas蛋白的结构和序列又可分为不同亚型, Class 1是由多亚基组成的多蛋白效应复合物, 包括Ⅰ 、 Ⅲ 和Ⅳ 型。 Class 2是单个效应蛋白, 包括了Ⅱ 型中的Cas9、 Ⅴ 型的Cas12a和Ⅵ 型的Cas13a等(图1)。 其中, Cas9具有核酸内切酶活性, 两个酶切活性结构域HNH和RuvC, 在两种RNA的引导下, 对含有原间隔序列邻近基序(Protospacer adjacent motif, PAM)的目标双链DNA(dsDNA)进行特异性识别并切割[4]。 Jennifer Doudna等将两种RNA整合为单向导sgRNA, 使其便于使用[5, 6]。 与Cas9不同, Ⅴ 型的Cas12a具有类似RuvC核酸酶结构域, 特异性别目标双链DNA(dsDNA)后会激活反式切割活性(Trans-cleavage activity), 继续非特异切割其他单链DNA(ssDNA)或RNA[7]。 Ⅵ 型的Cas13(Cas13a、 Cas13b 和Cas13c)包含HEPN结构域, 具有核糖核酸酶活性, 特异性识别靶标RNA后会非特异性切割周围的RNA。 近年来, CRISPR-Cas系统被广泛应用于各个领域中, Cas9对核酸的特异性识别作用使其成为理想的核酸检测工具, 结合核酸扩增法和特定的信号传感方式, 如荧光法, 可以实现高灵敏检测。 Cas12、 13和14的反式切割活性为核酸分子诊断提供了更多的可能性, 当这类Cas蛋白在gRNA的引导下与靶序列结合后, 形成三元复合物激发非特异性切割活性, 可以降解体系中存在的任何单链DNA或RNA, 当带有荧光团和猝灭基团的短链DNA或RNA加入体系时, Cas蛋白对靶序列进行识别并切割报告探针, 使荧光团远离猝灭基团而使荧光恢复[8, 9, 10, 11, 12]。 CRISPR-Cas系统已与一系列信号读出方式结合设计了多种生物传感器, 用于不同靶标的分析检测, 广泛应用于生命科学各个领域。
基于CRISPR-Cas的生物传感器能够以多种形式进行信号输出, 其中光学信号传感扮演了重要的角色。 光学信号传感根据物质吸收或发射的电磁波而建立起来的一类分析化学方法, 目前研究报道的CRISPR-Cas系统的光学信号传感包括荧光、 比色、 化学发光法等。 结合本研究进展, 主要阐述当前CRISPR-Cas系统生物传感器的光学信号传感方式的原理及特点, 总结了不同类型光学检测策略的探针分析性能, 如表1, 助力CRSPR-Cas系统在生物安全检测以及生物学研究等领域发挥更大的优势和作用。
| 表1 不同类型光学检测策略探针分析性能比较 Table 1 Comparison of analytical performance of different types of optical probes |
荧光分析方法是根据荧光团的光致发光特性所建立的传感方法, 具有灵敏度高、 操作简单、 分析速度快、 选择性好等优势, 能够提供激发光谱、 发射光谱、 发光寿命、 量子产率和各向异性等诸多信息。 荧光信号读出方式已广泛应用于CRISPR-Cas系统中, 包含了荧光强度信号和荧光数字信号。 荧光强度表示一段时间内大量荧光分子的平均行为, 是一种模拟信号的系综平均检测模式, 待测物质越多, 荧光强度信号越强。 但是, 这种模拟信号的检测模式使单个生物分子的行为平均化, 无法对单个信号源或分子进行系统研究。 荧光数字信号通过统计信号源的数量与目标物的浓度或质量之间建立联系, 基于信号有无开关统计信号数量, 即检测体积内含有超过泊松噪音极限数量的分子, 进而将分析物的浓度以数量的形式表达出来。
荧光团标记的ssDNA与可猝灭荧光的物质组合形成荧光信号开关来设计荧光强度信号探针。 Sun等使用MOF结构(UiO66)开发了一种SDA-RCA扩增方法, 用于大肠杆菌的荧光检测[20]。 荧光染料可与dsDNA特异性结合, 如溴化乙锭和SYBR Green I等, 结合到dsDNA骨架上, 与游离在溶液中相比, 其荧光强度明显增强。 Huang等利用SYBR Green I进行非共价DNA结合, 建立了CRISPR-Cas9诱导指数扩增方法(CAS-EXPAR), 结合实时荧光强度分析, CAS-EXPAR可在1 h内实现灵敏的DNA检测[21]。
荧光共振能量转移(Fö rster resonance energy transfer, fluorescence resonance energy transfer, FRET)是指当一个荧光分子(供体)的荧光光谱与另一个荧光分子(受体)的激发光谱相重叠时, 供体发射的荧光能激发受体发出荧光, 同时供体的荧光强度减弱。 FRET与供、 受体分子的空间距离紧密相关, 一般为7~10 nm时即可发生FRET, 随着距离延长, FRET呈明显减弱[22, 23]。 基于Cas12a、 Cas13和Cas14系统反式切割活性的荧光报告探针多采用FRET原理而设计, 即在探针的两端分别修饰荧光团和猝灭基团[24], 靶标激活Cas蛋白反式切割活性后, 对荧光报告探针进行非特异性切割, 荧光恢复, 荧光强度与靶标浓度呈正比[25]。
单链荧光报告探针, 即ssDNA或ssRNA荧光报告探针, 具有构造简单, 易于设计和可控性高等优点。 Wang等利用Cas12a的反式切割活性, 以荧光ssDNA报告(HEX-N12-BHQ1)为探针, 开发了一小时低成本多用途高效检测系统HOLMES(an one-hour low-cost multipurpose highly efficient system)可用于目标DNA和RNA的快速检测[26]。 在HOLMES中, Cas12a和crRNA组成的二元复合物会与靶标DNA结合, 激活反式切割活性, 非特异性切割系统中ssDNA荧光报告探针, 使荧光基团与猝灭基团距离变远, 进而荧光恢复。 通过测定荧光强度, 可最低检测0.1 nmol· L-1靶标; 结合PCR, 检测限可降低至10 amol· L-1。
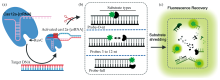
单链荧光报告探针与CRISPR的结合应用已非常广泛, 但是基于双链荧光报告探针却鲜有报道。 Mehmet V Yigit等证明了激活的CRISPR-Cas12a不但可以对ssDNA进行非特异性切割, 也可以对双链进行非特异性切割[27], 依此设计了基于Cas12a反式切割活性的双链荧光报告探针(如图2)。 通过改变发光基团和猝灭基团之间距离, 通过测定荧光强度信号比较Cas12a对于不同的dsDNA探针的反式切割情况, 证明了无论是否带有经典ssDNA片段(TTATT)缺口, Cas12a均可非特异性切割dsDNA, 且不带有缺口的dsDNA探针具有较低的背景和更快的荧光恢复率优点[如图2(b, c)]。
 | 图2 CRISPR-Cas12a切割双链荧光报告分子示意图[27]Fig.2 Schematic representation of CRISPR-Cas12a cleaved doucle-stranded fluorescent reporter molecules[27] |
富含鸟嘌呤(Guanine)的DNA序列能够通过Hoogsteen氢键折叠成G四链体结构(G-quadruplexes)。 在G四链体结构两端修饰荧光团和猝灭基团, 可用作CRISPR反式切割的荧光探针[28]。 Li等分别在人端粒G四链体((TTAGGG)4)的5’ 端和3’ 端标记FAM和TAMRA, 证明了Cas12a能够非特异性切割G四结构, 通过FRET, CD、 凝胶电泳和NMR等技术进行验证, 为推进CRISPR-Cas12a系统和G四链体荧光报告探针在生物传感和生物化学中的应用提供了新途径。 由于G四链体结构稳定性高, 需要较长时间(长达几个小时)才能裂解。 G三链体(G-triplex)由富含三段连续G碱基的富G核酸序列组装而成, 与G四链体具有相似的生物学功能。 进一步研究了Cas12a对G三链体探针的反式切割活性, 证明了Cas12a在10 min内就可以对G3结构进行反式切割, 且其切割速率远远高于G4结构[15]。 基于此设计Cas12a 和G三链体荧光生物传感器, 对未扩增和扩增的HPV16的检测灵敏度分别为50 pmol· L-1和0.1 amol· L-1, 比基于ssDNA荧光报告探针的Cas12a检测系统提高了20倍。
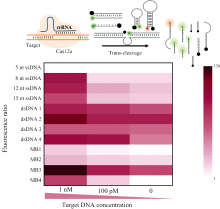
分子信标(molecular beacon, MB)是一种荧光标记的寡核苷酸链, 特殊的发夹结构使其具有背景信号低、 灵敏度高和特异性强的优势, 已广泛应用于分子生物学中, 但较少用于CRISPR系统的荧光分析检测中。 以Cas12a为例, 设计了不同类型的分子信标, 将MB与ssDNA、 dsDNA荧光报告探针进行了对比, 证明了Cas12a的反式切割不仅限于ssDNA、 dsDNA, 还可以扩展到MB[16]。 在相同的实验条件下, 四种不同长度的ssDNA荧光报告探针(长度分别为5、 8、 12和15 nt)最低可以检测到100 pmol· L-1的靶标; 四种dsDNA荧光报告探针, 最低可以检测到100 pmol· L-1, 而MB荧光报告探针, 灵敏度可以提高至 20 pmol· L-1。 在优化了反应条件(包含FRET对、 反应温度、 报告探针浓度以及Cas12a 与crRNA浓度的比例)之后, 发现标记Texas Red和BHQ2的MB荧光报告探针在无扩增的情况下, 最低可以检测到100 fmol· L-1靶标, 比ssDNA和dsDNA荧光报告探针灵敏度提高了3个数量级, 依此开发了一种高灵敏度的SARS-CoV-2检测方法, 选取SARS-CoV-2基因组图谱中13 328~13 437的ORF1a基因作为模型靶点, 以冠状病毒GX/P2V代替SARS-CoV-2进行实际样品检测, 检测限低至27 copies· mL-1, 在临床分子诊断中具有巨大应用前景。
上述荧光强度信号是一种模拟信号的系综平均的检测模式, 荧光数字信号是将反应体系等量的分装成上万个微反应单元, 每一份可能是pL至fL数量级的体积, 每个微反应单元中包含的分子数服从泊松分布, 每个微反应单元内的目标分子进行单独的扩增反应产生荧光亮点信号, 在显微镜下对荧光亮点的数量进行统计, 实现定量检测。 目前CRISPR-Cas分子诊断仍然需要单独的预扩增步骤, 由此产生的多步骤扩增和分析方法使其难以进行荧光数字检测。
Park等报道了荧光数字化信号读出的CRISPR/Cas一锅法(digitization-enhanced CRISPR/Cas-assisted one-pot virus detection, deCOViD), 并用于SARS-CoV-2检测中[30]。 利用商用微流控数字芯片, 将RT-RPA预扩增的Cas12a反应分散到0.7 nL反应小孔中, 将靶标RNA通过RT-RPA扩增成DNA, 激活Cas12a-crRNA的反式切割活性, 继续切割ssDNA荧光报告探针产生荧光, 在反应小孔内每个拷贝的靶标快速扩增产生信号, deCOViD具有高信号背景比、 宽动态范围和高灵敏度, 可以实现15 min内定性检测, 30 min内定量检测, 为推进 CRISPR/Cas 辅助核酸检测和对抗COVID-19大流行及其他疾病开辟了新途径。 Liu提出了一种数字化热启动CRISPR(dWS CRISPR)分析方法, 用于灵敏定量检测SARS-CoV-2[31]。 他们利用商用QuantStudio 3D数字芯片, 将dWS-CRISPR反应混合物分散到0.7 nL的反应小孔中, SARS-CoV-2 RNA靶标在每个反应微孔发生dWS-CRISPR反应, 在52 ℃孵育时产生强烈的绿色荧光(阳性点), 而没有靶标的反应微孔不产生(阴性点)。 dWS CRISPR在50~55 ℃时有效启动, 从而防止在室温下过早的靶点扩增, 并实现核酸的精确数字量化, dWS-CRISPR与之前相比, 可以无需提取RNA直接检测热处理后唾液样本中的SARS-CoV-2。
基于芯片的数字化检测模式可以实现高灵敏多重检测, 在传染性病原体常规监测和综合诊断具有重要应用价值, Ackerman等利用数字化Cas13发展了一种大规模多重核酸检测方法(CARMEN)[32], 打破了上述两种数字化CRISPR只能进行单一目标物诊断的弊端。 该微孔阵列芯片由聚二甲基硅氧烷(PDMS)制成, 具有大量微孔, 每个核酸样本通过RPA或PCR扩增, 并与特定荧光编码结合实现信号读出, 能够在单个阵列上对超过4 500个靶标进行稳定测试, 同时区分169种人类相关病毒, 可用于病原体高通量检测。
相比其他光学检测方法, 荧光信号传感方式具有其独特的优点, 也在向着更便携, 更简单的方向发展。 但与此同时, 它仍然需要专业的光学仪器的协助和专业人员的操作才可以完成检测。
比色法通过贵金属纳米颗粒聚集前后吸收光谱的变化来实现对目标物的测定, 它最大的优势在于可以实现裸眼检测, 对仪器设备要求低, 适用于非专业条件下的快速定性与半定量检测。
Yuan等利用Cas12a/crRNA对dsDNA特异性识别和Cas13a/crRNA对RNA的特异性识别, 分别激活其反式切割ssDNA和ssRNA的性质, 产生DNA功能化的 AuNPs聚集, 溶液颜色改变实现裸眼检测[17]。 同年, Raffaele Velotta采用SARS-CoV-2诱导AuNPs相互作用的比色生物传感器用于检测鼻咽拭子中病毒颗粒[33]。 当含有病毒粒子的溶液混合时, 携带SARS-CoV-2的三种表面蛋白的抗体功能化的金纳米粒子的消光谱在几分钟内发生红移, 检测限接近实时荧光PCR。
相对于上述需要进行扩增的比色信号传感器, Choi课题组在2021年开发了基于CRISPR-Cas12a的无核酸扩增的荧光和比色双模式生物传感器, 他们利用DNA功能化金纳米颗粒通过金属增强荧光(MEF)原理检测游离DNA(cell-free DNA, cfDNA)。 当cfDNA激活CRISPR-Cas12a复合体后, 非特异性切割AuNPs和荧光团之间的ssDNA, 金纳米颗粒发生颜色从紫色到红紫色的变化[34]。 在该研究中, 作者引入了两个大小不同的AuNPs(20和60 nm), 并且由一个7 nm长的dsDNA和一个9 nm长的ssDNA连接, 异硫氰酸荧光素(FITC)标记在dsDNA一端, 由于距离接近60 nm 的AuNPs而保持猝灭状态。 有靶标存在时, 激活了Cas12a的反式切割活性, 将两金纳米之间的连接链切断, FITC远离60 nm的AuNPs依然与20 nm的AuNPs保持连接, 由荧光恢复状态转变为荧光增强状态, 溶液颜色由紫色转变为紫红色, 通过裸眼定性检测, 通过荧光信号实现定量检测, 无需进行核酸扩增, 检测限可以达到飞摩尔浓度级别。
不同纵横比的金纳米棒(GNR)具有独特的横向表面等离子体共振(TSPR)和纵向表面等离子体共振(LSPR)模式, 表现出不同的颜色。 这些特性使得GNR在显色反应中具有良好的显色性能。 Xu课题组利用转化酶-葡萄糖氧化酶级联反应引起GNR变色和Fenton反应读出信号[35]。 靶标DNA在 crRNA引导下激活 Cas12a系统的反式切割活性, 切断了功能磁珠和转化酶之间的ssDNA, 磁分离后上清液中释放的转化酶进行级联反应催化蔗糖水解, 生成的葡萄糖立即被GOx氧化生成H2O2。 在酸性环境下, H2O2通过Fe2+诱导的Fenton反应迅速转化为活性中间体(· OH)。 · OH主要沿纵轴方向刻蚀GNR, 所产生的长径比明显变化, 从而改变溶液颜色。 因此最终的颜色可以有效地与目标DNA的浓度相关联, 并且可以通过裸眼轻易地区分出来。 更重要的是, LSPR位移的峰值波长可用于紫外-可见分光光度法对目标DNA的半定量检测, 在重组酶辅助扩增(recombinase-aided amplification)后, 对目标DNA灵敏度达1 amol· L-1, 与荧光报告探针的灵敏度相当。
比色检测可将目标物识别事件转变为颜色变化, 具有操作简单、 肉眼可见和不需要昂贵或复杂的仪器等优势, 并逐步向一体化、 高灵敏方向发展。
侧流层析法(lateral flow dipstick, LFD)是检测病原体、 小分子、 癌症和基因分型最常用的方法之一, 研究者将CRISPR-Cas系统与侧流层析试纸结合, 已发展出多种简单便携的生物传感器[36, 37, 38, 39, 40], 因其技术成熟, 操作简便, 适用于居家核酸或者抗原检测产品开发, 具有极其广泛的应用前景。
侧流层析试纸条一般由样品垫、 结合垫、 硝酸纤维素膜和吸附垫组成, 由于试纸条的毛细作用, 将待测样品滴到样品垫后, 样品将随液体在试纸条上向前移动。 在结合垫处靶标样本和显色物质, 如纳米金标记的抗体结合形成复合物继续向前移动。 当复合物移动至抗体标记的检测线处, 抗原和抗体特异性结合, 检测线显色, 表明待测靶标存在; 继续向前移动, 显色物质标记的抗体和对照线处的抗体特异性结合, 对照线显色, 表明侧流试层析纸条正常工作。 若检测线显色而对照线不显色或检测线对照线均不显色, 则检测结果无效。 若检测线和对照线均显色, 则检测结果为阳性; 如检测线不显色, 对照线显色, 则检测结果为阴性。
AuNPs-LF(gold nanoparticles-based lateral-Flow, AuNP-LF)检测是一种快速发展的技术, 它利用AuNPs作为免疫反应的信号报告探针。 与其他报告标签探针相比, AuNPs具有良好的生物相容性和较小的生物毒性, 在体外和体内诊断方面具有极大的优势。 迄今为止, AuNP-LF检测已被广泛用于检测各种目标物, 包括感染性病毒、 蛋白质、 药物和其他病原体[41]。 Benjamin等发展了一种half-strip形式的侧流层析试纸条, 即不使用样品垫和结合垫, 将样本和纳米金纳米颗粒标记的抗体混合, 而后插入硝酸纤维素膜和吸附垫组装而成的试纸条直接检测[42]。 该方法检测SARS-CoV-2 N蛋白的检测限为0.65 ng· mL-1。 Xie等利用核壳结构磁性量子点标签构建了一种双模式荧光侧流层析检测方法, 可以同时检测SARS-CoV-2的S蛋白和N蛋白[43]。 磁性量子点标签不仅可以高灵敏显色, 而且可以富集病毒颗粒, 该方法全程检测时间为10 min, 靶向SARS-CoV-2的S蛋白和N蛋白的检测限分别为1和0.5 pg· mL-1检测限有了很大的提高。 Garrit等在侧流层析试纸条检测的基础上, 采用同步辐射X射线荧光成像(synchrotron X-ray fluorescence imaging)技术读取检测结果, 检测限较普通的肉眼读取方式提高100倍[44]。
与上述不同的是, 纳米硒纳米粒子(SeNPs)作为LFIAs(lateral flow immunochromatographic assay)的定性探针, 具有更高的灵敏度和稳定性, 与胶体金相比, 硒纳米粒价格更低廉。 Ma课题组提出了一种基于纳米硒颗粒修饰的SARS-CoV-2核蛋白的侧流层析免疫检测试剂盒, 该试剂盒可以检测人血清中抗SARS-CoV-2 IgM和抗SARS-CoV-2 IgG, 结果可在10 min内裸眼读取[45]。 多种信号读出方式的联合使用可以将检测效率大大提高。
基于侧流层析试纸条检测方法在检测速度和检测成本方面具有很大的优势, 其操作简单, 快捷高效, 安全可靠。 2018年, 张锋团队开发了基于Cas13的分子诊断技术(specific high-sensitivity enzymatic reporter UnLOCKing, SHERLOCK)[46], 利用Cas13a对RNA特异性识别后反式切割荧光团和猝灭基团修饰ssRNA探针, 利用荧光强度信号实现对多种病毒RNA的定性检测。 该团队通过加入Csm6开发了SHERLOCK v2[19], 将检测灵敏度提高了3.5倍, 通过不同颜色的荧光报告探针, 实现多重靶标检测。 通过商业化的试纸条, 裸眼直接观察颜色来判读结果, 且不需要特殊仪器。 ssDNA两端修饰FAM和生物素, 无靶标时, ssDNA没有被切断, ssDNA的FAM端与抗FAM抗体修饰的金纳米结合后流向C线, ssDNA的biotin端与C线的链霉亲和素结合, 无法到达Test线, 无信号。 当有靶标存在时, CRISPR/CrRNA的激活反式切割活性被激活, 非特异性切割ssDNA, 切断后的ssDNA的FAM端与抗FAM抗体修饰的金纳米结合后流过C线后, 无法被固定, 继续流向T 线, 显示信号。 部分ssDNA未被切断, ssDNA的FAM端与抗FAM抗体修饰的金纳米结合后流向C线, ssDNA的biotin端与C 线的链霉亲和素结合, 作为对照。 2020年初, 该团队将CRISPR/Cas13的SHERLOCK技术用于新型冠状病毒检测, 以侧流层析试纸条信号读出, 开发了SHERLOCKTM CRISPR SARS-CoV-2 检测试剂盒, 在1h内完成检测, 适合居家新冠病毒核酸大规模初筛检测。
侧流层析试纸条信号传感方式综合了色谱优异的分离能力和传统免疫分析的高特异性和高灵敏性的优点, 已成为现场即时检测的一种重要工具。 采用目视法读取定性或半定量结果, 检测灵敏度和准确度显然不能满足当下对分析物高灵敏定量检测的要求。
现今血糖仪和智能手机几乎是每个家庭必备的设备, 运用血糖仪或者智能手机进行检测或者信号的读出无疑是一种非常方便的选择。 Ning等报道了一种RT-RPA CRISPR 的荧光检测系统, 可以精确地量化唾液中存在的SARS-CoV-2, 而无需分离RNA, 并将该检测方法应用于智能手机读取芯片, 用于即时使用, 并评估了其分析性能, 智能手机读取的CRISPR-FDS分析结果与RT-qPCR结果之间的一致, 适用于超灵敏的现场COVID-19筛查工作[47]。 Pimkhuan Hannanta-anan课题组介绍了一种基于智能手机设备以及机器学习软件[48]。 该设备由一个3D打印外壳和低成本的光学组件组成, 可以激发荧光报告探针, 并选择性地将荧光信号传输到智能手机上实现信号读出。 凝胶电泳分析也可以用于CRISPR-Cas的终点信号读出, Mehmet V Yigit等利用杂交链式反应(hybridization chain reaction, HCR)与Cas12a结合, 以烟草卷梢病毒(TCSV)和乙型肝炎病毒(HepBV)为模型靶标, Cas12a识别靶标后激活反式切割活性, 切割了体系中的引物链, 使得HCR无法进行, 从凝胶电泳上无法看到HCR扩增产物, 利用凝胶电泳终点读出, 凝胶显示引物的条带强度随着目标量的增加而降低, 灵敏度达1.5 fmol, 比单纯的HCR灵敏度提高了100倍[49]。
CRISPR-Cas系统研究正在逐步改变着传统的基因编辑和分子诊断模式, CRISPR-Cas技术具有简单、 特异、 灵敏的特点, 可与多种技术相结合。 基于本课题组的研究进展, 总结了CRISPR-Cas系统构建的光学传感器的信号传感方式原理及特点, 并介绍了当前各课题组的研究进展。 随着科技的发展, 基于CRISPR-Cas系统信号读出正向智能化、 便携式方向发展。 目前绝大多数CRISPR-Cas系统的分析检测依赖预扩增过程, 如何利用信号读出的方式摆脱预扩增步骤, 简化操作是未来的发展方向之一。 高灵敏高通量的检测在新冠病毒等病原体感染性疾病的大规模初筛具有重要意义, 如何利用CRISPR-Cas系统的顺、 反式切割活性设计信号读出, 实现高灵敏高通量的检测也是分析化学追求的目标之一。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|