作者简介: 袁江涛, 1995年生, 宁夏大学食品科学与工程学院硕士研究生 e-mail: histyjt@163.com;郭佳俊, 1996年生, 宁夏大学食品科学与工程学院硕士研究生 e-mail: jiajunguo2023@163.com;袁江涛, 郭佳俊: 并列第一作者
生育酚当量抗氧化能力(TEAC)是评估肌肉内源抗氧化程度的指标之一, 可用于评估亲水化合物的抗氧化活性及清除自由基的能力。 为探究快速检测滩羊肉中TEAC的可行性, 采用可见近红外(Vis/NIR)高光谱成像技术, 建立基于光谱信息融合图像纹理特征(TFS)的TEAC定量预测模型。 将不同部位样本集根据3∶1的比例随机划分成校正集和预测集, 在400~1 000 nm范围内采集反射光谱图像, 提取每个样本图像的感兴趣区域(ROI)以获取原始光谱数据; 采用中值滤波(MF)、 基线校准(Baseline)、 卷积平滑(S-G)和多元散射校正(MSC)四种算法对原始光谱中散射及干扰信息进行校正, 并建立偏最小二乘回归(PLSR)模型, 将光谱数据与TEAC值进行关联。 采用间隔随机蛙跳(IRF)、 变量组合集群分析(VCPA)、 竞争性自适应加权抽样(CARS)和迭代变量子集优化(IVSO)算法提取TEAC浓度的特征波长。 采用灰度共生矩阵(GLCM)算法对肉样的主要TFS依次进行提取。 基于特征光谱及图谱融合信息建立反向传播人工神经网络(BP-ANN)和最小二乘支持向量机(LSSVM)模型, 对滩羊肉中TEAC含量预测并进行对比分析。 结果表明, (1)最优预处理为Baseline建立的PLSR模型, 其 Rc为0.912 1, RMSEC为0.963 5, Rp为0.868 3, RMSEP为1.277 0; (2)采用IRF、 VCPA、 CARS和IVSO分别提取出了71、 9、 22和39个特征波长, 占全光谱的56.8%、 7.2%、 17.6%和31.2%; (3)基于多元特征提取算法建立的BP-ANN和LSSVM模型, 对TEAC含量进行预测时Baseline-IVSO-LSSVM( Rc=0.913 2, RMSEC=0.962 0, Rp=0.864 6, RMSEP=1.288 3)具有最优预测性能; (4)相比于单一的光谱信息模型, TEAC相关的TFS图谱融合模型IVSO-TF1-BP-ANN显示出更好的效果, 其 Rp为0.8916, 较特征波长数据建模提高了0.028 6。
YUAN Jiang-tao and GUO Jia-jun: joint first authors
Trolox-Equivalent Antioxidant Capacity (TEAC) is one of the endogenous antioxidant indexes of muscle, which can be used to determine the antioxidant activity of hydrophilic compounds and free radical scavenging ability. The visible near/infrared (Vis/NIR) hyperspectral imaging technology was used to explore the feasibility of rapid detection of the TEAC in Tan mutton, a quantitative prediction model for TEAC based on spectral information fusion of image texture features (TFS) was built. The samples from different parts were randomly split into calibration set and prediction set according to the ratio of 3∶1. The spectral reflectance images were collected in the range of 400~1 000 nm, and the regions of interest (ROI) were selected to obtain raw spectral data. Four algorithms, including Median Filtering (MF), Baseline, Savitzky-Golay (S-G) and multiplicative scatter correction (MSC), were used to correct the scattering and interference information in the original spectrum, and the Partial Least Squares Regression (PLSR) model was established to correlate spectral data with TEAC values. Representative characteristic spectra of TEAC concentrations were extracted using Interval random frog (IRF), Variable combination population analysis (VCPA), Competitive adaptive reweighted sampling (CARS), and Iteratively variable subset optimization (IVSO) algorithms. The meat's main texture features were extracted sequentially by using the Gray level co-occurrence matrix (GLCM) algorithm. Based on the characteristic spectrum and spectral fusion information, the Back-propagation artificial neural network (BP-ANN) and Least-squares support vector machines (LSSVM) model were established to predict and compare the TEAC content in Tan mutton. The results showed that (1) The PLSR model established by the preprocessed spectra of Baseline was the best with Rc of 0.912 1, RMSEC of 0.963 5, Rp of 0.868 3, RMSEP of 1.277 0; (2) The 71, 9, 22 and 39 characteristic bands based on the original spectral were extracted by IRF, VCPA, CARS and IVSO methods, respectively, accounting for 56.8%, 7.2%, 17.6% and 31.2% of the total bands; (3) Compared with model effects of BP-ANN and LSSVM models in feature variables extraction based on multiple algorithms, the optimal prediction model for TEAC content was Baseline-IVSO-LSSVM ( Rc=0.913 2, RMSEC=0.962 0, Rp=0.864 6, RMSEP=1.288 3); (4) The fusion model IVSO-TF1-BP-ANN showed better results ( Rp=0.891 6) with improving by 0.028 6, compared with model based on the characteristic wavelength.
肉类食品中富含的蛋白质和脂肪极易氧化, 导致肉品色泽、 质地以及风味劣变[1]。 生育酚是一种天然抗氧化剂, 有猝灭自由基的功能[2]。 羊肉中生育酚来源为维生素E的水解产物, 是非酶类内源性抗氧化物质之一。 生育酚当量抗氧化能力(trolox-equivalent antioxidant capacity, TEAC)是可以全面反映肉类内源抗氧化能力的综合性指标之一, 但传统检测方法过程复杂, 效率低, 亟需一种实时、 无损、 高效的检测方法。
可见/近红外(visible/near infra-red, Vis/NIR)高光谱作为近年来应用最为广泛的无损检测技术之一, 可定性和定量分析物质的内部属性及结构特征。 肉类中的不同成分, 如水、 蛋白质、 脂肪酸或脂类等差异是导致光谱不同的主要因素。 这些组分的官能团具有一定化学键(O— H、 C— H、 N— H、 C=O等), 这些键经辐射后激发振动, 即当辐射频率与样品中的特定化学键具有相同振动频率时, 可产生对应的光谱, 从而可对样品的化学组成及结构特性进行分析[3]。 考虑到饲喂滩羊的牧草中许多抗氧化物质如酚类物质、 维生素C、 类胡萝卜素等可沉积于肌肉组织中, 同时光谱图像结合纹理特征(texture features, TFS)信息可提高对与TFS信息相关指标的预测能力[4], 因此在TEAC光谱模型中加入图像的纹理数据是非常必要的, 但尚未见到使用高光谱成像技术监测肉类中TEAC的研究。
以滩羊肉为研究对象, 采用Vis/NIR高光谱成像技术结合图像TFS信息对羊肉中TEAC进行无损检测, 并提高其预测性能, 为实时快速定量检测滩羊肉中TEAC提供参考。
滩羊肉采购于宁夏盐池鑫海食品有限公司。 分别取不同滩羊背最长肌、 前腿肉和后腿肉, 保存在0~4 ℃保温箱中运至实验室, 剔除肉样表面多余的筋膜、 结缔组织和脂肪, 4 ℃冰箱排酸24 h, 分割为30 mm× 20 mm× 10 mm的羊肉样本180个(随机选取6只滩羊三个部位采样, 每部位肉样10份, 真空包装后标记。 按3∶ 1的比例划分为校正集和预测集。
Vis/NIR高光谱成像系统有125个波长, 波长范围为400~1 000 nm(Headwall Photonics公司, 美国)。 该系统由成像光谱仪、 电控位移平台、 4个光纤卤素灯(150 W)、 增强型CCD相机、 计算机和数据采集软件组成。 使用高光谱前应预热30 min, 图像采集前进行黑白校正[5], 肉样用纸巾擦拭表面水分。
参考Anticona[6]的方法并进行修改。 吸取6 mL ABTS· +溶液和50 μ L的肌肉提取液进行混合, 30 ℃下孵育6 min, 用50 μ L超纯水和6 mL ABTS· +溶液混合做空白对照。 在734 nm下测定吸光度, 通过已知浓度的生育酚溶液绘制标准曲线。 结果以μmol· g-1生育酚当量表示。
1.4.1 感兴趣区域提取
使用ENVI 4.8软件, 采用阈值分割法提取感兴趣区域(region of interest, ROI), 设置阈值为0.12, 将样本与背景分开得到二值化图像, 并将整个样本图像(约2500像素)作为感兴趣区域[7]。
1.4.2 预处理方法
原始光谱数据含有暗电流噪声和仪器基线漂移等, 因此需要通过光谱预处理方法来消除和校正干扰无关信息。 其中平滑是消除噪声最常用的方法之一, 通过减少图像噪声对数据点数值的干扰, 提高信噪比。 主要有中值滤波(median filtering, MF)、 基线校准(baseline)、 卷积平滑(savitzky-golay, S-G)。 多元散射校正(multiplicative scatter correction, MSC)作为校正变换类的光谱预处理方法, 不仅能够增强光谱信息的显示, 而且能有效消除样品散射所导致的基线偏移和平移现象, 提高光谱信噪比[8]。 故采用以上四种算法以增强光谱信号并提高模型预测性能。
1.4.3 特征波长选择方法
间隔随机蛙跳(interval random frog, IRF)算法可以根据所定义的策略来更新变量的子集。 在满足迭代时间后, 计算每个波段被选择的概率, 并按降序排列[9]。 变量组合集群分析(variable combination population analysis, VCPA)算法根据模型总体分析, 在可变子集总体构建的模型群体中提取统计信息, 以迭代方式进行最优的变量子集选择[10]。 竞争性自适应加权抽样(competitive adaptive reweighted sampling, CARS)算法将有较大绝对回归系数的波长识别为关键波长, 权重小的波长[11]。 迭代变量子集优化(iteratively variable subset optimization, IVSO)算法具有较高的稳定性, 原因是该算法使用顺序加法和加权二进制矩阵采样的方法, 以竞争方式消除非关键变量信息, 同时降低丢失关键变量的风险[12]。
1.4.4 图像TFS信息的提取
TFS信息表示相邻像素之间的强度关系, 常用于解释和量化图像中的灰度值。 灰度共生矩阵(gray level co-occurrence matrix, GLCM)常用于提取TFS, 具有适应性强、 鲁棒性好的特点。 用于计算图像中像素之间不同灰度组合频率, 以此反映样本外部特征中最重要的指标。 本研究基于主成分分析(principal component analysis, PCA)选择的特征图像, 依次选取羊肉样本信息表达量前三的PC图像在0° 、 45° 、 90° 、 135° 四个方向下的角二矩阵、 对比度、 相关性和熵的特征参数来表示羊肉样本的TFS[13], 分别记为TF1、 TF2、 TF3、 TF4。
1.4.5 模型建立
基于光谱信息和图像纹理信息建立羊肉中TEAC的反向传播人工神经网络(back-propagation artificial neural network, BP-ANN)和最小二乘支持向量机(least squares support vector machines, LSSVM)预测模型。 BP-ANN在前向传播过程中若输出信息的期望值存在误差, 则误差将沿着原始路径重新传播, 通过逐个修改每一层神经元的权重, 以减少误差, 直到结果的输出满足精度要求[14]。 LSSVM是一类基于核的学习方法, 广泛应用于执行非线性多元数据分析[4]。 以校正集相关系数(Rc)、 校正集均方根误差(RMSEC)、 预测集相关系数(Rp)、 和预测集均方根误差(RMSEP)进行评估预测模型性能的可靠性和准确性。
1.4.6 数据处理工具
数据处理软件采用MATLAB R2019 a、 ENVI 4.8和Origin 2018。 数据分析用Intel core I5-10210 CPU, 主频1.6 GHz、 RAM为8 GB、 NVIDIA MX250、 显存为2 GB的计算机。
滩羊肉样品在Vis区域(400~750 nm)和NIR区域(750~1 000 nm)均有谱带。 对样品的平均反射率曲线进行了比较和分析。 从光谱曲线可以看出, 不同部位肉样的平均光谱曲线具有相似的趋势, 但光谱纵向位移有很大的差异。 如图1所示, 不同部位的滩羊肉样品在434、 545、 573、 756和967 nm处均具有吸收带。 在434 nm左右的吸收是由于肉样中血红素辅基中的π — π * 键振动, 这种色素与肉色有关[15]。 545~573 nm范围内的吸收, 与肌红蛋白(氧合肌红蛋白和脱氧肌红蛋白)和肉色的a* 值高度相关[16]。 760 nm附近的吸光度带与O— H拉伸第三倍频或肌红蛋白氧化相关, 970 nm左右的吸收峰与O— H弯曲的第二倍频有关[17]。
为了提高重叠数据的光谱分辨率, 利用四种预处理方法建立PLSR的预测模型。 同时在不同预处理下提取潜在变量(latent variables, LV)以降低数据维度, 去除冗余信息, 同时保留了原始数据中的有效信息。 通过建立PLSR模型评价不同预处理方法的效果, 结果如表1所示, 与TEAC的原始光谱模型效果相比, S-G预处理后的光谱模型性能较低, 可能是由于S-G算法消除了与TEAC含量相关的有效光谱信息。 使用Baseline预处理建立的PLSR模型呈现出最高的相关系数, Rc为0.912 1, Rp为0.868 3, RMSECV为1.407 5。 这可能是Baseline对光谱图像具有较好的滤波作用, 与此同时可以保护信号的边缘数据。 Dixit[18]等在对牛肉成分分析时同样发现经Baseline处理后可获得更清晰的样品特征。 因此, 将Baseline预处理作为TEAC的全样本数据信号, 并进行后续建模。
| 表1 PLSR模型利用不同预处理数据的预测结果 Table 1 Modeling results of PLSR using different pretreatment methods |
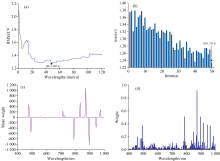
为了在一定程度上提高模型的预测性能, 采用IRF、 VCPA、 CARS和IVSO对全波段光谱进行降维处理, 结果如图2所示。 图2(a)为IRF算法提取特征波长结果, 当组合间隔为48时可使RMSECV达到最小值(1.268 4)。 此时选取前48个组合波段, 共71个波长, 分布在434~607和665~823 nm区域, 占全光谱的56.8%。 图2(b)为应用VCPA算法提取特征波长结果, 在RMSECV达到最小值(1.234 5)时, 此时采样数为50, 共筛选出478、 492、 550、 607、 703、 785、 790、 871和895 nm 9个特征波长, 占全光谱的7.2%。 图2(c)为采用CARS算法提取特征波长的结果, 共筛选出22个特征波长, 占全光谱的17.6%, 分别为434、 454、 458、 478、 492、 559、 579、 603、 607、 670、 713、 727、 785、 790、 828、 833、 867、 871、 895、 924、 943和972 nm。 图2(d)为IVSO算法权重值变化, 通过IVSO算法筛选出39个特征波长, 占全光谱的31.2%, 分别为434、 444~458、 478、 487~497、 506、 550~564、 574、 579、 607、 636、 684、 727、 780~795、 833、 847、 857、 867、 871、 881、 895、 910、 915、 924、 943、 963、 977~987和996 nm。 分析发现478、 492、 607、 785、 790 nm为共有特征波长, 其中492 nm与高铁肌红蛋白有关[19], 而高铁肌红蛋白会受到细胞内氧化还原水平影响, 这可能与TEAC含量存在间接关系。 提取特征波长后, 可以大幅减少图像大小, 特征更加突出, 这在一定程度上会增加非线性关系, 但同时又有助于减轻硬件需求和计算负载[20]。
使用ENVI 4.8软件, 通过正向主成分统计方法对滩羊肉样本光谱图像进行主成分分析, 如图3所示, PC1、 PC2、 PC3累计贡献率大于99%, 其中PC1、 PC2、 PC3表达分别为98.76%、 0.75%、 0.09%, 可见PC1包含了部分信息的同时又能有效去除冗余信息。 因此对表达量最多的PC1分别从θ =0° 、 45° 、 90° 、 135° 角度下对角二矩阵、 对比度、 相关性和熵进行TFS信息提取。 通过对肉样感兴趣区域图像的四个纹理变量计算, 得到一个纹理信息矩阵, 180样本× 4变量。
2.5.1 特征光谱预测模型建立
基于多种特征波长筛选方法(CARS、 VCPA、 IRF和IVSO)建立的TEAC模型的预测效果如表2所示, 当采用BP-ANN建立预测模型时, 虽然CARS-BP-ANN模型具有最高的Rp, 但其校正集Rc仅为0.8189。 因此, IVSO-BP-ANN模型的效果最佳(Rc=0.887 5, RMSEC=1.113 1, Rp=0.863 0, RMSEP=1.126 6); IVSO-LSSVM模型体现出最佳的预测能力(Rc=0.913 2, RMSEC=0.962 0, Rp=0.864 6, RMSEP=1.288 3)。 对比不同特征波长提取算法下的两种模型, 发现LSSVM模型中Rc值整体比BP-ANN模型较高, 且使用IVSO方法显示出更好的预测能力, 说明IVSO算法可有效筛选出与TEAC含量相关的特征波长并去除冗余波段。 因此, IVSO-LSSVM模型预测TEAC性能最佳。
| 表2 BP-ANN和LSSVM模型在不同波长提取方法下的性能 Table 2 Performances of BP-ANN and LSSVM models using different wavelength extraction methods |
2.5.2 光谱数据结合TFS信息预测模型建立
在提取特征波长后, 数据维度大幅度降低, 特征更加突出, 在一定程度上会增加非线性关系, 而BP-ANN和LSSVM预测模型对非线性数据具有较好的预测能力。 根据特征波长建模结果, 使用IVSO算法提取的特征波长与肉样纹理信息融合建立TEAC含量的BP-ANN和LSSVM预测模型如表3所示, 表3给出了对不同TFS信息的校准集和预测集的性能。 结果表明, IVSO-TF1-BP-ANN图谱融合模型稳健性较好, 其Rp=0.891 6, 较特征波段提高0.028 6, 图谱融合模型较单一模型相比提高了预测能力。 纹理是肉样的外部属性, 是颜色灰度空间产生的图像模式, 融合后的模型具有更好的预测能力, 说明融合肉样的外部TFS可行。
| 表3 不同TFS融合BP-ANN和LSSVM模型对TEAC的评价结果 Table 3 Evaluation results of fusion BP-ANN and LSSVM models for TEAC by different TFS |
采用Vis/NIR高光谱技术结合图像纹理信息建立滩羊肉中TEAC预测模型, 主要结论如下:
(1)经不同算法预处理后建立PLSR模型, 发现Baseline-PLSR模型具有最佳性能, 其Rc=0.912 1, RMSEC=0.963 5, RMSECV=1.407 5, Rp=0.868 3, RMSEP=1.277 0。
(2)采用IRF、 VCPA、 CARS和IVSO分别提取出了71、 9、 22和39个代表性特征波长, 占全光谱的56.8%、 7.2%、 17.6%、 31.2%。
(3)评估多种特征波长算法下建立的BP-ANN和LSSVM模型性能, 结果显示: 最优的TEAC预测模型为Baseline-IVSO-LVSSM(Rc=0.913 2, RMSEC=0.962 0, Rp=0.864 6, RMSEP=1.288 3)。
(4)TEAC图谱融合模型IVSO-TF1-BP-ANN显示出较好的效果, 其Rp为0.891 6, 相比于单一的光谱信息模型提高了0.028 6。
采用Vis/NIR高光谱成像技术, 将光谱信息融合图像TFS建立了羊肉样品中TEAC的定量预测模型, 取得了较好的预测效果, 但仍有以下可改进的方面:
(1)可以优化光谱数据的采集和处理方法, 以提高数据的精确性和稳定性。
(2)利用高光谱显微成像方法, 以在细胞和组织水平上对物质进行分析。
(3)虽然光谱与纹理特征相结合对模型判断能力具有一定提升, 但如何从高光谱图像中提取与肉质高度相关的特征并进行融合, 有待进一步研究。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|