作者简介: 康 睿, 1990年生, 江苏省农业科学院助理研究员 e-mail: kangrui@jaas.ac.cn
如何实现对食源性致病微生物的早期快速检测是全球食品安全领域面临的挑战之一。 传统的致病菌生化检测方法虽然准确性高, 但是过程复杂、 耗时漫长, 易错过控制疫情爆发的窗口期。 该研究提出一种基于暗场显微高光谱成像技术的检测方法, 能够借助显微镜技术突破传统光谱成像的灵敏度和分辨率极限, 并利用可见/近红外光谱为单个致病菌细胞添加高分辨率的图像和光谱信息。 以空肠弯曲杆菌、 大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌为检测对象, 采用显微高光谱成像技术进行数字化表征和数据采集, 结合双向长短期记忆网络(Bi-LSTM)算法对各致病菌细胞的图像和光谱数据进行建模分类。 结果显示, 显微尺度的致病菌光谱数据呈现可判别的分布规律, 新采用的Bi-LSTM网络在光谱数据集中表现优异, 在三种致病菌的分类任务中取得了91.0%的平均准确率, 而传统的线性判别分类器(LDA)和支持向量机分类器(PCA-SVM)的平均准确率分别为80.1%和88.5%。 但是, 仅依赖光谱数据进行致病菌种类判别仍然存在较为严重的假阳性问题, 尤其是在大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌的分类中存在错误分类。 图像信息的加入则能够显著改善各分类的识别准确率, 其中Bi-LSTM分类器取得了高达98.1%的准确率, LDA和PCA-SVM均取得了95.3%的准确率。 研究结果表明, 显微高光谱成像技术在食源性致病菌的特异性光谱和图像表征中具有优势, 提出的Bi-LSTM网络能够直接处理高维的图谱特征, 两种技术的融合在食源性致病菌细胞级别的早期检测应用中展现潜力。
The early and rapid detection of food-borne pathogens still challenges global food safety. Although routine tests such as selective agars served as the gold standard for decades, these methods are time consuming, often exceeding the best control time for foodborne bacterial outbreak. A hyperspectral microscopic imaging (HMI) technology coupled with an artificial intelligence algorithm was proposed to detect the common foodborne bacteria. The HMI technology has a natural trait in generating robust high-resolution spatial and spectral characterization at the cellular level. In this study, the Bi-directional long short-term memory (Bi-LSTM) was employed in the classification of Campylobacter jejuni ( C. jejuni), Escherichia coli O157:H7 ( E. coli), Salmonella Typhimurium ( S. Typhimurium) based on morphological and spectral features extracted from single cell hypercubes. Compared with traditional linear discriminant analysis (LDA, 80.1%) and principal components analysis with support vector machine (PCA-SVM, 88.5%) classifiers, our proposed Bi-LSTM achieved the highest accuracy of 91.0% on the spectral dataset. Serious false-positive problems occurred in recognising E. coli and S. Typhimurium. However, with the involvement of morphological features, the discriminability of all classifiers was significantly improved. The proposed Bi-LSTM classifier achieved the highest accuracy of 98.1% based on the morphological-spectral feature dataset, while the LDA and PCA-SVM all achieved an accuracy of 95.3%. Our study demonstrated the applicability of HMI technology for foodborne bacterial cell characterization. Furthermore, with the advantage of Bi-LSTM in instantly processing the high-dimensional spatial-spectral features, the intelligent HMI shows great potential for rapid detection of foodborne pathogens.
食源性致病菌是导致食物中毒和食源性疾病的重要诱因, 严重威胁着消费者的生命健康。 如何实现这类微生物的早期快速检测是全球食品安全面临的挑战之一[1]。 常见的食源性致病菌有鼠伤寒沙门氏菌、 大肠埃希氏菌O157:H7和空肠弯曲杆菌等。 它们体型微小, 以食物为载体潜入人体快速繁殖, 诱发诸如恶心、 呕吐、 急性肠胃炎、 出血性腹泻、 肾或肝衰竭、 甚至癌症等各类疾病。 在2003年至2017年期间, 我国发生食源性疫情共计19 517次, 约有235 754人患病, 107 470人需住院治疗, 1 457人因此丧生。 由各类食源性致病菌引起的案例占比约为44.5%[2]。 为应对日益严峻的食源性致病菌传播风险, 我国自2013年起就出台了《食品中致病菌限量》等标准用于食品安全监管[3]。 然而, 目前作为金标准的分离培养检测法周期较长, 易错过防控食源性疫情爆发的最佳时机。
近年来, 各类快速检测技术发展迅猛, 光学与光谱成像法因其非侵入性检测的优势成为各国的研究热点。 当前用于致病菌检测的光学光谱法主要以菌落或者菌液为研究对象, 如普渡大学的Tang等利用激光散射技术对不同血清型非O157产志贺毒素大肠杆菌的菌落生长分布图像进行识别分类[4]。 Windham等则使用可见/近红外光谱技术对各类致病菌菌落进行光谱建模分析实现种类判定[5]。 虽然宏观尺度的致病菌光谱检测技术的成功令人瞩目, 但是如何在更早期如细胞阶段的致病菌光学或光谱数据采集依然是挑战。 常规光学显微镜技术在透明的细胞观测任务中存在不足, 当前的改良手段是通过添加荧光材料进行标记。 但是, 荧光染料通常具有毒性会导致细胞凋亡, 不易于实验的复现和深入研究。 显微高光谱成像技术成为食源性致病菌细胞检测的一种新型方式, 通过将非侵入性的光谱表征技术延伸至显微尺度, 有助于致病菌活菌细胞的检测和种类识别[6]。 Park等构建了一种基于暗场显微镜的高光谱成像系统, 实现了致病菌细胞微弱信号的捕捉和成像[7]。 Eady等则在实验步骤上进行优化, 通过增加特殊的烘干技巧将致病菌固定, 避免了图像采集时细胞移动导致的一系列问题[8]。 然而, 高分辨率的细胞高光谱图像数据量巨大, 如何实现细胞超立方体图像的高维特征提取和快速分析成为新的难题。
本研究以空肠弯曲杆菌、 大肠埃希氏菌O157:H7、 鼠伤寒沙门氏菌为对象, 利用自行构建的显微高光谱成像系统进行致病菌单个细胞的超立方体数据采集, 提取细胞的图像和光谱特征, 并尝试构建一种基于神经网络的智能算法实现致病菌高光谱数据的快速分析。
试验采用了三种常见的食源性致病菌, 分别为空肠弯曲杆菌(Campylobacter jejuni, C. jejuni)(ATCC 33291)、 大肠埃希氏菌O157:H7 (Escherichia coli, E. coli)(ATCC 700728)和鼠伤寒沙门氏菌(Salmonella Typhimurium, S. Typhimurium)(ATCC 14028)。 在试验前, 纯种菌株存放在-80 ℃的细菌库中。 根据每次试验需要, 将菌株从冰箱中取出并选择各自的生长培养基进行接种。 孵育时间、 温度和培养基的选择参考Difco & BBL微生物培养手册[9]。 试验步骤主要参照Park等的研究[10], 使用接种环从琼脂板上挑出各致病菌菌落, 并接种到100 μ L纯水中摇匀, 然后吸出3 μ L细菌悬浮液置于载玻片的中央, 并利用生物安全柜进行15分钟通风干燥制样。 随后, 在细菌载玻片中央滴入约0.8 μ L无菌水, 并用无色透明的玻璃盖玻片压住, 挤出多余气泡并在盖玻片顶部滴入少许香柏油, 便于显微高光谱成像系统进行观察和图像采集。
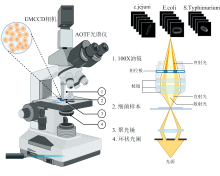
实验用显微高光谱成像系统如图1所示, 主要由Eclipse e80i直立显微镜(日本尼康), AOTF光谱仪(英国克罗姆, 光谱范围450~800 nm, 间隔4 nm), 高性能的16位EMCCD相机(北爱尔兰安道尔科技)以及卤钨灯(21 V, 150 W, 德国欧司朗)组成。 显微镜主体主要用于观察致病菌细胞, 光路传输方式参照暗场相差显微镜光路配置, 因此采集获得的细胞背景图像为暗。 AOTF光谱仪基于声光衍射原理, 能够通过调节不同频率的声波促使声光晶体材质进行特定波长滤波, 为后续CCD相机的图像采集准备。 EMCCD相机的采集参数调至最佳, 曝光时间设定为250 ms, 增益为15%。 通过移动载物台调整显微镜视场捕捉不同位置细胞的图像, 最终每类致病菌采集获得50张高光谱超立方体数据, 共计150(3× 50)张, 部分致病菌数据可在相关平台公开获得[8]。
数据预处理主要包括光谱数据和形态特征数据处理两部分。 由于每张高光谱图像中的细胞分布个数存在差异, 最终实验收集并挑选出各类致病菌细胞共计3 000个, 其中空肠弯曲杆菌、 大肠埃希氏菌O157:H7、 鼠伤寒沙门氏菌样本光谱各1 000个, 整理形成数据集。 光谱数据预处理采用多元散射校正(multiplicative scatter correction, MSC)算法消除光谱基线漂移等散射效应, 使同类细菌的光谱更具特异性。 其中, MSC算法实施步骤为先计算光谱矩阵A的均值光谱
细胞形态数据集则从在546 nm波段从高光谱数据中抽取细胞图像(此波段细胞成像最清晰), 并按照光谱数据集的编号顺序进行保存, 保证同一个细胞的光谱与图像数据编号一致。 细胞图像的形态特征采用自行编写的Python程序提取, 主要使用scikit-learn工具包提取细胞的面积、 周长、 长轴长、 短轴长等形态信息, 详细特征参数如表1所示。
| 表1 细胞形态特征汇总 Table 1 Morphological features of the bacteria cell |
对于形态数据集, 将首先使用标准归一化(Standard normal variate, SNV)的方式对形态数据以行为中心进行运算, 从而实现每行形态特征的中心化缩放, 矩阵运算方法如式(4)所示。
式(4)中,
光谱数据集和图谱数据集分别按照7∶ 2∶ 1的比例划分为训练集(700× 3)、 验证集(200× 3)和测试集(100× 3)。 模型评价指标采用准确率(Acc)、 精确率(Pre)、 F1值和Kappa系数(K), 它们的计算分别如式(5)— 式(8)所示。
其中, 准确率、 精确率和F1公式中的TP、 TN、 FP和FN分别表示真阳性、 真阴性、 假阳性和假阴性样本数, K值则用于模型的一致性评估, Pe表示偶然一致性, 可以通过对混淆矩阵进行运算获得。 实验中所有评价指标的公式运算均通过Python编程实现。 对各模型在图像、 光谱和图谱数据集的分类表现进行分析, 并探索利用图谱模型进行致病菌分类的可行性。
智能算法采用双向长短式记忆网络(Bi-directional long short-term memory, Bi-LSTM), 其主要通过正反向传播的LSTM网络组合一起实现, 对于一维光谱数据分析具有优势。 Bi-LSTM中的内部节点与LSTM网络相同, 包括输入门、 遗忘门和输出门, 主要结构如图2所示。
在每个单元的顶部, 新单元的状态Ct由ft(遗忘门), Ct-1(旧单元状态), it(输入门)和
其中, xt表示输入数据, ht-1表示旧隐含节点的数值, W表示权重, b表示偏差。 在输入门的处理过程中, 网络通过使用tanh层来不断更新
通过利用这种特定的单元结构, LSTM网络能够对任意光谱间隔内的数据流进行解析。 Bi-LSTM则在传统的LSTM前向传播的基础上添加反向层, 通过对前向层和后向层的隐藏向量进行堆叠实现输出, 具体堆叠方式如图3所示。
其中, 前向层隐藏向量
在建模分析环节, 采用光谱分析领域中经典的线性判别分析(linear discriminant analysis, LDA)和主成分分析结合支持向量机算法(principal components analysis and support vector machine, PCA-SVM)与Bi-LSTM网络进行分类比较。 其中, LDA是一种经典的线性分类器, 可以通过矩阵特征分解的方式实现光谱数据的线性降维识别。 PCA方法是目前常用的无监督光谱降维方法, 其与支持向量机的组合使用方式在光谱数据分类领域应用广泛。 各分类器将对1.3小节中划分的光谱数据集和光谱图像数据集进行各自建模, 各分类器内部的超参数将进行分别调优, 最终将分类结果汇总对比分析。
致病菌的光谱数据提取过程和结果如图4所示。 首先, 从致病菌高光谱数据[图4(a)所示]中提取较为清晰的单波段图像, 采用灰度化、 降噪滤波、 阈值分割等图像处理手段生成对应的掩膜文件[图4(b)所示]。 然后, 将原始高光谱数据中的每个波段的细胞原图像和掩膜进行异或操作, 利用阈值分割的方式提取感兴趣区域(regions of interest, ROI)。 接着, 将单波段的细胞图像按照波段序列重新叠加组合, 重新形成新的细胞ROI的超立方体数据[图4(c)所示]。 最后, 计算ROI区域内的所有光谱形成均值光谱, 并进行MSC算法校正。 图4(d)显示了不同致病菌在450~800 nm波段下的光谱曲线图, 各类致病菌的光谱峰形相似, 且都在522、 546、 574、 590和670 nm的波段取得峰值, 但是在光谱强度上表现出差异。 其中, 空肠弯曲杆菌的光谱强度最低, 大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌存在部分光谱重叠区域。 这些强度规律的形成与光子在细胞中透射、 散射和折射的相互作用有关。 首先, 入射光先通过细胞的胶囊、 细胞壁和细胞内膜进入内部, 与细胞液、 染色体DNA和核糖体相互作用, 产生光能的二次衰减。 由于各种致病菌细胞个体的差异, 导致细胞内部这些物质的含量不同, 因此光能衰减存在差异, 从而形成独特的“ 光谱指纹” [11]。
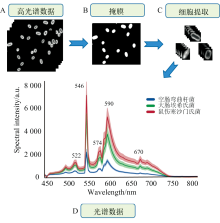 | 图4 (a) 致病菌高光谱数据; (b) 细胞图像对应的掩膜图像; (c) 提取获得的单个细胞高光谱图像; (d) MSC算法校正后的三种细菌平均光谱图Fig.4 (a) Hypercube of foodborne bacteria; (b) The mask of each image; (c) Single-cell hypercube; (d) MSC corrected spectra |
基于1.4所描述的模型判别方案, 建立LDA, PCA-SVM和Bi-LSTM模型。 其中, LDA使用自动调超参的方式进行优化; 在PCA-SVM分类器中, PCA选用前8个主成分, 光谱数据集的总解释变量为97.9%, 图谱数据集为94.9%。 SVM采用高斯核函数, 在两种数据集中参数C均为20, gamma参数均为0.15。 Bi-LSTM网络中内部输出维度参数units为200, dropout为0.2, 采用双向封装器对光谱数据进行前后双向运算。
在光谱数据集中, LDA、 PCA-SVM和Bi-LSTM分类器在测试集分别取得了80.1%、 88.5%和91.0%的准确率, 精确率分别为0.81、 0.89和0.91, F1值分别为0.80、 0.88和0.91, K值分别为0.7、 0.82和0.86, 详细结果如表2所示。 其中, 非线性模型PCA-SVM和Bi-LSTM性比线性LDA模型准确率更高。 各模型的精确率和F1值区别不大, 表明各模型的超参数均被调优, 评估性能稳定。 但是, 在一致性检测环节, LDA仅取得0.70的K值得分, PCA-SVM和Bi-LSTM则分别为0.82和0.86, 表明非线性模型在光谱分类任务中更加可靠。 综上, 光谱数据集的分类结果表明, 经典的PCA-SVM和神经网络算法Bi-LSTM相较于传统方法LDA, 准确率和稳定性更强。 在光谱和形态数据集中, 各分类器的分类结果均取得提升, 测试集分别取得了95.3%、 95.3%和98.1%的准确率, 精确率分别为0.96、 0.95和0.98, F1值分别为0.95、 0.95和0.98, K值分别为0.92、 0.92和0.97。 其中, LDA分类器准确率提升较大, 从80.1%提升至95.3%, 达到了与PCA-SVM同样的分类准确率。 光谱形态数据集的分类结果表明, 形态数据的加入能够显著提升各分类模型的性能, LDA提升了15.2%, PCA-SVM模型提升了6.8%, Bi-LSTM网络提升了7.1%。 Bi-LSTM分类器在图谱数据集中表现最为优异, 测试集取得了98.1%的准确率, 0.98的精确率、 0.98的F1-Score和0.97的K值。
| 表2 不同模型的分类结果 Table 2 Classification results from different models |
如图5所示, 通过混淆矩阵可视化手段能够直观展现各分类器在三种致病菌分类任务中详细表现。 图5(a)展示了三种分类器在光谱数据集上的致病菌分类情况, 各分类器的分均能较好的区分空肠弯曲杆菌, 但是在大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌的分类中表现较差。 已有的文献表明, 空肠弯曲杆菌体态为弧形, 与大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌存在明显差异[12], 显微高光谱成像则能够捕捉这些由各致病菌细胞物理结构和化学成分不同而产生的光子能量差异。 各分类器的性能也会影响最终判别结果, 如图5(a)所示, 有60个大肠埃希氏菌O157:H7样本被误判为鼠伤寒沙门氏菌, 而有51个鼠伤寒沙门氏菌样本被误判为大肠埃希氏菌O157:H7。 虽然使用性能更佳的PCA-SVM(误判的大肠埃希氏菌O157:H7个数为30, 鼠伤寒沙门氏菌个数为16)和Bi-LSTM(误判的大肠埃希氏菌O157:H7 29个数为, 鼠伤寒沙门氏菌个数为17)分类器能够改善假阳性问题, 但依然无法显著提升各类致病菌的分类效果。 综上, 基于光谱数据集的结果表明, 仅使用光谱特征进行分类易产生误判, 这是由于大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌均拥有相似的细胞壁、 细胞膜、 细胞质和细胞核等结构。 因此, 形态特征的加入或许能够为分类效果的改善提供帮助。 空肠弯曲杆菌为空间螺旋形, 细长且呈S形弯曲; 大肠埃希氏菌O157:H7与鼠伤寒沙门氏菌虽然菌体两端均呈钝圆形, 但在杆体长短等形态特征上存在差异。 基于图像和光谱的分类结果如图5(b)所示, 在LDA分类器中, 仅有25个大肠埃希氏菌O157:H7样本被误判为鼠伤寒沙门氏菌; PCA-SVM分类器中, 这两类致病菌的误判样本数分别为15和11; Bi-LSTM网络则能更进一步的解决假阳性问题, 仅有2个鼠伤寒沙门氏菌的样本被误判。 混淆矩阵可视化的结果表明, 大肠埃希氏菌O157:H7和鼠伤寒沙门氏菌的假阳性误判问题是制约各分类器准确率的重要原因, 使用图像和光谱特征结合先进的Bi-LSTM神经网络分类器则能够显著提高致病菌的分类精度。
在显微高光谱成像技术和图谱智能算法应用于食源性致病菌的快速检测和分类具有鲜明的数字化、 智能化的优势: (1)本研究提出的显微高光成像技术能够实现各类食源性致病菌的快速特异性表征, 为使用智能算法进行致病菌细胞级别的分类提供了数据支撑; (2)Bi-LSTM网络在光谱和图谱分类任务中具有优势, 它的端对端网络结构能够直接处理高维特征, 无需类似PCA-SVM方法使用降维预处理步骤, 并能取得比LDA更好的识别准确率; (3)形态特征和光谱数据融合形成的图谱数据集能够显著增强致病菌的特异性表达, 提升各分类器的识别精度, 与灵活且强大的Bi-LSTM网络进行结合能够进一步解决致病菌分类过程中出现的假阳性问题。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|