作者简介: 张 凡, 女, 1996年生, 河北农业大学食品科技学院博士研究生 e-mail: zf18730278721@163.com
鸭梨黑斑病在感染早期阶段引起感染区域外观的变化很微小, 肉眼难以观察, 因此对其早期识别仍然是困难的。 结合高光谱成像技术和Stacking集成学习算法, 实现了鸭梨黑斑病的潜育期识别检测。 首先, 获取健康和不同腐败程度黑斑病鸭梨样品的原始高光谱图像, 基于图像选取感兴趣区域(ROI), 然后对提取的平均光谱数据进行一阶导数(FD)、 二阶导数(SD)、 标准正态变量变换(SNVT)及组合SNV-FD和SNV-SD预处理后, 采用竞争性自适应权重取样法(CARS)提取特征波长的光谱信息。 最后基于筛选出的特征信息分别建立最小二乘支持向量机(LS-SVM)、 K最邻近法(KNN)、 随机森林(RF)和线性判别分析(LDA)分类模型。 其中, 预测效果最好的组合为SNV-FD-LSSVM, SNV-KNN和SNV-FD-RF, 准确率分别达到94%, 88%和88%。 四种算法建立的模型中, 测试集准确率不低于85.00%的个数分别为5、 3、 2和0, 因此优选出LS-SVM、 KNN和RF三个分类器用于后续的集成学习。 为提高模型准确率, 以优选出的LS-SVM、 KNN和RF三种模型作为基分类器构建Stacking学习框架, 并与单一分类器建模结果进行对比分析。 结果表明, 集成学习模型的总体识别正确率达到了98.68%, 较单一分类器模型提高了4.64%, 且对潜育期样品的识别率提高了11%。 证实了高光谱成像结合集成学习方法识别潜育期黑斑病鸭梨样品可行; 集成模型显著提高了单一模型的准确性; 为鸭梨黑斑病早期检测和病害分级提供一种新的方法, 同时为深入研究集成学习算法在光谱定性中的应用奠定了一定基础。
It is still difficult to identify black pear spots in the early stage of infection because the changes in the appearance of the infected area are very small and difficult to be observed by the naked eye. This study combined hyperspectral imaging technology and Stacking integrated learning algorithm to realize gley identification and detection of pear black spot. Firstly, a hyperspectral imaging system was used to collect the hyperspectral images of healthy pear samples and different disease grades. The region of interest (ROI) was selected based on the images, and the average spectrum was extracted. Then, First derivative (FD), Second derivative (SD), Standard Normal Variable Transformation (SNVT), SNV-FD and SNV-SD pretreatments were performed on the extracted original spectral data. Then, the Competitive Adaptive Weight Sampling (CARS) method was used to extract the spectral information of the characteristic wavelength. Finally, the Least Square support vector machines (LS-SVM), K-nearest neighbor method (KNN), Random Forest (RF) and Linear discriminant Analysis (LDA)classification models are established respectively based on the screened feature information. Among them, the combination of SNV-FD-LSSVM, SNV-KNN and SNV-FD-RF was better, with test set accuracy of 94%, 88% and 88% respectively. In the models established by LS-SVM, KNN, RF and LDA algorithms, the number of test set accuracy not less than 85.00% are 5, 3, 2 and 0 respectively. Therefore, three classifiers, LS-SVM, KNN and RF, are selected for subsequent ensemble learning. In order to improve the model accuracy, the optimized LS-SVM, KNN and RF models were used as the base classifier to construct the Stacking learning framework, and the modeling results of a single classifier were compared and analyzed. The results showed that the overall recognition accuracy of the integrated learning model is 98.68%, which is 4.64% higher than that of the single classifier model, and the recognition rate of gley samples is 11% higher. The results confirmed the feasibility of hyperspectral imaging combined with an integrated learning method to identify pear samples with a black spot in the gley stage. The integrated model significantly improved the accuracy of the single model. Moreover, it provides a new method for early detection and disease classification of black pear spots, and lays a foundation for further study on applying integrated learning algorithms in qualitative spectral analysis.
鸭梨(Pyrus bretschneideri)是中国著名的梨品种, 果肉鲜美多汁。 然而, 在贮藏过程中易受到Alternaria alternata真菌的侵染而产生黑斑病。 真菌孢子侵染到鸭梨外部, 经历潜育期后不断繁殖, 病症从隐性变为显性, 最终导致鸭梨外部和内部发生病变, 严重降低了鸭梨的商品价值, 造成资源浪费。 因此, 实现鸭梨黑斑病的快速检测和早期识别具有重要意义。
目前黑斑病的常规鉴定和检测方法主要是形态学鉴定和人眼识别。 由于链格孢菌分类系统的多样性和复杂性, 形态学鉴定费时费力、 效率低且具有破坏性。 人眼识别通过个人观察判断, 具有效率低、 主观性强等弊端[1]。 此外, 对于黑斑病潜育期的样品来说, 难以实现肉眼观察和识别, 无法准确判别样品是否侵染了病原菌; 因此准确、 快速、 无损的检测技术实现潜育期样品识别是必要的。 高光谱成像技术(hyperspectral imaging, HSI)是一种新兴技术, 样品的空间特征和光谱信息可同时得到, 是一种潜在的可行方法。 目前, 该技术被用于果蔬病害的检测方面[2, 3, 4]。 Yuan等[5]基于高光谱成像技术结合偏最小二乘判别分析实现了灵武长枣不同损伤阶段的鉴别。 Pan等[6]利用高光谱成像技术对桃子冷害进行检测, 测试集中正常样品与冷害样品分类准确率达到95%以上。 刘思伽等[7]采集了寒富苹果病害高光谱图像, 预测准确率高达100%。 以上研究表明HSI应用于果蔬病害识别是可行的, 但大多数研究集中在严重腐败样品识别分级上, 对潜育期样品的早期识别仍需深入研究。
定性模型的建立是高光谱分析的核心技术之一, 化学计量学是定性模型中的重要组成部分, 常用分类器主要包括最小二乘支持向量机(least squares support vector machines, LS-SVM)、 K-近邻算法(K-nearest neighbor algorithm, KNN)、 线性判别分析(linear discriminant analysis, LDA)和随机森林(random forest, RF)等。 以上单一分类器模型具有泛化性能不佳的弊端。 最近的理论和实证研究表明, 使用集成学习算法策略(将多个弱学习者的预测组合成一个预测器)分类和检测更准确[8], 源于集成分类器可以将单一分类器的优点有效结合。 在众多集成算法中, Stacking的主要思想为: 对训练集进行不断训练得到多个初级学习器, 此时将测试集代入初级学习器中进行预测, 最后将得到的输出值作为下一步训练的输入变量, 最终的标签作为输出值, 用于训练次级学习器。 Stacking算法在黑枸杞分级[9]、 单粒玉米种子水分检测[10]等研究上有所应用。 由于Stacking集成学习具有较强的学习能力, 将其与高光谱成像技术相结合, 有望实现鸭梨黑斑病潜育期的早期识别。
综合以上分析, 为了实现鸭梨黑斑病潜育期的早期识别, 结合高光谱成像技术, 采用Stacking集成学习算法, 将LS-SVM、 KNN和RF作为基学习器组合成强分类器, 建立鸭梨黑斑病的早期识别模型。 探讨Stacking融合算法应用于高光谱定性分析的适用性, 发挥Stacking综合多个模型预测结果的优点, 为鸭梨黑斑病潜育期识别和病害分级检测提供一种新的方法。
实验用鸭梨购自河北保定农贸市场。 挑选大小、 成熟度基本一致, 表面无明显缺陷、 物理损伤、 病菌感染的新鲜果实, 纸箱包装后立即运输至河北农业大学食品科技学院实验室, 用2%(V/V)的次氯酸钠溶液洗涤5 min, 再用蒸馏水进行冲洗干净, 室温下自然晾干备用。
病原菌A. alternata的分离培养: 以5个自然感染黑斑病的梨果实为实验材料进行分离, 取其一块腐败组织置于马铃薯葡萄糖琼脂(PDA)培养基上, 再放于培养箱(90%、 25 ℃)中培养7 d。 待培养基上长出大片绒毛状菌落后, 用一次性接种环从培养基表面挑取菌丝至新的PDA平板上进行纯化, 纯化三次后进行菌悬液的制备。
孢子菌悬液的配制: 将真菌孢子从PDA培养基表面去除, 悬浮在含有0.05%(V/V)吐温-80的无菌生理盐水中。 悬浮液经四层无菌纱布过滤, 最后用血细胞仪将滤液的孢子浓度调整至1× 106个· mL-1左右。
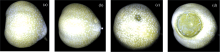
损伤接种: 用注射器将10 μ L的孢子菌悬液注射到鸭梨的赤道线附近组织中, 后转入恒温恒湿箱内贮藏, 保证环境湿度为90%, 温度为(25± 2) ℃。 为获得不同腐败程度的鸭梨样品, 每天接种8个, 整个试验持续17 d, 最终获得136个不同腐败程度的鸭梨样品和68个健康样品用于后续实验, 由于3个鸭梨样品出现了其他严重的磕碰损伤, 因此将这3个鸭梨去除。 培养过程中, 记录每个接种样品的病斑直径, 根据病斑面积将全部鸭梨划分为4个等级[11], 如图1所示: 健康鸭梨为1级、 染病无病斑潜育期样品为2级、 病斑面积≤ 20%为3级、 > 20%的为4级, 则共有68个1级样品, 75个2级样品, 34个3级样品和24个4级样品。
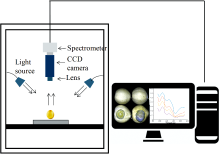
采用的高光谱成像系统如图2所示, 鸭梨样品的高光谱图像由其获得。 仪器波长范围为935~1 720 nm, 主要由CCD相机(SPECIM FX 17, 芬兰SPECIM公司)、 光源(15 V, 150 W)、 载物台、 计算机等部分组成。 共224个波段, 全波段采集成像速度为50 FPS。 实验开始前, 将仪器预热30 min保证其稳定性。 为了获得准确的高光谱数据, 在采集图像前, 需要确定成像距离、 CCD相机曝光时间、 样品平台的移动速度等系统参数。 经过反复试验, 上述三个参数分别设置为30 cm、 50 ms和7.5 mm· s-1。 为了避免环境杂散光的干扰, 图像采集过程在暗箱内完成。 采集样品前, 将1个样品按照病斑朝上的方式放置在位移平台上进行线扫描。
同时, 对原始高光谱图像(R0)进行标定以减少CCD相机暗电流影响, 采用反射效率为99%的聚四氟乙烯白板获得白板图像(Rw)。 暗场图像(Rd)是用一个不透明的盖子盖住镜头, 根据式(1)计算校正后的相对图像RI。
感兴趣区域(region of interest, ROI)的选择在HSI检测中是必要的, 直接影响了模型的性能。 ROI由具有相似特征的像素组成, 用于提取光谱特征。 对于未接种的新鲜鸭梨, 选取无磕碰、 无任何损伤部位为ROI; 等级2样品, 选取损伤接种点及接种点周围区域; 等级3、 4样品, 选取损伤接种点附近的病斑区域。 将ROI描绘成20 000像素点的方形, 通过Matlab 2014b计算其中所有像素的光谱作为最终提取信息。
仪器本身、 采集环境及样品变化等因素造成原始光谱数据存在一定的背景噪声和基线漂移。 因此, 采用了一阶导数(first derivative, FD)、 二阶导数(second derivative, SD)、 标准正态变量变换(standard normal variable transformation, SNV)及顺序组合: SNV-FD和SNV-SD共5种预处理方法对原始光谱进行预处理, 以减少各种因素带来的噪声。 光谱预处理在PLS_Toolbox 7.5中进行。
原始光谱数据中存在许多无关的共线变量信息。 这些冗余变量不仅影响模型的预测性能, 而且大大增加了计算时间和工作量。 通过特征波长筛选的方法, 可以实现有效波长信息的提取。 采用了竞争性自适应权重取样法(competitive adaptive weight sampling method, CARS)进行变量提取, 降低无关信息对预测性能的负面影响, 提高模型的分类准确率和建模效率。
1.6.1 基于单一分类器的模型构建
利用Kennard-Stone(KS)算法将原始数据集按3:1分为训练集(151个)和测试集(50个), 以LS-SVM、 RF、 KNN和LDA四种常用算法作为分类器构建黑斑病鸭梨的诊断模型。 选择分类器时应充分考虑学习器的多样性和相互独立性, 以保证构建集成学习模型时各模型间信息的有效互补。 LSSVM较传统支持向量机算法有所改进, 支持向量机具有的二次规划方法被最小二乘线性系统代替, 可以简化问题, 提高计算速度。 RF算法学习效率高泛化效果强, 适用于特征较多的高维数据集, 随机树的数目设置为500。 KNN算法的核心是用距离最近的k个样本数据的类别代表目标点的类别, KNN中相邻数目设置为9。 LDA通过确定特征的线性组合, 将一个组合划分为两个或多个组合, 这种多变量方法有效地使两组间的比例最大化, 使组内的比例最小化。
1.6.2 基于Stacking集成学习的模型构建
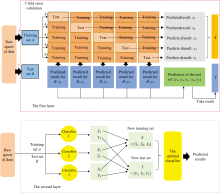
Stacking集成学习算法思想展示在图3中, 该思想包括两层网络结构, 第一层利用不同的单一分类器(基分类器)建立多个单一模型(基模型), 然后将多个基模型的预测结果作为第二层的输入信息, 最后将预测效果最佳的基分类器作为第二层的分类器进行训练。 综合分析上述四种单一分类器对鸭梨的早期识别效果, 优选出三种预测结果较好的分类器作为集成学习中第一层的基分类器, 以结果最佳的分类器作为第二层的分类器。
第一层: 基于五折交叉验证思想的基模型建立
(1)数据集的划分
基于交叉验证思想, 将划分好的训练集A(151个)平均划分为5份, 记为A1、 A2、 A3、 A4、 A5。
(2)基模型的建立
利用A2— A5的样品作为训练集建立模型并预测A1和B中的样品, 得到A1的预测类别x1和B的第一次预测类别y1。 同理, 用A1、 A3、 A4、 A5的样品建立模型并预测A2和B中的样品, 得到A2的预测类别x2和B的第二次预测类别y2。 以此类推, 最后得到了训练集A的预测类别x1— x5及预测集B的预测类别y1— y5。 最后, 将训练集的预测类别x1— x5合并得到训练集A的预测类别X1, 将预测类别的y1— y5取众数得到测试集B的预测类别Y1。 对其余两个基分类器进行同样的操作得到X2, X3和Y2, Y3。
第二层: 基于第一层基模型预测结果的鸭梨黑斑病诊断模型建立
将第一层各个基模型的预测标签合并, 得到新的矩阵X={X1, X2, X3}, Y={Y1, Y2, Y3}。 分别将X、 Y作为训练集及测试集, 利用建模效果最佳的最优单一分类器进行第二层网络中模型的建立和预测, 从而实现对单一分类器性能的综合利用, 增强模型的学习和泛化能力。
1.6.3 模型评价
模型性能通过准确率Accuracy(%)进行评估, 计算公式如式(2)
式(2)中, TP表示实际为特定类别(T)且被正确分类的样品数量; TN为不属于T且被分类器正确分类的样品数量; FP为不属于T但被分类器错误分类的样品数量; FN为属于T但被分类器错误分类的样品数量。
上述数据分析由ENVI 5.2, Matlab 2014b和Python 3.8软件完成。
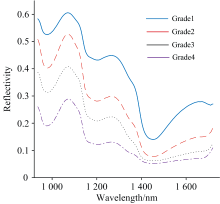
分别从68个健康(等级1)样品、 75个潜育期(等级2)样品、 34个等级3样品和24个等级4染病样品的ROI部位提取光谱, 然后分别对每个病害等级样品的光谱进行平均, 得到四条平均光谱如图4所示, 图中实线、 虚线、 点线和点划线分别代表等级1、 等级2、 等级3、 等级4样品。 由图4可以看出, 在整个光谱范围内, 各级样品的光谱曲线变化趋势类似, 但反射率值存在明显差异, 说明不同腐败程度鸭梨样品的化学成分组成大致相同, 但含量有所不同。 此外, 随着病害程度的加重, 其总体光谱强度逐渐降低, 这说明在黑斑病发病过程中, 梨中各化学成分含量均有一定降低, 主要原因是: 当病原菌Alternaria alternata侵染样品组织时, 细胞壁的有序结构被破坏[12], 造成一些营养成分的损失。 在960~1 380 nm范围内, 健康梨和黑斑病梨的光谱曲线有较大差异, 与病害发生中的果实内部水分含量和碳水化合物的变化有关。 病原菌侵染梨果实时, 不断消耗水果的蔗糖、 果糖和葡萄糖以维持自身生存, 同时果实细胞壁发生破裂, 多糖成分减少。 其中973 nm附近的吸收峰主要源于水和糖类成分的O— H键振动[13], 1 150、 1 210和1 270 nm属于C— H二级倍频伸缩, 1 446 nm处为O— H伸缩振动的一级倍频。
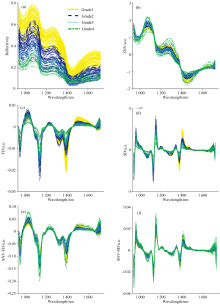
原始及经过SNV、 FD、 SD、 SNV-FD和SNV-SD五种方法预处理后的光谱如图5所示, 图中实线、 虚线、 点线和点划线分别代表等级1、 等级2、 等级3、 等级4样品的平均光谱。 对比图5(a— f)可以看出, 经过预处理后, 光谱数据保留了原始光谱的吸收特性, 并有一定的改善。 经过SNV预处理后的光谱[图5(b)]与原始光谱较为接近, 光谱的基线平移现象得到修正, 补偿由鸭梨样品的大小不均匀和表面散射引起的偏差。 经过FD处理后的光谱明显消除了基线和背景的干扰, 有效地削弱了其他无关因素对样品的影响。 此外, 原始光谱中出现的特征峰(973、 1 071、 1 203、 1 270和1 446 nm)更加明显, 说明一阶导数处理强化了微小特征峰, 从而提高了分辨率和灵敏度, 且出现了新的特征峰: 1 140、 1 400和1 500 nm。 经过SD预处理后[图5(d)], 消除了散射和一些重叠峰引起的基线漂移。 由于不同腐败程度鸭梨样品的复杂性, 单独的光谱预处理可能不能提供理想的预测结果, 因此也采用了不同的预处理方法组合对原始光谱进行处理。 图5(e)和(f)显示了SNV-FD和SNV-SD预处理后的光谱, 相比原始光谱, 由背景噪音带来的干扰得到了有效消除, 基线漂移也明显减弱, 在973、 1 071、 1 140、 1 203、 1 270、 1 400、 1 446和1 500 nm出现明显波峰。 由于光谱信号是十分复杂的数据, 直接观测光谱信号特征并不能筛选出最佳的预处理方法, 因此需要进一步结合建模结果对预处理效果进行评价。
采用CARS算法对进行特征波长筛选, 原始光谱以及SNV、 FD、 SD、 SNV-FD和SNV-SD五种预处理方法后筛选特征波长数分别为30、 17、 25、 19、 17和31, 较原始波段数量224个降低了86.16%~92.41%, 有效减少了计算量, 提高了分类模型的计算速度。
将原始光谱和预处理后的光谱经过CARS筛选后分别建立LS-SVM、 KNN、 RF和LDA四种分类模型。 其训练结果和预测结果如表1所示。 对比五种预处理方法对建模结果的影响发现, 基于预处理光谱建立的模型都在一定程度上提高了模型的预测准确率, 充分说明了经过预处理后的光谱数据减少了由基线漂移、 背景噪声等其他无关因素造成的负面影响, 提高了模型的识别率和灵敏程度。 对于LS-SVM分类器, 最优模型为SNV-FD-CARS-LSSVM, 测试集的准确率由未预处理的82.00%提高到94.00%。 对于KNN分类器, 最优模型为SNV-CARS-LSSVM, 测试集准确率为88.00%。 对于RF分类器, 最优模型为SNV-FD-CARS-LSSVM, 测试集准确率为88.00%。 对于LDA分类器, 最优模型为SD-CARS-LDA, 测试集准确率由74.00%提高至82.00%。
| 表1 单一分类器模型建模结果 Table 1 The modeling results of single classifier model |
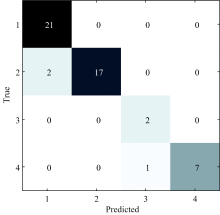
由表1可知, 基于SNV-FD预处理后的光谱数据的LSSVM模型结果最佳。 为进一步明确该模型对不同类别鸭梨的识别效果, 采用其混淆矩阵对结果进行分析, 如图6所示。 混淆矩阵的行和列分别为样品的真实类别和预测类别, 其对角线上的数字为被正确识别的样品数量, 对角线以外的数字为被错误识别的样品数量。 右侧颜色条代表样品数量, 颜色越浅, 表示数量越大。 如图6所示, 对健康样品、 等级3样品的识别率均为100%, 对4级样品和潜育期样品(等级2)识别率分别为89%和87.50%, 其中有1个4级样品被误判为3级样品, 此现象可能源于在腐败程度较重的样品中, 个别样品的病斑之间的差异较小, 从而导致误判。 有两个2级样品被误判为健康鸭梨, 这可能是由于鸭梨被真菌侵染后, 在储存时间较短的情况下, 外观变化不明显, 光谱变化较小, 从而导致被误判为健康样品的次数较多。 因此进一步采用Stacking集成学习方法, 提高模型的预测能力, 实现对潜育期样品的早期识别。
在LS-SVM、 KNN、 RF和LDA四种算法建立的模型中, 测试集准确率不低于85.00%的个数分别为5、 3、 2和0, 因此优选出LS-SVM、 KNN和RF三个分类器用于后续的集成学习。
为了进一步提升分类效果, 构建Stacking集成学习模型对鸭梨黑斑病级别进行识别分类。 将原始光谱经过SNV、 FD、SD、 SNV-FD、 SNV-SD预处理后, 采用CARS对其筛选特征波长, 以Stacking集成学习方法调用LS-SVM、 KNN、 RF三个分类器对光谱数据训练, 每个分类器得到的训练结果组成一个新的训练样本作为元分类器的输入, 第二层模型中的LS-SVM分类器的输出结果为最终的输出结果, 结果如表2所示。 综合比较, 经过集成分类器建立的模型准确率较单一分类器显著提高, 其中经过SNV、 FD、 SD、 SNV-FD预处理的光谱所建立的模型准确率均达到90%以上, 且最高准确率达到98%, 较单一分类器提高了4.64%。
| 表2 集成分类器模型建模结果 Table 2 The modeling results of integrated classifier model |
为了进一步观察各模型对不同等级鸭梨样品的分类效果, 进行了各个模型的混淆矩阵计算, 如图7所示。 由图可知, Stacking集成学习模型表现出良好的分类性能, 其中经过FD预处理后的光谱模型结果最佳, 对健康、 潜育期(等级2)和等级3样品的识别率均达到100%, 4级样品中有1个被误判为3级样品。 此模型的总体识别率为98%, 较单一分类器提高了4.64%。 另外, 对潜育期样品的识别率相较于单一分类器模型结果来说提高了11%, 表明基于Stacking算法的集成分类器能够较好地实现对鸭梨黑斑病的早期检测。
传统的机器学习方法在构建高精度估计模型和提高泛化能力方面存在一定的局限性, 但将多个不同的单一模型组合成一个模型的集成学习明显优于单一机器学习模型[13]。 本研究Stacking模型采用五折交叉验证进行训练, 缓解了过拟合现象, 保证模型的拟合能力较强的同时, 提升了估算精度, 增强了模型的适用性。 表明Stacking集成学习算法用于高光谱定性判别分析具有较强的泛化能力和较高的预测精度, 在光谱分析领域具有进一步研究、 探索的价值和意义。
采用高光谱成像技术结合Stacking集成学习实现了鸭梨黑斑病潜育期识别和病害等级的快速无损检测。 对比分析了健康(等级1)、 潜育期(等级2)、 病害等级3和等级4的黑斑病鸭梨的光谱曲线, 并以LS-SVM、 KNN、 和RF为基分类器构建了黑斑病病害程度的Stacking集成学习预测模型。 结果可得, 集成分类模型的总体正确率可达98.68%, 较单一分类器提高了4.64%, 对潜育期样品的识别率相较于单一分类器模型提高了11%。 表明集成学习更有利于结合各个分类器的优势, 加强单一分类器的分类效果, 有效提高定性模型的分类精度, 且对于算法的鲁棒性和泛化能力的增强也具有重要意义, 显著提高了鸭梨黑斑病潜育期识别效果。 本研究为鸭梨黑斑病早期防治、 精准施药及检测仪器开发提供科学依据, 后续可利用更多集成学习方法对光谱信息加以利用, 进一步提高建模预测结果。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|