作者简介: 周傲, 1997年生, 合肥工业大学资源与环境工程学院硕士研究生 e-mail: 2019170674@mail.hfut.edu.cn
微生物的生长代谢往往受盐度的影响, 因此筛选出耐盐性强的菌株对含盐废水的生物处理意义重大。 选取一株从海洋分离具备耐盐异化金属还原的功能细菌(DMRB)——耐盐希瓦氏菌( Shewanella aquimarina XMS-1)作为研究对象, 探究盐度对XMS-1还原Fe3+过程及胞外聚合物变化的影响。 考察了不同盐度下XMS-1的Fe3+还原能力和胞外聚合物(EPS)的含量, 并采用三维荧光光谱(3D-EEM)、 拉曼光谱(Raman spectra)、 红外光谱(FTIR)及其对应的二维相关光谱(2D-COS)分析了XMS-1还原Fe3+过程中EPS的变化。 结果表明, 蛋白为XMS-1 EPS中主要物质, 占EPS含量的80%以上, 多糖的含量相对较少, 3%盐度条件下会促进XMS-1的EPS产量, 表明XMS-1在高盐环境中会分泌更多的EPS来保护细胞进行正常的生理活动。 Fe3+还原过程在盐度为1%~4%时得到促进, 而在盐度高于5%时则受到抑制, 过高盐度会抑制XMS-1的生长, 导致Fe3+还原率下降, 其中3%盐度下Fe3+还原率最高可达44.1%, 相对于对照组增加了2.18倍。 FTIR和Raman光谱结果显示XMS-1 EPS中含有羧基、 羟基、 氨基和羰基等金属离子氧化还原功能基团, 其中3%盐度下EPS中蛋白酰胺类和多糖类代表峰增强, 蛋白酰胺类代表峰变化显著, 含O-和N-基团参与了Fe3+还原过程。 此外3D-EEM结果显示, Fe3+还原过程结束后, EPS中色氨酸和酪氨酸两种荧光组分强度均下降, 结合2D-COS光谱结果分析, 发现色氨酸类蛋白在Fe3+还原过程中先发生了显著变化, 表明这两种荧光组分参与了Fe3+的还原过程, 其中色氨酸类蛋白在还原过程中作用更强。 本研究不仅丰富了对耐盐菌EPS胞外电子转移过程的认识, 也突出了EPS在自然环境中铁氧化还原转化的意义。
The growth and metabolism of microorganisms are often affected by salinity, so screening the salt-tolerant strains is of great significance for the biological treatment of saline wastewater. In this paper, Shewanella aquimarina XMS-1, a marine strain with salt tolerant metal reduction function (DMRB), was selected as the research object to explore the effects of salinity on the reduction process of Fe3+ and the changes of extracellular polymers. The Fe3+ reduction ability of XMS-1 and the extracellular polymer (EPS) content under different salinity were investigated. Furthermore, Three dimensional fluorescence excitation-emission (3D-EEM), Raman spectra (Raman), Fourier transform infrared spectroscopy (FTIR), and Two-dimensional correlation spectroscopy (2D-COS) were used to analyze the changes in extracellular polymers during the reduction of Fe3+ by XMS-1. The results show that protein is the main substance in XMS-1 EPS, accounting for more than 80% of EPS content, and polysaccharide content is relatively small. 3% salinity conditions promote EPS production. XMS-1 will secrete more EPS in the high salt environment to protect cells from normal physiological activities. The reduction process of Fe3+ is accelerated at the salinity of 1%~4%, but is inhibited when the salinity is higher than 5%. Too high salinity would inhibit the growth of XMS-1, resulting in the decrease of the Fe3+ reduction rate. The reduction rate of Fe3+ was increased by 2.18 times and reached 44.1% at the salinity of 3%. FTIR and Raman spectra showed that XMS-1 EPS contained metal ion redox functional groups such as carboxyl, hydroxyl, amino and carbonyl. The peaks of protein amides and polysaccharides in EPS were enhanced at 3% salinity, and the representative peaks of protein amides changed significantly. The O- and N- groups were effective redox groups in Fe3+ reduction. In addition, the Three-dimensional fluorescence results showed that after the Fe3+ reduction process, the intensities of the two fluorescent components of tryptophan and tyrosine in EPS decreased. Combined with the analysis of 2D-COS spectral results, it was found that tryptophan-like proteins changed significantly during the reduction of Fe3+, indicating that the two fluorescent components were involved in the reduction process of Fe3+, and tryptophan-like proteins played a stronger role in the reduction process. This study enriched the understanding of the extracellular electron transfer process of EPS in halophilic bacteria, and highlighted the significance of EPS in iron redox transformation in natural environment.
含盐废水由于处理难度较大且成本较高, 其处理技术的创新与优化一直是国内外研究的热点问题[1]。 目前, 处理高盐度废水的方法包括膜分离、 离子交换、 电解等物化处理和生物处理。 生物处理技术具有经济、 无害、 操作简单等优点, 在高盐废水治理中具有更高的应用潜力[2]。 然而, 高浓度含盐废水会抑制微生物的生长, 甚至使其裂解死亡, 抑制了生物处理效率[3]。 因此, 筛选驯化对盐度耐受性强的菌株, 对于强化高盐废水生物处理的效率具有重要的实际意义。
胞外聚合物(extracellular polymeric substance, EPS)作为微生物的重要组分, 可以为细胞提供保护层, 从而使微生物在高盐环境中能够维持较高的活性[4]。 越来越多的研究者采用各种光谱技术方法对EPS组成和性质开展了深入研究。 Fatima[5]等采用傅里叶变换红外光谱(FTIR)对Pseudomonas entomophila PE3中提取的胞外多糖结构分析, 发现胞外多糖中存在与钠离子结合的重要官能团, 具有Na+积累能力。 王子超[6]等结合三维3D-EMM和FTIR技术研究了不同盐度下上流式厌氧污泥床的污泥胞外聚合物(EPS), 发现盐度增加促使EPS蛋白和多糖含量增加, 多糖含量的变化更为显著。 Chen等对厌氧膜生物反应器处理不同浓度NaCl合成废水的性能进行了评估, 3D-EEM分析表明随着盐度的增加, EPS中出现了更多的蛋白质类物质以及氨基酸[7]。 高盐体系中, 好氧颗粒污泥EPS的3D-EEM的荧光强度显著增强, 表明好氧颗粒污泥通过提高EPS的生成来保持细胞结构的稳定[8]。
异化金属还原菌(dissimilatory metal reducing bacteria, DMBR)可以将细胞内有机底物代谢获得的电子转移到细胞外电子受体, 在金属元素地球化学循环和环境污染物转化中起着关键作用[9]。 希瓦氏菌(Shewanella)是DMRB中常见的一种菌属, 也是研究微生物胞外电子传递的模式菌株。 从不同的环境中筛选出来的Shewanella具有独特的性质和适应能力, 其EPS对铁还原性能的重要性一直被不断报道。 Shewanella MR-1的EPS可以促进细胞粘附到铁氢化物表面, 驱动微生物Fe3+还原[10], 且还能够改变细胞-固相产物界面附近的环境, 间接影响Fe3+氧化物的结晶度以此调节细胞外电子转移[11]。 然而, 现有Shewanella菌株大多分离自淡水或陆地环境, 对高盐环境中分离的Shewanella菌株EPS在其Fe3+还原中的性能研究很少。
本研究基于一株从海洋沉积物中筛选获得的Shewanella aquimarina XMS-1[2], 结合FTIR, 3D-EEM, Raman和2D-COS等光谱分析方法, 通过对不同盐度条件下该菌株对Fe3+还原能力、 EPS含量及组成等, 探究了高盐条件下XMS-1 EPS对Fe3+还原过程的影响及其主要作用机制, 表明了耐盐菌XMS-1 EPS重要的电子传递功能。 在基于希瓦氏菌的高盐工业废水生物处理中, 可调控其EPS的产生, 加速电子传递能力, 还原高价有害重金属, 降低重金属毒性。
实验所用希瓦氏菌菌株XMS-1, 在GenBank的登录号为MT032499。
培养基为矿物盐培养基, 其组成为: NH4Cl 0.46 g· L-1, K2HPO4 0.225 g· L-1, KH2PO4 0.225 g· L-1, MgSO4· 7H2O 0.117 g· L-1, (NH4)2SO4 0.225 g· L-1, 20 mmoL· L-1乳酸钠为电子供体, 以及10 mL Wolfe微量元素100× 浓缩液。
采用热提取法从希瓦氏菌XMS-1菌液中提取EPS。 在6 000 r· min-1的转速下将细菌悬液离心5 min去除上清液得到菌体, 用0.85%NaCl溶液将菌体离心洗涤三次, 之后重悬于0.85%的NaCl溶液中, 置于38 ℃水浴锅30 min, 再以6 000 r· min-1的速度离心10 min, 上清液通过0.22 μ m滤膜得到EPS。 EPS中多糖和蛋白分别用硫酸蒽酮法[12]和Lowry法[13]测定。
采用荧光分光光度计(F-7100, Hitachi, Japan)测定EPS溶液的激发发射矩阵荧光光谱(EEM), 荧光参数的设置: 激发光波长范围为200~400 nm, 发射光波长为250~550 nm, 两者的狭缝宽度和步长均为5 nm, 扫描速度为1 200 nm· min-1。 通过拉曼显微镜系统(Renishaw 2000, UK)记录拉曼光谱, 用He Ne激光器(633 nm, 25 mW)激发, 激光功率约为7 MW, 利用积分时间为1 s和放大率为50倍(N.A.=0.75)的透镜。 傅里叶红外光谱仪(Vertex 70, Bruker)的扫描波长范围400~4 000 cm-1, 分辨率4 cm-1。
通过将常规光谱拓升一个维度, 提高光谱分辨率, 得到二维相关光谱(2D-COS)。 2D-COS能够观察到在常规光谱中不易观察到的细微变化, 识别结构变化的相对方向和特定顺序。 分析结果以同步和异步图的形式呈现, 同步图显示光谱变量, 波数“ 同相” 变化, 即信号同时变化; 异步图提供有关“ 异相” 变化, 揭示了光谱中信号的变化顺序。 2D-COS由强度变化比例解释, 其中红色、 蓝色和白色分别对应于正、 负和无(即零)相关性[14]。
二维荧光分析同样采用荧光分光光度计(F-7100, Hitachi, Japan), 荧光参数的设置: Ex和Em波长狭缝宽度为5和10 nm, 且二者波长差λ =70 nm, Ex波长扫描范围200~550 cm-1, 扫描速度为240 nm· min-1。
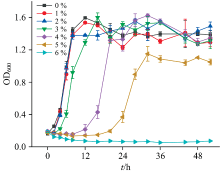
XMS-1在不同NaCl浓度条件下的生长曲线如图1所示。 与对照组相比, 1%盐度对于菌体生长速率几乎没有影响, 而2%~5%盐度体系菌体生长速率分别降低了6.43%, 7.96%, 19.65%和60.39%。 当盐度为1%~4%时, 菌体最终生长浓度基本一致。 盐度提高到5%时, XMS-1迟滞期长达近15 h, 且稳定期OD600值显著降低。 而当盐度继续增加到6%时, XMS-1生长完全受到抑制。 高盐度下XMS-1的生长会受到明显的抑制, 盐度会抑制ATP的消耗, 直接降低微生物可利用的能量[4]。 耐盐菌Ochrobactrum sp.(HXN-1)和Aquamicrobium sp.(HXN-2)能在2%盐度下生长[15], XMS-1具有更强的耐盐特性, 可以更好地应用于高盐废水体系。
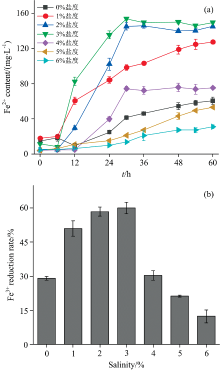
如图2(a, b)所示, 盐度对希瓦氏菌XMS-1还原Fe3+过程影响显著。 1%~4%盐度对Fe3+还原有促进作用, 其中3%盐度时实现了60% Fe3+的还原, 分析认为NaCl可以促进细菌从培养基中获得更高的营养物质[16]。 当盐度继续升高至5%和6%时, 菌株的Fe3+还原能力受到抑制, 与对照组相比Fe3+的还原率分别降低了7.73%和16.75%。 综合考虑, 选取Fe3+还原率最高的3%盐度条件, 探究耐盐希瓦氏菌XMS-1 Fe3+还原过程中EPS特性及变化。
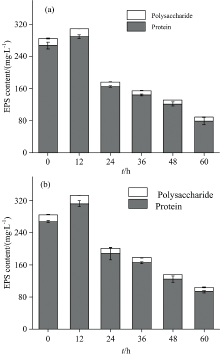
图3(a, b)分别显示了不同盐度下Fe3+还原过程中EPS组成和含量的变化。 结果表明, 蛋白质为EPS中的主要物质, 占EPS含量的80%以上, 多糖的含量相对较少。 在12 h时EPS含量显著增加, 之后开始逐渐降低。 分析认为XMS-1初期在LB培养基中富集培养, 而Fe3+还原过程实验为矿物盐培养基, 培养基的转化可能导致XMS-1释放出更多的EPS。 与对照组相比, 3%盐度促进了Fe3+还原过程和EPS产量, 有研究显示EPS产量增加有利于微生物应对NaCl的胁迫[17]。 XMS-1在高盐环境中会分泌更多的EPS来保护细胞进行正常的生理活动, 具有更强的耐盐性。
2.4.1 荧光光谱
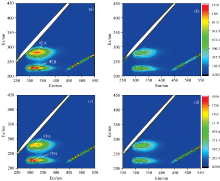
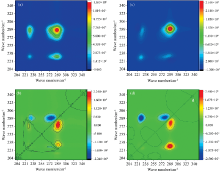
XMS-1在培养36 h内基本完成Fe3+还原, 为了更好地明确EPS在Fe3+还原过程中的作用, 对不同生长时期XMS-1的EPS进行光谱分析, 鉴定其中氧化还原活性基团的种类和组成。 分别如图4(a— d)所示, EPS三维荧光图谱中出现两个芳香蛋白类荧光峰, 峰A(Ex/Em: 275~280/328~334 nm)属于色氨酸, 峰B(Ex/Em: 220~230/322~336 nm)属于酪氨酸[18], Fe3+还原后, 实验组和空白组中荧光峰A和B的强度均降低, 与EPS中蛋白含量降低的趋势相一致, 表明色氨酸和酪氨酸类蛋白物质参与了Fe3+还原过程。 3%盐度下, 色氨酸和酪氨酸两种蛋白类物质荧光峰比对照组更强, 表明盐度会促使芳香类蛋白物质的生成, 加速Fe3+的还原。 Zhang等研究中, 发现EPS中色氨酸和酪氨酸具有强的供电子能力以及与配体络合性质, 能参与金属还原过程且与金属络合, 与本研究中色氨酸和络氨酸促进Fe3+的还原结果一致[19]。
为了更好地理解EPS在Fe3+还原过程荧光组分的变化, 进一步用二维相关同步光谱技术对三维荧光光谱分析结果进行解析。 在二维荧光图谱中波长200~300 nm代表蛋白质类物质、 300~390 nm代表富里酸类物质、 390~550 nm代表腐殖质类物质[图5(a, b)]。 峰A和峰B表示蛋白质类物质中的色氨酸和酪氨酸, 峰C为富里酸类物质, 峰D为腐殖质类物质。 2D-COS给出了具有更高分辨率的EPS荧光组分, 图中显示, EPS中荧光组分主要为蛋白质类物质, 微量的富里酸和腐殖质类物质在2D-COS中不显示。
图6(a— d)分别为二维荧光光谱的同步图和异步图, 对角线观察到的峰值表示相同振动频率的自相关峰230和280 cm-1; 位于非对角线位置的为交叉峰, 代表自相关峰对应的两个独立波数(230, 280 cm-1)相关性。 XMS-1 Fe3+还原过程中, 同步图中两个自相关峰230和280 cm-1都是正值, 280 cm-1峰强于230 cm-1, 表明色氨酸物质更多, 与3D-EEM结果中色氨酸荧光峰更强结果一致。 交叉峰两个独立波数(230, 280 cm-1), 在同步图中为正值, 异步图中为负值, 符号相反, 因此280 cm-1会优先发生变化, 即色氨酸类蛋白质峰会优先发生变化。 二维荧光谱图结果, 表明色氨酸类蛋白质峰在Fe3+还原过程优先发生变化且变化最为显著。 氨基酸自由基是电子跳跃的” 驿站” , 金属还原时电子转移途径依赖于氨基酸自由基的形成[20], 并且色氨酸残基还可以作为氧化还原酶、 水解酶的电子转移的辅助因子[21]。
2.4.2 红外光谱分析
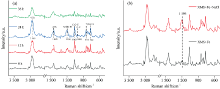
红外光谱鉴定了EPS中有助于Fe3+还原的有机基团随实验过程的变化。 如图7(a, b)所示, 在Fe3+还原过程3%盐度下EPS的FTIR峰值和数目基本一致, 但峰强发生变化, 化学基团的相对含量存在差异。 3 400 cm-1处的峰可归属为碳水化合物中结合水的O— H键的伸缩振动[22]; 1 650, 1 596和1 176 cm-1附近的峰表征蛋白特有二级结构, 分别为酰胺Ⅰ 带中B-折叠片状蛋白质结构的C=C和C=O伸缩振动、 蛋白结构中N— H弯曲和酰胺Ⅲ 的C— N的伸缩振动[23]; 1 450 cm-1处的峰属于C— H键, 为蛋白质分子中甲基的对称和反对称弯曲振动峰; 1 039和1 088 cm-1附近的峰归属于多糖或类多糖物质中的对称和不对称C=O伸缩振动[24]; 1 000 cm-1以下的峰属于指纹区域, 是和氨基酸相关的伸缩振动峰[25]。 结果表明, XMS-1 Fe3+还原过程中EPS含有蛋白和糖类等能促进金属离子氧化还原的功能基团, 如羧基、 羟基、 氨基、 羰基等。
由图7(a)可知, 24 h时各红外振动峰表现最强, Fe3+还原结束后, 蛋白多糖相关功能基团都减弱, 证实蛋白多糖参与了Fe3+还原过程。 同时, 比较了对照组与3%盐度体系24 h的红外光谱变化[图7(b)], 1 213 cm-1附近蛋白C— N吸收峰和1 060 cm-1附近多糖C=O吸收峰峰强增加, 表明在3%盐度下EPS中的蛋白和多糖含量增加。 蛋白C— N吸收峰和多糖C=O吸收峰发生了蓝移, 振动所需能量增强, 基团更加稳定, 说明3%盐度体系中EPS中蛋白和多糖在Fe3+还原过程中有更强的作用。
进一步对3%盐度体系Fe3+还原过程中红外光谱进行了二维相关光谱分析[图8(a, b)], 两个独立波数为(1 089, 1 176 cm-1)。 同步图[图8(a)], 对角线的自相关峰1 089和1 176 cm-1均为正, 代表Fe3+还原过程中特征峰是相同的变化趋势。 交叉峰在同步和异步图中同为正, 1 089 cm-1的峰比1 176 cm-1峰优先发生变化, 表明多糖物质的C=O伸缩振动峰在Fe3+还原过程会先受到影响, 但是蛋白酰胺Ⅲ 带中C— N的伸缩振动变化明显, 与荧光类蛋白物质变化显著结果相符。
 | 图8 3%盐度体系下FTIR分析生成的2D-COS图 (a): 同步图; (b): 异步图Fig.8 2D correlation contour maps by FTIR analysis in 3% salinity system (a): Synchronous; (b): Asynchronous |
2.4.3 拉曼光谱
如图9(a, b)所示, 拉曼光谱能够提供更多关于EPS基团的信息。 2 969 cm-1为— CH2非对称伸缩振动峰; 1 445 cm-1为— CH2剪切振动峰起源于蛋白质和碳水化合物的结合[26]; 1 196 cm-1为蛋白质结构中酰胺Ⅲ 所代表的峰[27]; 1 063 cm-1为C— O— C对称的糖苷连接或C— N和C— C的伸缩振动; 1 003 cm-1为C— C芳香环的伸缩振动; 762~844 cm-1为与酪氨酸相关的峰[28]。
和红外光谱一样, Fe3+还原结束后, 蛋白多糖相关功能基团都减弱, 同时24 h 3%盐度体系中1 196 cm-1附近处出现了一个归属于蛋白质的酰胺Ⅲ 代表峰, 进一步说明含盐条件下细胞会分泌更多的蛋白类物质来应对高盐环境, 促进EPS产量。
由于EPS样品中荧光信号较强, 拉曼光谱测试会受到影响, 出现较多小杂峰, 也导致其二维拉曼光谱的清晰度较差。 由图10(a, b)拉曼二维相关光谱结果可知, 出现多个独立波数的相关变化(ν 1, ν 2)。 表1纵向数值代表独立波数ν 1, 横向数据代表独立波数ν 2, 对角线中的+代表同步图中自相关峰均为正, 强弱为2 969 cm-1> 1 196 cm-1> 762 cm-1> 844 cm-1> 1 445 cm-1> 1 003 cm-1> 1 063 cm-1; 交叉峰也均为正向, 通过对两个独立波数的变化(表1右上方)比较, 峰波数变化顺序为1 445 cm-1> 2 969 cm-1> 1 003 cm-1> 1 063 cm-1> 844 cm-1> 762 cm-1> 1 196 cm-1, 1 445 cm-1附近与蛋白相关的的— CH2剪切振动峰最先受到影响, 进一步表明蛋白类物质在Fe3+还原中显著变化。
 | 图10 3%盐度体系下Raman分析生成的2D-COS图 (a): 同步图; (b): 异步图Fig.10 2D correlation contour maps by Raman analysis under 3% salinity system (a): Synchronous; (b): Asynchronous |
| 表1 3%盐度体系下同步(Ф )和异步(Ψ , 括号中)相关等高线图中各交叉峰的标志 Table 1 Signs of each cross-peak in the synchronous (Ф ) and asynchronous (Ψ , in the Brackets) correlation contour maps of 3% salinity |
综上所述, 多种光谱技术的联合分析发现, XMS-1 Fe3+还原结束后, EPS中色氨酸、 络氨酸蛋白类物质减少, 蛋白、 多糖等能对金属离子氧化还原的功能基团都减弱。 3%盐度下, XMS-1 Fe3+还原结束后, EPS变化更为显著, 色氨酸、 络氨酸蛋白类物质减少但含量高于对照组, 蛋白多糖等能对金属离子氧化还原的功能基团在减弱的同时, 有新的功能基团出现, Raman光谱中出现新的蛋白酰胺Ⅲ 特征峰, 表现出更强的Fe3+还原作用。 Raman光谱中3%盐度下EPS中出现了一个新的归属于蛋白质的酰胺Ⅲ 代表峰, 表现出更强的Fe3+还原作用。
耐盐希瓦氏菌XMS-1在1%~4%盐度下生长良好, 对Fe3+还原有明显的促进作用, 其中3%盐度下Fe3+还原率最高, XMS-1会分泌更多的EPS以应对盐度的胁迫。 EPS中蛋白含量占80%。 5%以上的盐度会抑制XMS-1的生长, Fe3+还原率下降。 3D-EEM表明, EPS中色氨酸和酪氨酸类蛋白在Fe3+还原过程中峰强减弱, 参与了Fe3+还原过程, 同时2D-COS中发现色氨酸类蛋白优先发生变化且变化显著, 在Fe3+还原过程中有更强的作用。 FTIR和Raman鉴定了EPS在Fe3+还原过程中的官能团, 显示EPS中含有羧基、 羟基、 氨基和羰基等金属离子氧化还原功能基团; 盐度作用下, 蛋白酰胺类特征峰比多糖特征峰变化明显, 其中含O-和N-基团是Fe3+还原的过程中的有效功能基团。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|