作者简介: 陈 蕊, 1995年生, 黑龙江八一农垦大学信息与电气工程学院硕士研究生 e-mail: chenrui_0118@sina.com
为建立快速近红外光谱定量分析模型, 特征波长筛选是提高定量分析预测精度较为有效的方法之一。 它能够筛选出有效波长信息, 减少数据冗余、 提高数据有效性。 随机森林(RF)作为一种集成算法, 可根据计算特征重要性进行特征筛选。 RF将基于袋外数据(OOB)的平均精度下降(MDA)方法计算均方误差平均值作为特征重要性结果, 通过设置特征重要性阈值筛选特征变量构成特征波长子集, 但该阈值范围的设定无理论依据, 因此需要对特征重要性阈值范围进行探究。 另一方面, 由于RF的随机特性, 特征波长子集中可能包含无效甚至是干扰变量, 并不能保证所选变量的有效性。 故而进一步提出RF-iPLS波长筛选方法。 区间偏最小二乘法(iPLS)筛选出的特征波长多为连续特征波段的特性, 对特征波长子集划分区间, 弥补RF因自身随机性造成的无效变量问题; 同时, RF筛选的离散特征波长解决了iPLS筛选的连续波段中含冗余信息的问题。 为了说明RF-iPLS算法的合理性, 特征子集经过蒙特卡洛(MC)方法500次样本特征采样后, 构建RF-MC-iPLS算法。 虽然RF-iPLS与RF-MC-iPLS算法结构接近, 但运行时间缩短了11.12%, 结果说明RF-iPLS算法在预测模型中的特征波长筛选是有效的, 且具有较低的时间复杂度。 为了进一步验证改进的RF-iPLS算法的有效性, 应用一组公开谷物蛋白质近红外光谱数据, 建立PLSR模型, 并与全谱的PLSR模型以及基于不同波长筛选方法的PLSR模型进行比较。 实验结果表明, 相比于全谱的117个波长, RF-iPLS优选出12个特征波长, 建模集的RMSEC从2.61降到0.64, 预测精度提升了约75.5%, 预测集的RMSEP从2.63降到0.69, 预测精度提升了73.8%, 极大地提高了预测精度且预测结果最优, 说明RF-iPLS是一种有效的特征波长筛选方法, 可以简化近红外光谱定量分析模型的复杂度并实现高效降维。
In the rapidly establishing quantitative analysis model of near-infrared spectroscopy, feature wavelength selection is one of the more effective methods to improve prediction accuracy. Through selecting effective information, redundant data is reduced, and the effectiveness of the data set is improved. Random Forest (RF) is an integrated algorithm. The feature importance of spectroscopy wavelength can be calculated by using RF. And the mean square error average value is used as the feature importance result based on the mean decrease accuracy (MDA) method of Out-of-Bag data (OOB). The feature variables are selected to form the feature wave subset by setting the feature importance threshold. However, there is no theoretical basis for setting the threshold range. So it is necessary to explore the range of feature importance thresholds. On the other hand, due to the random characteristics of RF, invalid or even interfering variables may be included in the characteristic wavelength subset, and the selected effectiveness variables cannot be guaranteed. Therefore, the RF-iPLS feature wavelength selection algorithm is further proposed.The feature wavelength subset is divided into intervals by interval partial least squares (iPLS), which makes up for the problem of invalid variables caused by RF randomness and redundant information by iPLS. In order to illustrate the rationality of the RF-iPLS algorithm, the RF-MC-iPLS algorithm is constructed using by Monte Carlo (MC) method. The comparison feature subset is generated after 500 samples.Although the structure of RF-iPLS is similar to that of RF-MC-iPLS, its running time is shortened by 11.12%. The results show that the feature wavelength selection of the RF-iPLS algorithm is effective and has low time complexity in the prediction model. Furthermore, to verify the algorithm’s effectiveness, RF-iPLS was applied to grain protein near-infrared spectroscopy data sets and PLSR models were established. It is compared with the full spectrum PLSR and PLSR models based on different wavelength selection methods. The results show that compared with 117 wavelength points of the full spectrum, RF-iPLS selects 12 feature wavelength points. The RMSEC of the modeling set is reduced from 2.61 to 0.64. The prediction accuracy is improved by about 75.5%. The RMSEP of the prediction set is reduced from 2.63 to 0.69, and the prediction accuracy is improved by 73.8%. The prediction accuracy and optimal prediction results show that RF-iPLS is an effective feature wavelength selection method, and it can simplify the complexity of the near-infrared spectral quantitative analysis model and achieve efficient dimensionality reduction.
近红外光谱分析是基于近红外光照射样品, 得到由含氢基团的倍频和组频吸收峰组成的样品分子特征信息。 通常这些吸收峰较弱且重叠严重, 如果直接用全谱建模, 不仅某些光谱区域与待测组分相关性弱, 而且相邻的波长相关性较高, 包含大量冗余信息[1]; 同时, 近红外光谱存在数据维度高和高频噪声等问题, 直接采用全谱建模不但增加建模复杂度, 同时也会影响模型的预测精度和效率。 因此, 为了剔除不相关变量, 降低数据维度, 简化模型, 特征波长筛选成为提升建模质量的关键环节[2, 3]。
随机森林(random forest, RF)是由Leo Breiman和Adele Cutler[4]提出的能够处理非线性数据、 解决回归和分类问题的高度灵活的机器学习方法, 还可以根据其特征重要性度量进行特征选择[5]。 RF计算特征重要性主要有基于基尼系数以及基于袋外数据(out of bag data, OOB)两种方式。 Nicodemus[6]等发现, 基于基尼系数计算特征重要性多用于离散数据中, 且特征重要性结果与特征变量的顺序选择相关联, 并对离散特征的筛选存在偏差。 宋述芳[7]等发现基于OOB计算特征重要性可以直接度量每个特征变量对模型准确率的影响程度, 不存在偏差问题, 对处理微阵列数据, 尤其是光谱数据有明显的优势。 近年来, 基于RF计算特征重要性的波长筛选方法已被运用到近红外光谱处理中, 王其滨[8]等采用基于OOB误差进行特征重要程度衡量, 剔除近红外光谱中的噪声和冗余信息后, 结合直接正交信号模型传递方法对光谱进行校正处理, 发现能够实现去除光谱噪声的同时, 还可以保留光谱中的有效信息, 能够较好地完成近红外光谱的模型传递。 秦玉华[9]等通过基于OOB准确率作为改进的RF重要性度量方法, 选出对外界变化敏感较低的特征波长变量, 建立偏最小二乘回归(partial least square regression, PLSR)模型预测不同水分和温度下的烟叶中总糖含量, 与全波长和去水分波段相比, RF波长优选可以有效地去除对外界变化敏感的波长, 提升模型预测精度且更加简化稳定。
虽然基于RF特征重要性能够筛选出特征波长, 但存在两方面的不足 : (1) 基于RF计算特征重要性筛选特征波长时, 对特征重要性阈值的设定在现有研究中没有可参照的理论依据, 因此需要对其阈值范围进行探究; (2) 由于RF具有随机抽样和随机特征选取[10]的双重随机特性, RF特征重要性筛选的波长存在随机性现象, 可能存在无效变量, 且无法保证选择出一个规模较小的优化特征子集[11]。
因此, 针对上述两点, 通过采用RF计算特征重要性筛选特征波长, 构成特征子集, 再结合区间偏最小二乘法(interval partial least square, iPLS)对特征子集进行二次波长筛选, 提出随机森林区间偏最小二乘(RF-iPLS)算法, 并将该算法应用于一组公开的谷物蛋白质近红外光谱数据, 分别利用RF和RF-iPLS进行特征波长筛选, 建立回归模型预测谷物蛋白质含量, 与连续投影算法(successive projection algorithm, SPA)、 遗传算法(genetic algorithm, GA)等波长筛选方法对比。 此外, 结合蒙特卡洛(Monte Carlo, MC)方法[12], 将特征子集经过500次样本特征采样[13]后, 构建RF-MC-iPLS算法, 以验证RF-iPLS算法筛选特征波长的有效性。
RF-iPLS算法的有效性验证试验采用的近红外光谱数据集为谷物蛋白质CGL_NIR, 数据来源http://eigenvector.com/resources/data-sets/。 该光谱数据集包含231个谷物样本, 117个光谱波长, 样本光谱波长范围1 104~2 495 nm。 将231个谷物样本随机划分为70%建模集和30%预测集, 建模集包含161个样本, 预测集包含70个样本。 以谷物蛋白质的含量作为因变量进行近红外光谱数据波长选择建模预测分析。 使用Python语言调用scikit-learn工具包搭建RF及PLS的回归模型。
RF通过抽样放回(Bootstrap)的方法从原始样本集N个样本中有放回地随机抽取n个样本, 原始样本集的2/3样本组成决策树的训练集, 剩余1/3未抽取的样本构成OOB, 用于计算特征重要性[14]。 由于RF的双重随机性, 仅使用特征属性在RF决策树中的出现频率来体现特征重要性并不可取, 因此, 为了更准确地反映特征的重要性, 选择基于OOB数据的平均精度下降(mean decrease accuracy, MDA)方法计算均方误差平均值作为特征的重要性。 假设RF中有T棵决策树, 则基于OOB数据的MDA方法计算特征变量重要性过程如下:
(1)对于每棵决策树tm(m=1, …, T), 输入OOB数据的矩阵XOOB, 预测结果为Yp, 预测值Yp与真实值Y的均方误差ε OOBm=mse(Yp-Y)2。
(2)OOB数据的其他特征变量保持不变, 只打乱XOOB中的第i列特征变量顺序重新排列, 即
(3)特征变量
(4)重复(1)— (3), 遍历整个随机森林模型, 得到特征变量
通过RF特征重要性进行特征波长的初选, 得到的波长对于因变量变化最为敏感; 同时, 因模型输入的变量存在随机性, 筛选出的特征波长还存在一定的冗余, 难以保证特征波长筛选的有效性。 iPLS选择的特征波段区间建立的PLS模型效果较好, 但选择的波长较多且不能选取离散特征波长, 连续特征波段中依然存在信息冗余[15]。 RF特征重要性筛选的离散特征波长解决了iPLS筛选的连续波段中含冗余信息的问题, 而iPLS通过对特征波长子集划分区间, 弥补了RF因自身随机性造成的无效变量问题。 因此, 提出RF-iPLS相结合的二次特征波长筛选方法, 对RF筛选出的特征子集进行改进, 结合iPLS方法, 将特征子集划分为n个子区间, 比较各个区间PLS模型的RMSEC的值, 将最小的RMSEC对应区间的波长作为最优特征波长子集, 消除特征波长的随机性, 避免无信息变量及干扰噪声。
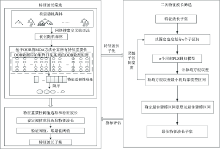
RF-iPLS算法流程如图1所示。 RF-iPLS算法主要为根据RF特征重要性筛选特征波长子集以及利用iPLS算法消除特征波长的随机性并得到最优特征子集的过程。 RF-iPLS算法的具体方法和步骤如下:
(1)建立初始RF模型, 然后使用网络搜索交叉验证方法优化RF模型, 利用式(1)对全光谱进行特征重要性计算并降序排序;
(2)设置特征重要性阈值, 若特征变量重要性大于此阈值, 则提取这部分变量, 并将特征变量按照特征重要性结果从大到小依次排列, 构成特征波长子集;
(3)将特征波长子集通过iPLS进行二次波长筛选, 调整子区间宽度, 将离散特征波长组合成局部连续特征波段, 建立局部PLSR回归模型, 以寻找RMSEC最小为目标进行iPLS二次优选特征波长。
近红外定量分析模型中, 模型评价包括建模集和预测集样本对模型的评价, 常用的评价指标包括建模集均方误差RMSEC和决定系数
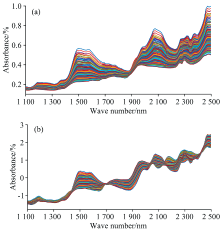
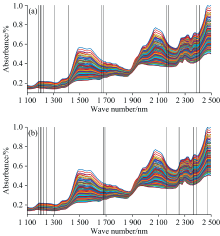
图2为231个谷物样本在1 104~2 495 nm范围内的近红外光谱图。 由图2(a)可见, 谷物蛋白质近红外光谱存在明显的基线漂移和噪声干扰引起的不平滑等问题。 为了在全光谱区域内校正光谱基线、 消除相关噪声的干扰, 减少光谱数据在一定程度上的线性相关, 利用SNV方法对原始光谱数据进行预处理, 预处理后的近红外光谱图如图2(b)所示。
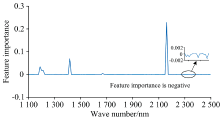
根据上述RF的MDA方法, 对117个全光谱波长计算特征重要性并排序, 图3为RF特征重要性结果与近红外光谱之间的关系, 当光谱特征变量和因变量存在强相关性时, 特征重要性结果为正值[7]。 当某变量的特征重要性值越大时, 说明该变量在RF模型中与因变量存在强相关性[17, 18], 那么优选该变量为特征变量。
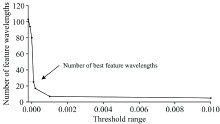
从图3可知, 多个变量的特征重要性结果为零和负值, 表明这些变量与因变量之间存在无关性和弱相关性。 因此, 特征重要性阈值选取的范围为{-0.000 2, -0.000 1, 0.000 0, 0.000 1, 0.000 2, 0.001, 0.01}, 阈值与特征波长筛选的数学关系如图4所示, 可以看出阈值与特征波长个数之间呈现反比例关系, 即特征重要性阈值越接近零, 特征波长个数越多。
对上述不同阈值范围内筛选出的特征波长建立PLSR预测模型, 比较不同阈值范围内的特征波长预测结果。 如表1所示, 随着特征重要性阈值不断增大, 特征波长数和运行时间不断减小, RMSEP的结果变化整体趋势为先减小后增大,
| 表1 不同阈值范围内的特征波长预测结果 Table 1 Prediction results of characteristic wavelength in different threshold ranges |
将经过RF特征重要性筛选出的25个特征波长, 按照特征重要性值的大小对其进行提取并排序, 得到特征波长子集。 采用iPLS算法对该特征波长子集完成二次特征波长筛选, 消除特征子集中因随机性产生的无效波长变量或干扰变量, 筛选出关键特征波长进行建模。 将得到的特征波长子集进行区间划分, 区间数量n从1依次增加到5, 并基于这些区间的特征变量分别构建PLSR模型, 以RMSEC最小为原则筛选最佳的建模区间。 图5为不同区间个数时, iPLS模型的最佳建模区间评估参数的变化趋势。 随着n逐渐增大, RMSEC和RMSEP值呈现先减小后逐渐增加,
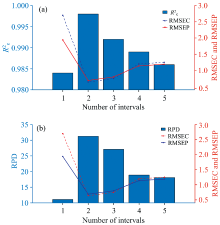 | 图5 不同区间个数时模型评估参数的变化趋势 (a): RMSE与R2; (b): RMSE与RPDFig.5 Variation trend of model evaluation parameters at different interval numbers (a): RMSE and R2; (b): RMSE and RPD |
| 表2 不同区间个数的RF-iPLS优选结果 Table 2 Optimization results of RF-iPLS model with different interval numbers |
根据表2结果可知, iPLS在挑选特征波段的过程中, 区间个数的选择在很大程度上影响RF-iPLS, 且随着区间个数的增加, RF-iPLS模型的计算量随之增大; 同样, iPLS波长筛选时也会受到区间个数的影响, 有时甚至会选入大量的无关信息[21], 导致预测模型效果不佳。 因此, 对RF-iPLS算法, 选择合适的区间个数, 对特征波长信息的筛选及建立良好预测性能的模型至关重要。
2.4.1 波长筛选结果分析
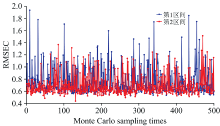
为了说明RF-iPLS算法区间划分的合理性, 将特征波长子集经过MC的500次特征波长样本随机采样后, 如前所述, 按照区间个数为2时进行二次波长筛选。 如图6所示, MC采样次数为131时, RMSEC在第2区间中有最小值, 最小值为0.44, 此时筛选的波长变量数为13个。 13个波长变量分别是2 160、 1 188、 1 212、 1 200、 1 680、 1 308、 2 388、 1 224、 2 364、 2 256、 1 692、 1 248和2 472 nm。
将谷物蛋白近红外光谱数据运用RF-iPLS和RF-MC-iPLS算法, 分别获取到12个和13个特征波长, 构建基于PLSR的预测模型。 结果对比如表3所示, 经过MC对特征波长子集进行波长随机采样后, RF-MC-iPLS的模型中建模集的RMSEC和
| 表3 RF-iPLS与RF-MC-iPLS结果分析 Table 3 Results analysis of RF-iPLS and RF-MC-iPLS |
RF-iPLS与RF-MC-iPLS算法筛选的波长结果对比可知, 二者相同的波长为2 160、 1 188、 1 212、 1 200、 1 680、 1 308、 2 388和1 224 nm, 主要分布在1 110~1 680 nm区域内, 该区域包含蛋白质的氨基N— H键、 水O— H键和醇O— H键的伸缩振动的一级倍频, 碳链C— H键(— CH, — CH2和— CH3基团)的第二组合频和二级倍频等结构光谱信息, 表明这8个特征波长可能与样品中的蛋白质浓度有较高的相关性; 同时, RF-iPLS与RF-MC-iPLS算法筛选波长的结果存在差异, RF-iPLS算法筛选出1 416、 1 668、 2 412和2 172 nm, RF-MC-iPLS算法筛选出2 364、 2 256、 1 692、 1 248和2 472 nm, 范围在1 248~1 692和2 172~2 472 nm之间, 其中在2 172~2 472 nm区域内, 对应着蛋白质的碳链C— H键的组合频和一级倍频、 C=O的伸缩振动一级倍频以及N— H伸缩振动与AmideⅡ 的合频, 基本涵盖RF-iPLS算法筛选的上述4条波长; RF-MC-iPLS比RF-iPLS算法筛选的特征波长多1条且预测结果略优于RF-iPLS模型, 原因可能是该区域中某些波长与样品中的谷物蛋白质浓度特别相关。 总体来看, 通过设置MC对特征波长子集进行随机采样, 验证了RF-iPLS算法能够筛选出有效波长, 提高模型预测能力; 同时, RF-iPLS算法筛选出的波段与图7(b)近红外光谱曲线吸收峰的位置基本一致, 符合Ridgway等[22]的论点, 即光谱曲线上的凸起区可能对蛋白质含量预测有实际意义。
 | 图7 特征波长分布图 (a): RF-iPLS; (b): RF-MC-iPLSFig.7 Feature wavelength distribution (a): RF-iPLS; (b): RF-MC-iPLS |
2.4.2 定量分析的结果比较
为验证RF-iPLS模型的性能, 分别将全光谱和GA, SPA, RF和iPLS方法筛选的特征波长作为PLSR模型的输入。 各个波长筛选算法的特征波长数量和预测结果如表4所示, 全光谱模型的特征波长数最多, 模型运行时间最长, 预测效果最差。 相较于全光谱模型FULL-PLSR, 5种特征波长筛选算法都有效地提高模型的预测能力, 结合模型预测结果、 建立模型的波长数和谷物蛋白真实值与预测值拟合关系来看, 显然, 在近红外光谱分析过程中, 特征波长筛选是可行的、 有必要的。
| 表4 不同波长筛选的预测结果比较 Table 4 Comparison of prediction results of different wavelength selection |
模型预测结果如表4所示, 在波长筛选方法对比中, 虽然GA和SPA的建模集效果RMSEC和
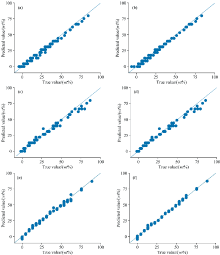
全光谱、 GA、 SPA、 RF、 iPLS和RF-iPLS方法筛选的特征波长建立谷物蛋白质预测模型, 模型的真实值与预测值的散点拟合图如图8所示。 从图8(f)中可以更直观地看出, 六个模型中RF-iPLS模型的预测效果最好, 数据点集中在拟合直线的两侧, 而其他模型的数据点分布较分散, 由此可见, 近红外光谱结合RF-iPLS算法筛选特征波长并建立预测模型, 具有很高的预测准确度, 可以实现谷物中蛋白质含量的定量分析。
RF-iPLS特征波长筛选方法, 将改进的RF特征重要性运用到近红外特征波长筛选中, 并结合iPLS算法将孤立的特征波长联合起来成为特征区间, 完成二次波长优选, 以解决RF特征重要性筛选出的特征子集中可能包含干扰变量或无效变量的问题。 将RF-iPLS应用于一组谷物蛋白质近红外光谱数据集, 通过比较不同RF特征重要性阈值, 确定波长特征子集, 进一步结合iPLS方法完成二次波长筛选, 建立预测模型, 试验证明RF-iPLS模型预测精度较优, 能够实现谷物蛋白质含量的快速定量分析; 并结合MC方法, 将特征子集经过500次样本随机采样后建立模型, 验证了RF-iPLS算法的有效性。 为了进一步探究RF-iPLS波长筛选方法的有效性, 通过比较不同波长筛选方法, 相较于全谱、 GA、 SPA和iPLS的PLSR模型, RF-iPLS极大的简化模型复杂度, 提高了预测精度。 说明了RF-iPLS能够有效筛选近红外光谱特征波长, 提高预测精度, 简化模型的复杂度, 提升模型运行效率, 是一种有效的特征波长筛选方法。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|