作者简介: 路文静, 女, 1996年生, 北京工业大学环境与生命学部硕士研究生 e-mail: L2384130472@163.com
外泌体(Exosome)是直径大小为30~150 nm的膜性囊泡, 包裹DNA/RNA, miRNA, 蛋白质和脂质等多种物质并参与微环境中的生物信息传递, 是理想的癌症生物标志物, 在液体活检领域具有重要的应用潜力, 有望成为癌症快速检测的手段之一。 表面增强拉曼光谱(SERS)是分子振动光谱, 可从分子水平上探测物质的精细结构和信息变化, 具有“指纹图谱”的特征。 采用差速离心结合超速离心的方法获得乳腺癌细胞来源的外泌体, 以金溶胶为增强基底, 收集外泌体及其母细胞的SERS图谱, 结合多元统计分析, 进行乳腺癌细胞的快速鉴别与区分。 研究结果表明, 乳腺癌细胞及其外泌体在500~1 600 cm-1波段范围内有特征拉曼信号, 采用非标记检测所获得的图谱信息是样品“whole-organism fingerprint”整体信号的呈现。 根据外泌体的拉曼表型并结合OPLS-DA分析, 能够100%分辨3种不同类型的乳腺癌细胞。 单细胞SERS检测联合PCA-LDA分析, 区分乳腺癌细胞的准确率为83.7%。 通过比较乳腺癌细胞及其外泌体的拉曼特征图谱发现, 二者在拉曼谱图的波数高度表现一致, 但是外泌体在特征波数上显著增强。 具体体现为在506~569、 1 010~1 070 cm-1等波段二者存在相似性, 但外泌体在735、 963和1 318 cm-1等处的特征信号显著高于细胞。 分析认为外泌体结构比细胞更为简单, 核酸、 蛋白质等生物大分子信息更容易被表征。 同时也提示了通过SERS检测外泌体实现快速鉴定乳腺癌的可行性。 采用非标记、 直接检测, 建立了快速检测单细胞及外泌体的SERS分析技术, 结合多元统计分析能够快速鉴别不同类型的乳腺癌细胞, 并从拉曼组学角度探究了外泌体与母源细胞的关系。 该方法具有非标记、 快速、 灵敏、 准确、 简便的优势, 为乳腺癌的体外快速诊断与筛查提供有效的技术手段, 为临床应用奠定基础。
Exosomes are nano-sized phospholipid bilayer-enclosed vesicles secreted by all cells into the extracellular milieu. Released exosomes contain cell-specific proteins, membrane lipids, mRNA, DNA and microRNA that can perform versatile roles in normal or diseased processes. Exosomes are ideal biomarkers of cancer, which have important application potential in liquid biopsy and are expected to become one of the means for rapid cancer detection. Surface enhanced Raman spectroscopy (SERS) is a molecular vibration spectrum, which can detect the fine structure and information changes of substances at the molecular level, and has the characteristics of a “fingerprint spectrum”. In this study, the exosomes were isolated via differential centrifugation combined with ultracentrifugation of the supernatants of breast cells. The SERS profiles of breast cancer cells and their exosomes were collected with colloidal Au nanoparticles as the enhanced substrate, and multivariate statistical analysis was used to identify and distinguish breast cancer cells. The results showed that breast cancer cells and their exosomes had characteristic Raman signals in the range of 500~1 600 cm-1. Their Raman phenotypes obtained by non-labeled detection were the presentation of all the signals of the“whole-organism fingerprint” of the sample. The accuracy rate reached 100% by using exosome-SERS detection and OPLS-DA analysis. Single-cellular SERS detection combined with PCA-LDA analysis showed that the accuracy of differentiating breast cancer cells was 83.7%. Breast cancer cells and their exosomes showed similarity at the bands of 506~569 and 1 010~1 070 cm-1, but the characteristic Raman peaks of exosomes at 735, 963 and 1 318 cm-1 were significantly higher than those of cells. It may be because the structure of exosomes is simpler than that of cells, and the information of biological macromolecules such as nucleic acids and proteins can be characterized more easily, indicating the feasibility of rapid identification of breast cancer by detecting exosomes by SERS technology. In summary, this study established a non-labeling and direct detection method for rapidly detecting single cells and their exosomes by SERS analysis. Combined with multivariate statistical analysis, different types of breast cancer cells could be quickly identified, and the relationship between exosomes and maternal cells was explored from the perspective of Raman omics. This method has the advantages of non-labeling, rapidness, sensitivity, accuracy and simplicity, which provides an effective technical means for rapid diagnosis and screening of breast cancer in vitro, and lays a foundation for clinical application.
外泌体(exosome)是由细胞主动释放的小膜泡, 直径大小为30~150 nm, 包含大量由母细胞分泌的功能性蛋白质、 核酸、 脂类等多种生物活性物质[1, 2]。 不同细胞来源的外泌体其内容物不一样, 行使不同的功能。 有研究表明, 肿瘤细胞来源的外泌体含有肿瘤特异性的分子, 成为一种新兴的生物标志物, 可以作为癌症检测的一个重要指标, 对于诊断和预后有着重要应用价值[2]。 外泌体是母细胞的缩影。 与常规的单标志物检测肿瘤相比, 外泌体检测是综合性信息的体现, 能有效反映母细胞的生理、 病理特征。 因此, 外泌体在液体活检领域具有重要的应用潜力, 有望成为癌症快速检测的手段之一。
表面增强拉曼光谱(surface enhanced Raman spectroscopy, SERS)是分子振动光谱, 可从分子水平上探测物质的精细结构和信息变化, 具有“ 指纹图谱” 的特征[3]。 作为一种非侵入式的检测方法, 拉曼检测对样品损伤小, 能快速、 灵敏地获取分子的丰富指纹信息, 同时测量不受水的影响, 在生命科学研究领域具有广阔的应用前景。 最近有研究报道, 采用SERS技术对胞外囊泡和外泌体进行快速检测, 从而达到鉴别癌症的目的[4, 5, 6, 7, 8]。 SERS检测主要有标记检测和非标记检测。 Fan等通过将拉曼报告分子与赖氨酸偶联实现了外泌体拉曼信号的精确控制, 提高了检测的灵敏度, 该生物传感器能够成功监测小鼠模型中的术后肿瘤复发, 并将癌症患者与健康受试者区分开[4]。 Li等构建的高灵敏度SERS 探针检测系统, 可以直接定量分析真实样品中的特定外泌体, 对于胰腺癌的早期诊断具有巨大潜力[5]。 然而, 标记的方法是基于特征标记的检测, 基底合成过程复杂, 因此限制了临床应用, 尤其是即时检测。 有研究表明, 采用无标记SERS检测能够收集外泌体的信号, 从而实现对不同来源外泌体的鉴别和分类, 采用的手段有单粒子自动拉曼捕获分析的SPARTA系统[6], 磁性表面增强拉曼散射平台[7]等。 Wang等开发的微流控拉曼生物芯片则是在原位分离和分析外泌体, 已成功应用于临床血清样本中外泌体的检测, 这种新型工具具有作为前列腺临床外泌体分析工具的潜力[8]。
目前, 大多数的研究报道是通过分别检测外泌体或者细胞来实现癌症的鉴别。 外泌体由母细胞分泌而来, 二者之间在光谱上是否有一些相关性尚未见文献报道。 本研究将以此为出发点, 首先采用非标记、 直接检测的方法, 收集乳腺癌细胞及其外泌体的SERS信号, 同时结合多元统计分析, 建立不同类型乳腺癌细胞及其外泌体的判别模型, 并从拉曼组学角度探究外泌体与母源细胞的相关性, 为乳腺癌的快速诊断与筛查提供有效的技术手段, 为临床应用奠定基础。
细胞培养: 人正常乳腺上皮细胞MCF-10A和乳腺癌细胞MCF-7, MDA-MB-231购自国家实验细胞资源共享平台(National Infrastructure of Cell Line Resource, NICR)。 MCF-10A和MCF-7在DMEM/High Glucose培养基中培养, MDA-MB-231则用RPMI 1640培养基培养, 并补充10%无外泌体的胎牛血清和1%青霉素-链霉素, 37 ℃ 5% CO2培养箱中培养。
纳米金溶胶(colliodal Au nanoparticles, AuNPs)采用化学还原法合成[9], 其在532 nm处有最大吸收峰, 金颗粒的平均粒径约为(50± 12) nm。 以金溶胶为增强基底, 采用显微共聚焦拉曼光谱仪(Renishaw, inVia, UK)收集细胞和外泌体的SERS信号。
单细胞SERS检测: 制备细胞悬液滴加至放有石英玻片的培养皿中, 于37 ℃, 5% CO2条件下培养4 h, 待细胞贴壁后补加新鲜的培养液2 mL。 待细胞密度长至70%左右时, 取出石英玻片并用PBS清冼3遍, 滴加100 μ L AuNPs进行检测。 检测条件为785 nm, 1 200 l· mm-1光栅, 1% (1 mW)激光功率, 积分时间20 s, 积分1次, 光谱分辨率为2 cm-1。
外泌体SERS检测: 按常规方法培养细胞, 待细胞长到60%~70%后, 更换新鲜DMEM培养基, 添加10%无外泌体胎牛血清和1%青霉素-链霉素, 在恒温37 ℃、 5% CO2细胞培养箱中连续孵育48 h。 待细胞铺满瓶壁90%后, 取5 mL细胞培养上清液。 按照差速离心结合超速离心方法进行外泌体收集[10]。 取3 μ L外泌体提取液与3 μ L AuNPs混合后滴加到石英玻片上立即检测。 检测条件为: 功率0.5% (0.5 mW) , 积分时间为10 s, 其余同上。
原始数据用激光共聚焦拉曼光谱仪(Renishaw)配置的Wire 4.1软件(inVia)进行预处理, 从原始光谱中去除宇宙射线, 并进行基线校正, 以减少荧光背景和仪器噪声的干扰。 通过Savitsky-Golay平滑算法对数据进行平滑处理, 采用Origin (2018)软件进行绘图, 使用SPSS 26.0和SIMCA 14.1软件进行多元统计分析。
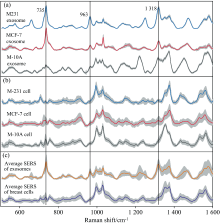
采用差速离心结合超速离心的方法获得细胞来源的外泌体, 呈圆盘状, 平均粒径主要为110 nm, 与文献报道一致[7, 8]。 如图1所示, 以AuNPs为增强基底, 将外泌体与AuNPs混合后直接收集其信号, 在10s内可以获得其拉曼图谱, 分别采集了乳腺癌细胞MDA-MB-231、 MCF-7和乳腺上皮细胞M-10A来源的外泌体SERS图谱35张[图2(a)]。 在图2(a)中, 彩色实线为35组数据的平均值, 灰色区域为标准偏差, 三组外泌体的标准偏差分别为2.13%、 3.82%和3.57%, 说明该方法具有较高的重现性。 外泌体在500~1 600 cm-1波段范围内有特征拉曼信号, 特征峰清晰显著, 不同外泌体的峰形、 峰位置不同, 说明该方法具有较高的特异性和灵敏度。 由于所采用的共聚焦显微拉曼光谱仪的光斑面积为1.8 μ m× 30 μ m, 能覆盖整个外泌体, 因此收集的拉曼光谱信号是外泌体整体信息的呈现。 同样, 分别采集了乳腺癌细胞 MDA-MB-231、 MCF-7和乳腺上皮细胞M-10A的SERS图谱40、 48和59张, 绘制其平均图谱, 标准偏差分别为9.14%、 12.50%和8.37%[图2(b)]。
为了比较外泌体与细胞的整体特征, 本实验共采集255 780组数据。 将外泌体与细胞的拉曼数据分别取平均值, 绘制SERS平均图谱, 如图2(c)所示, 外泌体与细胞的误差分别是9.97%和11.34%。 从图2中可以清晰地看到, 外泌体在650~760 cm-1波数范围内, 特征峰信号更强, 峰更加尖锐, 尤其是735 cm-1, 该特征峰归属于核酸振动。 除此之外, 特征峰963和1 318 cm-1在外泌体中的信号也较强。 这可能是因为外泌体结构相较于细胞而言更为简单, 生物大分子的信息更容易被展现。 综上, 采用直接检测的方法简单、 无需标记, 显著缩短了SERS信号的收集时间, 短短几十秒即可获得细胞和外泌体的整体生化信息, 且信号稳定, 是所采集
样品的整体信号的呈现, 为肿瘤快检提供一种新的研究思路。
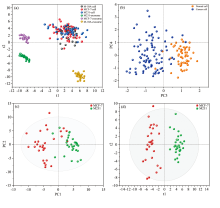
将收集的所有SERS数据进行正交偏最小二乘判别分析(orthogonal partial least squares discrimination analysis, OPLS-DA)和主成分线性判别分析(principal component analysis-linear discrimination analysis, PCA-LDA), 将光谱数据转化成模型更直观地呈现。 OPLS-DA和PCA-LDA均是无监督学习算法。 OPLS-DA将变量从255 780减少到5个预测主成分(t), 占总变量的85.17%。 以t1(18.21%)和t2(18.41%)绘制得分散点图, 如图3(a)所示每一个点均代表一个样本, 可以看出三株细胞样本与外泌体样本可以完全区分开来。 同时, 三株细胞来源的外泌体也能够100%区分, 说明将外泌体与细胞的SERS数据混合时, 仍然具有很强的区分度。 从图3(a)中可以看出, 三株细胞样本聚集在了一起, 提示三株细胞的相似性高, 共性更强。
采用PCA分析, 对非肿瘤细胞(M-10A细胞)和癌细胞(MCF-7和M231)进行进一步的鉴别, 提取特征值大于1的主成分, 以PC3和PC4作图, 可以看出乳腺癌细胞和乳腺上皮细胞被有效的聚集成两类, 说明该模型能够将乳腺癌细胞与正常细胞区分开来, 准确率为83.7%[图3(b)]。 乳腺上皮细胞主要聚集在PC3大于0且PC4小于1的区域, 根据PC3载荷图提取特征信息, 发现乳腺上皮细胞与乳腺癌细胞在732~738、 753~761、 996~1 037、 1 317~1 367和1 567~1 568 cm-1等波段存在差异。
为了进一步区分不同类型的癌细胞, 采用PCA-LDA和OPLS-DA分析, 对两株乳腺癌细胞进行建模判别。 以60组拉曼图谱作为训练集建立分类模型, 结果如图3(c)和(d)所示, PCA-LDA和OPLS-DA模型中的大部分样本落在95%的置信区间(椭圆区域), 只有少数几个样本落在置信区间以外。 但在PCA-LDA模型中, 有一些样本存在重叠, 而OPLS-DA模型则将MCF-7和M231细胞100%进行了归类。 以18组MCF-7细胞和10组M231细胞的SERS数据作为测试集, 在上述建立的判别模型中进行Cross-validation验证。 PCA-LDA和OPLS-DA判别模型对MCF-7细胞和M231细胞的判别准确度分别为82.12%和85.71%(表1), 表明两个模型均适用于不同类型乳腺癌细胞的鉴别, OPLS-DA模型的灵敏度和特异度更高。 该结果说明OPLS-DA模型更适合对不同类型的癌细胞进行判别, 同时也提示将SERS技术与OPLS-DA分析相结合有望成为揭示细胞异质性规律的有利工具。
| 表1 PCA-LDA和OPLS-DA模型的判别结果 Table 1 Classification results of PCA-LDA and OPLS-DA model |
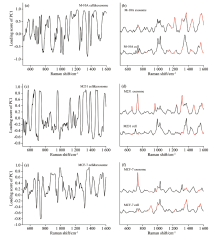
为进一步探索外泌体与其母源细胞SERS图谱的相关性, 将每株细胞及其外泌体进行PCA分析, 提取主成分, 根据PC1载荷图信息, 选取Loading value大于等于0.7的值进行特征提取[图4(a, c, e)], 得出三株细胞与其外泌体的差异波段, 如图4(b, d, f)中的红色标记。 对比分析图4(b, d, f), 不难发现, 在650~760、 1 200~1 300和1 400~1 500 cm-1等波段, 三组外泌体的SERS信号均相较其细胞更强, 峰更典型, 峰归属见表2。 说明这些波段所代表的核酸、 蛋白质等信息在外泌体里更容易被表征, 这可能与外泌体的结构更为简单有关。
| 表2 乳腺细胞与其外泌体的特征峰归属与比较 Table 2 Attribution and comparison of the characteristic peaks of breast cells and their exosomes |
细胞与外泌体之间存在许多相似的波段, 如506~569、 580~638、 748~847 cm-1等[图4(b, d, f)黑色标记], 这些波段中既有代表核酸的特征峰, 也有代表蛋白质、 脂质的特征峰, 峰归属见表2, 这些特征峰不是很强, 所代表的物质可能属于细胞和外泌体的基础组成成分, 这些相一致的特征峰说明了外泌体的“ 母源性” 。
外泌体作为肿瘤新型标志物, 在SERS图谱上呈现出与其母源细胞相似的一些信息, 这些信息主要归属于核酸、 蛋白质、 脂质等物质。 外泌体与细胞在650~760、 1 200~1 267、 1 300~1 336 cm-1等波段呈现出差异。 通过对乳腺癌细胞和外泌体的SERS快速检测与比较, 可以将二者之间的“ 相关性” 更直观地呈现出来, 为探究外泌体在肿瘤微环境以及肿瘤的侵袭与转移中的作用提供新的视角。
以金溶胶为增强基底, 采用非标记、 直接检测的方法对乳腺癌细胞及其外泌体进行SERS图谱的采集, 该方法简单、 灵敏、 快速、 无需标记, 收集到的图谱信号稳定, 是所采集样品的整体信号的呈现。 结合多元统计分析, 可以实现不同类型乳腺癌细胞的分类和鉴别。 进一步对外泌体与其母细胞SERS图谱进行对比分析, 发现二者在650~760、 1 200~1 300和1 400~1 500 cm-1等波段存在差异; 在506~569、 580~638、 748~847和1 010~1 070 cm-1等波段存在相似性。 这些“ 异同” 信息提示SERS技术可以从光谱学的角度解析外泌体与母细胞之间的相关性, 进而为探究肿瘤微环境以及癌症转移性提供一种新方法。 外泌体的SERS检测在体外检测方面具有巨大的应用潜力, 可为癌症的快速筛查和早期诊断提供技术指导, 为临床应用奠定基础。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|