作者简介: 何庆元, 1978年生, 安徽科技学院教授 e-mail: heqingyuan1@163.com
品质性状的化学测定操作繁琐且存在破坏性和耗时较长等不足的问题, 光谱测定具有高效、 快速、 成本低等优点, 但测定准确度受到不同仪器以及不同机型的影响。 为了建立和优化快速测定苜蓿样品的粗蛋白(CP)、 粗脂肪(EE)、 酸性洗涤纤维(ADF)和中性洗涤纤维(NDF)近红外漫反射光谱的模型, 更好的测定苜蓿品质性状。 选取了25份苜蓿材料147份试验样品, 采用傅里叶变换近红外光谱技术(NIRS)扫描, 获得扫描光谱范围4 000~10 000 cm-1的光谱值, 软件TQ Analyst v9选用偏最小二乘法(PLS)和OPUS7.0选用定量2方法建立定量模型并优化, 并进一步交叉验证和外部检验评估模型效果。 结果表明利用2种软件建立的模型都能很好的预测CP的含量, 建模决定系数(
The chemical determination method of quality traits is cumbersome, destructive and time-consuming. The spectral determination method has the advantages of high efficiency, speed and low cost, but the accuracy is affected by different instruments and models. In order to establish and optimize the model for rapid determination of crude protein (CP), ether extract (EE), acidic detergent fiber (ADF) and neutral detergent fiber (NDF) using near-infrared diffuse reflectance spectra of alfalfa samples, and better determine the quality traits of alfalfa. A total of 147 samples of 25 alfalfa materialswere selected. The scanning spectral values of the spectral range of 4 000~10 000 cm-1 are obtained by scanning with Fourier transform near-infrared spectroscopy (NIRS). The software TQ Analyst V9 adopts partial least squares (PLS), and OPUS 7.0 adopts the quantitative 2 methods to establish and optimize the quantitative model and further carry out cross-validation and external test to evaluate the effect of the model. The results showed that the models for determining CP content were also through two software. Two modeling coefficient of determination (
苜蓿因产量高、 蛋白质含量高、 富含奶牛所需的各类营养因子和良好的适口性被称为“ 牧草之王” [1]。 青干草是苜蓿始花期刈割后经自然或人工干燥而成的饲草类型, 是苜蓿最普遍和高效的利用类型。 苜蓿青干草中的营养成分丰富, 但不同品种和茬次的苜蓿营养成分存在显著性差异, 干燥晾晒过程中也可致干草营养价值的改变[2]。 然而, 当前常用的化学测定饲草营养价值(粗蛋白质的凯氏定氮法、 粗脂肪的索氏提取法、 酸性洗涤纤维和中性洗涤纤维的范氏测定法[3])方法操作过程复杂、 耗时, 对操作人员有一定危险性, 并且需要消耗大量试剂, 导致环境的污染。 因此, 寻找一种快速、 准确, 高效检测苜蓿营养价值的方法对促进苜蓿产业的快速发展具有重要意义。
近红外反射光谱(near infrared reflectance spectroscopy, NIRS)分析方法具有方便、 快速、 高效、 准确和成本较低, 不破坏样品、 不消耗化学试剂、 不污染环境, 可同时测定多个组分等优点。 近红外光谱已被广泛开发应用于农产品品质的测定, 欧洲许多发达国家甚至在很多领域内将该技术作为行业产品质量评定的标准技术, 几乎完全取代广泛使用的化学分析方法[4]。 已有研究表明NIRS 技术建立的检测多种饲草饲料粗蛋白(CP)、 中性洗涤纤维(NDF)和酸性洗涤纤维(ADF)含量的模型与常规化学方法的检测精度相当[5]。 有报道将NIRS技术应用于苜蓿进行品质分析的部分研究, 包括建立分析苜蓿的青干草[6]、 草捆[7]的CP、 NDF和ADF以及多种营养品质[8]的模型等。 然而, 品质性状光谱测定模型受到不同仪器以及不同机型的影响, 特别是还未见与苜蓿粗脂肪(EE)含量相关的光谱模型测定方法。 本研究通过光谱测定, 比较不同方法建立苜蓿的粗蛋白、 粗脂肪、 中性洗涤纤维、 酸性洗涤纤维和相对饲用价值的光谱模型, 为快速测定苜蓿品质性状奠定基础。
种植于安徽省五河县秋实草业有限公司25个苜蓿品系[9], 随机区组试验设计3个重复, 在苜蓿始花期收获, 共5茬, 经105 ℃杀青后, 80 ℃烘干至恒重, 用FZ102型植物粉碎机粉碎后过50目筛, 获得苜蓿干粉装袋保存, 进行化学测定和光谱扫描。
样品粗蛋白(CP)和粗脂肪(EE)测定按照国家标准的化学方法GB/T 6432和GB/T 6433。 酸性洗涤纤维(ADF)和中性洗涤纤维(NDF)按范式(Van Soest)测定。
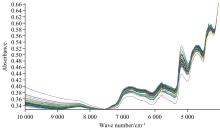
实验中使用Antaris Ⅱ (美国Thermo Fisher公司)红外光谱仪, 漫反射积分球附件。 工作参数: 光谱扫描范围4 000~10 000 cm-1, 扫描次数64, 分辨率4 cm-1。 在样品扫描之前, 仪器预热1 h, 取约50 g干粉装入内径10 cm旋转式石英样品杯, 摇匀使表面平整后放置在光谱仪上进行扫描得到光谱。 样品的近红外光谱见图1。
采用TQ Analyst v9 软件和OPUS7.0软件分别建模, 从147份中选取117份建模, 30份验证。 TQ Analyst v9选用偏最小二乘法(PLS)建立定标模型的化学计量方法, 光谱扫描范围(4 000~10 000 cm-1), 逐步筛选最佳的光谱预处理方法, 根据建模决定系数(
由表1可以看出4种营养成分含量具有较大的变异幅度, 其含量范围能够覆盖4种营养成分的含量, 它们的变异系数较大, 能够很好地代表苜蓿草粉营养成分含量的变化, 除EE峰度和偏度较大, CP、 ADF和NDF峰度和偏度都小于1, 说明样品中CP、 ADF和NDF的含量分布较均匀, 但EE含量主要集中在平均值附近, 并且小值较多。 整体分析数据符合建模要求。
| 表1 化学法测定的苜蓿样品品质性状分析 Table 1 Descriptive statistics of chemical value of alfalfa samples |
2.2.1 TQ Analyst v9建模效果分析
TQ Analyst v9用PLS进行全谱解析, 设置不同性状的范围, 输入化学值, 将光谱调入程序, 对获取的光谱进行预处理, 通过优化建模, 获得预测值, 将预测值与实际值偏差超过实际值5%的样品剔除, 通过逐步优化, 最终获得最佳模型。 其4种品质性状的最佳有效波段范围CP为9 056.08~9 476.49 cm-1, EE为8 585.53~9 931.60 cm-1, ADF为8 867.09~9 923.89 cm-1, NDF为7 239.46~8 987.65 cm-1。 由表2可知采用TQ Analyst v9软件建模决定系数(
| 表2 TQ Analyst v9对苜蓿4种营养成分建模效果 Table 2 Evaluation of the four calibration models and validation using TQ Analyst v9 |
2.2.2 OPUS7.0建模效果分析
表3为采用OPUS7.0优化建模后, 在最佳建模光谱范围和光谱预处理方法下得到的苜蓿4项指标含量校正模型的建模参数和交叉验证效果, 其4种品质性状的最佳有效波段范围CP为4 246.50~6 101.07 cm-1, EE为7 497.90~9 403.20 cm-1, ADF为4 601.30~4 246.50 cm-1, NDF为5 449.80~6 101.70 cm-1。 由表3知: CP、 EE、 ADF和NDF含量的校正模型的
| 表3 OPUS7.0对苜蓿四种营养成分建模结果 Table 3 Evaluation of the four calibration models and validation using OPUS 7.0 |
采用所建立的2种模型分别预测30份苜蓿样品的4种品质性状值, 从表4中可以看出光谱预测平均值与化学测定值符合度良好, 表明所建立的模型具有很好的预测效果。 2种模型的CP决定系数(R2)均超过0.97, 预测CP含量结果与实际测定的化学测定值符合度都很好。 TQ Analyst v9预测的EE值与化学测定值决定系数达0.929 3, 但OPUS7.0预测值和化学测定值决定系数为0.823 4, 因此EE预测应选用TQ Analyst v9模型进行分析。 OPUS7.0预测ADF和NDF的决定系数和PRD均高于TQ Analyst v9, 因此应选用OPUS7.0模型进行分析(图2和图3)。
| 表4 2种软件所建立模型的预测效果 Table 4 Prediction effect of models established by two softwares |
 | 图2 TQ Analyst v9近红外分析品质性状含量值与化学值的相关性Fig.2 Correlation between chemical value and spectral analysis value of TQ Analyst v9 |
 | 图3 OPUS7.0近红外分析品质性状含量值与化学值的相关性Fig.3 Correlation between chemical value and spectral analysis value ofOPUS 7.0 |
近红外光谱技术作为一种间接测量方法, 具有快速、 无损和低成本等优点, 然而模型的预测值不仅受到仪器的性能、 检测环境、 样品化学组成的影响, 而且也受到建模时所采用的导数、 多元散射校正和平滑处理等光谱数据预处理手段的影响[10, 11], 因此优化建模方法, 获得更理想的模型, 可更准确地预测相关组份含量。 本研究通过TQ Analyst v9和OPUS7.0两种建模软件, 优化建模模型, 以期获得更准确的预测值, 实验表明CP在2种建模模型上均能获得良好的效果。 而EE采用TQ Analyst v9建模能获得更准确的预测值, ADF和NDF通过OPUS7.0建模相对准确性更高。 4种品质性状建模效果表明, 分子结构相对简单的蛋白质和脂肪, 采用TQ Analyst v9建模更准确, 而对于分子结构更复杂的纤维素, OPUS7.0的预测效果更好。
比较TQ Analyst v9和OPUS7.0两种软件建立了适合不同品种、 不同茬次苜蓿的近红外反射光谱技术测定苜蓿草粉种品质性状指标的校正模型。 粗蛋白2种建模模型均获得良好效果, 化学值和光谱值决定系数(R2)超过0.97, PRD大于6.0; 粗脂肪利用TQ Analyst v9更佳, 其R2和PRD分别为0.929 3和3.89; 酸性洗涤纤维和中性洗涤纤维用OPUS7.0建模更佳。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|