作者简介: 王琦标, 1991年生, 四川轻化工大学计算机科学与工程学院讲师 e-mail: wangqibiao@suse.edu.cn
快速、 准确检测酒醅酸度, 可显著提高白酒出酒率和成品酒品质。 近红外光谱(NIR)提供了分子的倍频和合频, 即有机物中含氢基团(C—H、 N—H、 O—H)的振动信息, 通常用于样品中含氢化合物的定性和定量分析。 采用NIR能简单、 迅速的测定酒醅酸度, 克服了传统化学分析方法检测周期长、 试剂消耗大、 人为误差等不足。 由于NIR是一种间接分析技术, 如何建立校正模型是准确检测酒醅酸度的关键。 作为深度学习中的典型模型, 卷积神经网络(CNN)具有局部区域连接, 分享权值等优点, 不仅能从复杂的光谱数据中提取关键特征, 还能减少网络模型的复杂度。 因此, 提出基于CNN和NIR的酒醅酸度定量分析方法, 以某酒企生产线中采集的545个酒醅样本光谱数据作为研究对象, 采用标准正态变换(SNV)、 Savitzky-Golay (SG)滤波和一阶求导(1stD)三种算法相结合对原始光谱进行预处理; 利用无信息变量消除法(UVE)选择光谱数据的特征波长; 使用CNN建立酒醅酸度模型。 结果表明: (1)对光谱数据进行预处理后, 消除了原始光谱中的基线偏移, 噪声等问题; 经过预处理后的光谱数据模型相较于原始光谱建模, 预测集决定系数提升了22.85%, 预测集均方根误差降低了0.049 5, 提高了酒醅酸度与光谱反射率的相关性。 (2)对光谱数据进行波长筛选后所建立的模型相较于全波段建模, 预测集决定系数提升了2.04%, 预测集均方根误差降低了0.004 8。 (3)基于CNN建立的酸度预测模型, 预测集决定系数为0.955 5, 预测集均方根误差为0.039 1。 相较于偏最小二乘回归模型, 预测集决定系数提升了1.03%, 预测集均方根误差降低了0.097 6; 相较于反向传播神经网络模型, 预测集决定系数提升了1.16%, 预测集均方根误差降低了0.099 4。 该方法可实现对酒醅酸度的快速、 准确测量, 为后续酒醅酸度在线检测提供方法支撑。
Rapid and accurate detection of the acidity of fermented grains can significantly improve the yield of Baijiu and the quality of finished liquor. Near infrared spectroscopy (NIR) mainly contains information on octave and ensemble frequencies of molecules, i. e., the vibrations of hydrogen-containing groups (C—H, N—H, O—H) in organic matter. It is usually used for qualitative and quantitative analysis of hydrogen-containing compounds in samples. The NIR can be used to determine the acidity of fermented grains in a simple, rapid overcoming the shortcomings of traditional chemical analysis methods, such as long detection cycles, large reagent consumption, and human errors. As NIR is an indirect analysis technology, establishing a calibration model is the key to accurately detecting the acidity of fermented grains. As a typical model in deep learning, convolutional neural networks (CNN) have the advantages of local area connection and weight sharing. It can not only extract critical features from complex spectral data, but also reduce the complexity of network models. Therefore, a quantitative analysis method for the acidity of fermented grains based on CNN and NIR is proposed in this work. The research object is the spectral data of 545 fermented grains samples collected in the production line of a wine enterprise, and the original spectra are preprocessed using a combination of three algorithms: standard normal variation (SNV), Savitzky-Golay(SG) filtering and first derivative (1stD); uninformative variable elimination (UVE) is used to select the characteristic wavelength of spectral data; CNN is used to establish the acidity model of fermented grains. The results show that: (1) The pre-processed spectral data eliminated the baseline shift and noise problems in the original spectra, increased the prediction set coefficient of determination by 22.85%, and decreased the root mean square error by 0.049 5 compared with the original spectral modeling, which improved the correlation between the acidity of fermented grains and spectral reflectance. (2) The model established after wavelength screening of spectral data increased the determination coefficient of the prediction set by 2.04% and decreased the root mean square error of the prediction set by 0.004 8 compared with full-wavelength modeling. (3) The acidity prediction model based on CNN had a determination coefficient of 0.955 5 and a root mean square error of 0.039 1. Compared with the partial least squares model, the determination coefficient of the prediction set is increased by 1.03%, and the root mean square error of the prediction set is reduced by 0.097 6. Compared with the backpropagation neural network model, the determination coefficient of the prediction set is increased by 1.16%, and the root mean square error of the prediction set is reduced by 0.099 4. The research results can realize the rapid and accurate measurement of the acidity content of fermented grains and provide method support for subsequent online detection of the acidity of fermented grains.
酒醅主要由稻谷壳、 小麦、 高梁等含氢化合物组成, 是酿造白酒的主要原材料[1]。 酸度作为酒醅主要检测成分之一, 含量在酒醅中约占0.2%, 对出酒率和成品酒的口感、 质量、 风味方面起着重要作用[2, 3, 4]。 如何对酒醅的酸度进行快速、 准确的检测, 对于提高出酒率、 提升成品酒品质有着重要意义[5]。 传统的酒醅酸度化学分析方法存在时间长、 人为误差、 试剂消耗大、 不能及时指导生产等缺点, 无法满足白酒生产过程中快速、 准确检测的需求[6]。 有机物以及部分无机物分子中含氢基团X— H(X=C、 N、 O、 S)跃迁的倍频和合频吸收形成了近红外光谱(near infrared spectroscopy, NIR)中的吸收谱线, 通过测量物质所吸收的近红外光能量的大小, 可对含量为0.1%以上的有机物成分进行定量、 定性分析[7, 8]。 高畅[9]等利用偏最小二乘回归(partial least squares regression, PLSR)对白酒基酒中的总酯光谱数据建立了定量分析模型, 模型的决定系数为0.937。 韩四海[10]等以100个白酒基酒样品光谱数据作为实验材料, 将偏最小二乘法应用于白酒基酒乙醇含量预测, 结果显示预测模型的决定系数为0.954 8。 陈斌[11]利用离散余弦变换结合反向传播神经网络(back propagation neural network, BPNN)对市面上常见的几种品牌白酒的酒精光谱数据建立了预测模型, 结果显示预测模型的决定系数为0.974 4。
以上研究表明, 由于NIR技术快速、 准确检测等特性, 已有研究人员将其结合传统建模法在白酒基酒、 酒醅成分检测领域进行探索。 但是随着研究的深入, 传统预测模型的缺点也在逐渐显现, 如PLSR并不能有效的处理非线性的光谱数据; BPNN在训练过程中易陷入过拟合, 导致预测效果不佳, 不能满足酒醅酸度检测需求。
卷积神经网络(convolutional neural networks, CNN)是一种前向传播(forward propagation, FP)类型的人工神经网络, 是深度学习(deep learning, DL)的代表算法之一[12, 13]。 与传统神经网络结构不同, CNN由卷积层、 池化层和全连接层组成, 卷积层与前一层的连接采用权值共享和局部连接的方式, 这样特殊的连接方法在实现每个卷积层包含多个特征映射的同时有效的降低了网络的参数个数, 既减少了数据量又能保留有用信息, 缓解了模型的过度拟合, 在处理非线性问题方面有较为广泛的应用[14]。 综上所述, 选择将CNN结合NIR应用于酒醅酸度的定量分析。
采集生产过程中的酒醅样本的NIR数据作为研究对象。 采用标准正态变换(standard normal variation, SNV)、 Savitzky-Golay(SG)滤波和一阶求导(1st Derivative, 1stD)三种算法相结合对原始光谱进行预处理, 消除原始光谱中的基线偏移, 提升光谱数据的信噪比; 利用无信息变量消除法(uninformative variable elimination, UVE)选择特征波长, 降低光谱数据维度, 提高模型预测准确率; 使用CNN建立酒醅酸度预测模型, 对测试集数据进行预测。
样品取样位置为生产线酒醅料斗中部及中下部; 两处各取50 g, 混合均匀后装入密封袋中; 样品数量为545个, 按照7∶ 3的比例随机划分为训练集和测试集。 样品的酒醅酸度在24 ℃室温环境下按照DB34/T2264— 2014《固态发酵酒醅分析方法》化学分析方法测得, 如表1所示。
| 表1 酒醅样本酸度统计结果 Table 1 Statistical results of acidity content of fermented grains samples |
样品的NIR数据通过威斯派克傅里叶变换近红外光谱仪Q2000扫描获取, 具体参数如表2所示。 光谱采集方式为漫反射方式, 光谱采集前对酒醅样品进行压片操作, 以减少颗粒不均带来的影响。 在采集到数据后, 利用SNV、 SG和1stD这三种预处理算法相结合, 对光谱数据预处理。
| 表2 设备参数 Table 2 Equipment parameters |
采用连续投影算法(successive projections algorithm, SPA)、 主成分分析法(principal component analysis, PCA)和UVE对酒醅光谱数据进行特征波长选择。
SPA算法是利用向量的投影分析, 将波长依次投射到其他波长上, 比较投影向量的大小, 将最大的投影向量作为待选波长, 然后基于校正模型的效果选择最终的特征波长[15]。 PCA算法将多项指标转化为几项综合性指标, 从一组特征出发, 根据重要性大小, 计算出一组从大到小排列的新特征, 它们是原特征的线性组合, 由新特征计算出原特征的映射值, 即为处理后的样本数据[16]。 UVE算法是利用噪声的无关变量信息去选择光谱自身的特征变量, 首先通过光谱变量结合噪声的自变量矩阵对目标矩阵的回归系数进行变量判断, 然后借助噪声域的上下限剔除界限内的光谱变量, 最终确定特征光谱波长[17]。
CNN结构主要分为3层: 卷积层、 池化层和全连接层[18], 如图1所示。 卷积层由多个特征映射组成, 每个特征映射又包含多个神经元, 单个卷积层只能提取到部分简单的特征, 采用多层卷积组合可从低级特征中迭代的提取出复杂和高级的特征; 池化层一般由2× 2的过滤器构成, 它利用局部相关性进行亚采样, 逐步减小空间大小, 减少网络中的参数量和计算量的同时保留有用信息, 从而控制过拟合; 全连接层由若干个神经元构成, 完全连接前一层中的所有神经元, 将经过卷积层和池化层提取的局部特征组合, 传递给输出层[19]。
选择酒醅酸度预测值和实际值的决定系数(coefficient of determination, R2)、 交叉验证均方根误差(root mean square error of cross validation, RMSECV)和预测均方根误差(root mean square error of prediction, RMSEP)作为模型预测精度评价指标。 R2用于评价样本集预测值与实测值之间的线性相关程度, 其值越接近于1, 则表明预测的效果越好; RMSECV主要评价建模算法可行性及预测能力, 在模型校正过程中采用交叉验证的方法来计算误差值; RMSEP主要用于评价所建模型对外部样本的预测能力, RMSEP越小, 表明模型对外部样本的预测能力越强[20]。
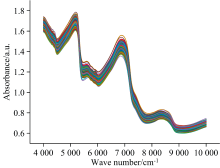
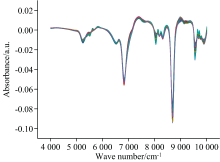
近红外光谱仪测得的酒醅光谱数据如图2所示。 由于原始光谱中不仅包含有用信息, 还可能存在噪声和基线漂移等问题, 为消除这些干扰, 利用SNV、 SG和1stD算法对光谱图像进行预处理, 结果如图3所示。 为研究经过预处理后的光谱数据对模型预测准确率的影响, 将其建立CNN模型, 结果如表3所示。
| 表3 预处理算法用于CNN模型的结果 Table 3 The results of combined pretreatment in CNN model |
由图3可知, SNV、 SG和1stD预处理算法组合在保留有用信息的同时, 消除了基线漂移, 提高了光谱信噪比; 由表3可知, 经过预处理后, 预测集R2提升了22.85%, RMSEP降低了0.049 5, 优于原始光谱建模。
利用SPA、 PCA和UVE算法筛选出光谱特征波长组合, 然后分别建立CNN预测模型, 预测结果如表4所示。 由表4可知, 波长选择算法可以有效的减少变量个数, 提升建模效率, 优化模型预测效果; 通过对比三种算法, UVE在预测集R2和RMSEP方面均优于其余两种算法, 故选择UVE作为酒醅光谱特征波长选择算法。
| 表4 不同波长选择算法在CNN模型中的结果 Table 4 Results of different wavelength selection algorithms in CNN model |
由于CNN模型特殊的网络结构, 需要将输入该模型的光谱数据向量转化为二维光谱信息矩阵, 具体操作如下:
设X为某一酒醅样本的光谱数据, 且为行向量形式, 则该样本的二维光谱矩阵为
该矩阵在反映数据方差和协方差相对大小的同时包含原始光谱的关键信息, 符合统计学要求; 在光谱分析方面, 将光谱向量转化为光谱矩阵, 虽然增加了数据的维度, 但可以在一定程度上反映出光谱数据的起伏, 使CNN能够全面学习光谱数据的内在特征, 提高预测模型的准确率[21]。
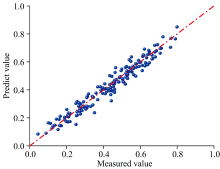
借助Keras框架建立CNN模型基本结构, 实验过程中不断调整神经网络层数和神经元个数, 最终确定神经网络的参数, 如表5所示。 为避免梯度饱和、 模型欠拟合等问题, 选择Relu作为激活函数, 选择Adam作为模型优化器, 学习率由默认的0.001调整为0.000 01, 迭代次数为500。 利用CNN模型预测酒醅酸度, 结果如图4所示, 预测集R2为0.955 5, RMSEP为0.039 1, 预测集样本点密集的分布于拟合直线两侧。
| 表5 CNN模型参数设置 Table 5 The parameter of CNN model |
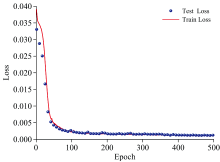
CNN训练过程中的损失函数曲线如图5所示。 在训练初期, 随着迭代次数的增加, 损失率逐渐降低, 说明学习率是适合的, 符合梯度递减的过程; 经过一定阶段的学习, 损失函数曲线趋于平滑, 无过拟合现象出现。
将目前光谱分析研究中常用的BPNN、 PLSR模型分别建立酒醅酸度预测模型。 BPNN的隐含层为3层, 神经元的个数依次为64、 32、 16, 激活函数均为Relu; PLSR回归因子设置为35。 对预测结果进行汇总对比, 见表6。
| 表6 PLSR模型、 BPNN模型和CNN模型预测效果比较 Table 6 Comparison of prediction effects of PLSR model, BPNN model and CNN model |
以生产线上的酒醅样品作为研究对象, 采集NIR以及酸度数据。 利用SNV、 SG和1stD预处理算法组合对原始光谱进行预处理; 采用UVE算法提取光谱特征波段; 使用BPNN、 PLSR和CNN分别建立酒醅酸度预测模型, 对比后得到酒醅酸度的最优模型, 结果表明:
(1) 利用SNV、 SG和1stD预处理算法组合, 减少了噪声、 消除了酒醅原始光谱中的基线漂移; 经过预处理后, 预测集R2提升了22.85%, RMSEP降低了0.049 5, 提高了酒醅酸度与光谱反射率的相关性。
(2) 波段选择算法在一定程度上降低了数据维度, 减少了后续模型所需的计算时间, 提高了模型预测准确率。 基于UVE算法提取出的特征波段数据所建立的CNN模型, 相较于SPA算法, 预测集R2提升了1.33%, RMSEP降低了0.101 5; 相较于PCA算法, 预测集R2提升了1.41%, RMSEP降低了0.102 4。
(3) CNN模型的预测集R2为0.955 5, RMSEP为0.039 1, 相比于传统模型PLSR、 BPNN, 预测集R2分别提升了1.03%、 1.16%; RMSEP分别下降了0.097 6、 0.099 4。
将CNN结合NIR可实现对酒醅酸度快速、 准确检测, 为后续酒醅酸度在线检测提供方法支撑。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|