作者简介: 蔡 羽,女, 1987年生,湖北中医药大学药学院讲师 e-mail: yucai2018@hbtcm.edu.cn
山药为薯蓣科植物薯蓣的根茎, 其中的多糖、 多酚、 皂苷、 黏蛋白和维生素C等成分使山药具有抗肿瘤、 抗氧化、 抗炎症、 降血糖和降血脂等作用。 不同产地的山药由于生长条件存在差异, 致使药用成分含量显著不同, 结合独特的炮制工艺, 进而导致市场价格差别大, 所以山药饮片的产地识别至关重要。 为对山药饮片进行产地溯源, 本文基于激光诱导击穿光谱(LIBS)技术提出多元散射矫正-改进遗传算法-支持向量机(MSC-IGA-SVM)模型对山药产地进行精确识别。 使用八个不同产地的山药饮片进行LIBS实验, 八种产地的山药饮片磨粉过筛后制成粉末压片, 通过采集山药饮片的LIBS光谱, 分别使用单一分类器与使用光谱预处理、 特征提取及模式识别算法的模型对光谱的识别结果进行对比。 将光谱信号按2:1的比例划分为训练集和测试集, 使用5次交叉验证K-邻近算法(KNN)模型的测试集准确率作为预处理参数优化的评价指标。 各类药材的平均光谱整体趋势一致, 所含谱峰基本相同, 但因产地不同导致峰值强度各不相同, 道地山药对一些金属元素(K, Na, Ca, Mg, Al)的富集能力大于非道地产区山药, 其中, K元素特征谱线(769.90 nm)的峰值最高, 即山药饮片中K元素含量最多, 相关研究表明山药根茎对K元素的富集能力最强。 选取35条关键谱线进行分析, 在识别种类多、 识别难度大的情况下, 改进遗传算法(IGA)比主成分分析(PCA)更能清楚辨别光谱中的非线性关系, 同时受噪声的影响更小。 MSC-IGA-SVM模型的产地溯源效果最好。 MSC-IGA-SVM模型的交叉验证集准确率为96.9%, 测试集的准确率为97.32%, 与直接使用原信号建立的最好模型支持向量机(SVM)(96.43%)相比, 测试集准确率提高了0.87%。 同时, MSC-IGA-SVM模型将输入变量的维度减少了99.93%。 结果表明, LIBS技术结合MSC-IGA-SVM模型能够快速、 准确对山药饮片进行产地溯源。
Rhizoma Dioscoreae contains polysaccharides, polyphenols, saponins, mucins and vitamin C, which have anti-tumor, anti-oxidant, anti-inflammatory, hypoglycemic, and hypolipidemic effects. Due to the differences in growth conditions of Rhizoma Dioscoreae from different origins, resulting in significantly different contents of medicinal ingredients, combined with unique processing technology, which in turn lead to large differences in market prices, it is crucial to identify the origin of Rhizoma Dioscoreae Tablets. In order to trace the origin of Rhizoma Dioscoreae Tablets, this paper proposed a Multiplicative signal correction-improved genetic algorithm-support vector machine (MSC-IGA-SVM) model based on Laser-induced breakdown spectroscopy (LIBS) technique for accurate identification of Rhizoma Dioscoreae origin. In the paper, LIBS experiments were conducted using eight Rhizoma Dioscoreae Tablets of different origins. The Rhizoma Dioscoreae Tablets of eight origins were ground and sieved to make powder pressed tablets. The recognition results of the spectra were compared by collecting LIBS spectra of Rhizoma Dioscoreae Tablets using a single classifier and a model using spectral preprocessing, feature extraction and pattern recognition algorithms, respectively. In the research, the spectral signals were divided into training and test sets in the ratio of 2:1, and the test set accuracy of the K-Nearest Neighbor (KNN) model using five cross-validations was used as an evaluation index for the optimization of preprocessing parameters. The overall trend of the average spectra of all herbs was consistent. The contained spectral peaks were the same, but the peak intensities varied due to different origins, and the enrichment ability of some metal elements (K, Na, Ca, Mg, Al) was greater for Rhizoma Dioscoreae growed in the Dao-di Areas than for those not growed in the Dao-di Areas, among which, the peak of the characteristic spectral line of element K (769.90 nm) was the highest, i. e., the Rhizoma Dioscoreae Tablets contained the most element K. Related studies showed that the root of Rhizoma Dioscoreae has the strongest enrichment capacity for element K. Thirty-five key spectral lines were selected for analysis. Improved Genetic Algorithm (IGA) could discriminate the nonlinear relationships in the spectra more clearly than Principal Component Analysis (PCA) in the case of many identification species and difficult identification while being less affected by noise. The MSC-IGA-SVM model had the best origin traceability. The accuracy of the MSC-IGA-SVM model was 96.9% for the cross-validation set, and the accuracy of the test set was 97.32%, which was 0.87% higher than the best model Support Vector Machine (SVM) built directly using the original signal (96.43%) for the test set. Meanwhile, the MSC-IGA-SVM model reduced the dimensionality of the input variables by 99.93%. The results showed that the origin of Rhizoma Dioscoreae Tablets could be traced by the LIBS technique combined with the MSC-IGA-SVM model quickly and accurately.
山药为薯蓣科植物薯蓣的根茎, 其中的多糖、 多酚、 皂苷、 黏蛋白和维生素C等成分使山药具有抗肿瘤、 抗氧化、 抗炎症、 降血糖和降血脂等作用[1]。 不同产地的山药由于生长条件存在差异, 致使药用成分含量显著不同[2, 3, 4], 结合独特的炮制工艺, 进而导致市场价格差别大。 然而, 不同产地的山药外形相似, 难以通过肉眼观测识别。 商家若有意或无意将不同产地的山药饮片混淆, 会导致药用成分产生差异, 致使药材低效或无效, 严重时会与其他药材发生反应甚至产生毒副作用[5]。 常用的药材产地鉴别技术主要有色谱技术[6, 7]、 电泳方法[8, 9]以及同位素质谱分析[10]等, 但上述方法存在预处理繁琐、 耗时长、 费用昂贵、 设备维护复杂及无法原位在线分析等问题。 因此, 寻找一种预处理简单、 快速、 准确的药材产地溯源检测技术具有重要意义。
激光诱导击穿光谱(laser-induced breakdown spectroscopy, LIBS)作为一种新兴的基于原子发射光谱的元素分析技术, 凭借样品前处理简单、 微损甚至无损、 在线原位检测等优点, 已被广泛应用于地质勘探[11, 12]、 工业监测[13, 14]、 医学检测[15]等方面, 是一种极具应用前景的成分分析技术。 近年来, LIBS在中药成分检测领域逐渐兴起。 2015年, Liu等运用LIBS信号强度变化率结合移动窗标准差法快速评价朱砂和雄黄混合过程, 结果证明了LIBS在监测中药制药生产方面的潜力[16]。 2017年, Daniel等采用LIBS技术测定了18种波兰草药中钙、 钾和镁等其他金属元素(钠、 铜、 铁、 锰、 锌等), 并对其中的钙、 钾和镁进行定量分析, 结果与ICP-OES一致[17]。 2018年, ChanghwanEum等将近红外光谱与LIBS光谱相结合来区分国产和进口黄芪, 使用近红外光谱的SVR系数与35个LIBS谱峰值进行识别准确率达95.8%, 与单独使用近红外光谱(91.5%)相比, 准确率提高了4.3%[18]。
以上研究证明了LIBS技术在中药成分检测中的可行性[19, 20, 21]。 然而, 基于LIBS技术的中药产地溯源研究较少, 仍处于起步阶段, 目前的研究都存在药材产地数量少, 识别准确率较低等问题[22, 23]。 因此, 基于LIBS技术结合人工智能算法对不同产地的山药进行系统性研究。
我们搭建LIBS试验系统并结合多元散射矫正-改进遗传算法-支持向量机(multiplicative signal correction-improved genetic algorithm-support vector machine, MSC-IGA-SVM)模型来识别8种不同产地的山药饮片。 八种产地的山药饮片磨粉过筛后制成粉末压片, 通过采集山药饮片的LIBS光谱, 分别使用单一分类器与光谱预处理、 特征提取及模式识别算法的模型对光谱的识别结果进行对比。 结果显示, LIBS技术结合MSC-IGA-SVM模型可以准确且快速识别山药产地。
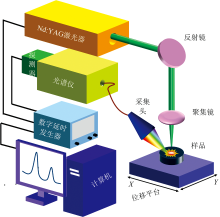
所用的LIBS实验装置如图1所示, 由Nd:YAG激光器、 光谱仪、 探测器等几部分组成。 在数字延时发生器(Stanford Research Systems, DG645)的控制下, 触发信号被输入到由Q开关控制的Nd:YAG激光器(北京镭宝光电, Nimma-400型, 波长: 532 nm; 脉冲宽度8 ns)中产生激光脉冲, 经过反射镜和聚焦镜后聚焦到样品表面, 与样品作用产生等离子体。 使用采集头采集等离子体的发射光信号, 将其耦合进光纤并传输到六通道光纤光谱仪(Avantes, AvaSpec-ULS4096CL-EVO, 光谱范围: 196~874 nm, 最小门宽: 9 μ s)。 光谱仪分光后的信号由数字延时发生器控制的探测器(日本滨松, Complementary metal-oxide-semiconductor, CMOS)进行光电转换, 在计算机中生成LIBS光谱。 本实验中, 经过参数优化激光能量、 重复频率、 延时和门宽分别设置为35 mJ, 10 Hz, 1 μ s和9 μ s。 5个脉冲的光谱均值作为一幅光谱, 每个样本采集80幅光谱, 共得到1 920幅光谱。
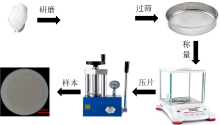
所用八类不同产地的山药饮片由湖北中医药大学鉴定并提供, 如表1所示, 由于山药饮片产地、 炮制工艺以及价格各不相同, 导致它们药用价值存在差异。 实验中样本的处理流程如图2所示。 首先, 使用研钵将饮片研磨成粉过100目筛。 然后, 使用电子天平称量9 g硼酸粉末和1 g样品粉末, 在压样机30 t压力下将待测样品压制成直径为40 mm的圆饼状压片。 每种产地的山药饮片使用3个重复样本, 共制备24个压片。
| 表1 实验所用药材饮片 Table 1 Herbal tablets used for experiments |
多元散射校正(multiplicative signal correction, MSC)、 Savitzky-Golay(SG)卷积平滑法和小波变换(wavelet transform, WT)是常用的光谱数据预处理方法。 MSC校正谱线漂移和基线平移, 能使同类光谱数据向平均光谱靠拢, 提高数据的稳定性。 SG卷积平滑[24]和WT这两种预处理方法注重于去除光谱噪声, 使谱线更加平滑。
主成分分析(principal component analysis, PCA)和遗传算法(genetic algorithm, GA)是常用的特征提取算法。 PCA特征提取主要是在线性相关假设下, 消除变量之间可能存在的多重共线性。 GA是在模拟自然进化的过程中逐步淘汰对选择过程影响较小的特征, 其使用概率机制迭代, 所选个体随机, 容易陷入局部最优解的状况[25]。 将GA改进为IGA(improved genetic algorithm, IGA)。 IGA是记录在300个GA迭代过程最优个体的特征, 并选择累积次数超过150次的特征作为最终的特征, 其能够有效解决局部最优解的问题。
本文使用几种常用的模式识别算法, 包括有K最近邻(K-nearest neighbor, KNN)、 支持向量机(support vector machine, SVM)和集成学习算法(ensemble machine learning, EML)。 KNN使用目标最邻近的K个样本标签值来表示目标的标签[26]。 SVM目的是寻找一个最大化间隔的超平面, 使得两类数据被超平面分开, 同时两类中最接近超平面的点到超平面的距离最大[27]。 EML的原理是将多个弱分类器相结合进行分类识别[28]。
将光谱信号按2:1的比例划分为训练集和测试集, 使用5次交叉验证KNN模型的测试集准确率作为预处理参数优化的评价指标。
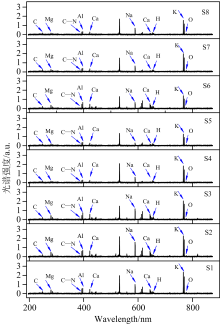
各类药材饮片(S1— S8)的平均光谱及典型峰值如图3所示。 各类药材的平均光谱整体趋势一致, 所含谱峰基本相同, 但峰强度各不相同, 分析认为不同产地的山药富集能力存在差异, 道地山药对一些金属元素的富集能力大于非道地产区山药。 图中, 谱峰代表的金属元素有K, Na, Ca, Mg, Al等, 非金属元素有C, H, O及C— N键等。 其中, K元素特征谱线(769.90 nm)的峰值最强, 即山药饮片中K元素含量最多, 相关研究表明山药根茎对K元素的富集能力最强[29]。
为剔除光谱中大量的冗余信息, 观察图3并选择峰值较为明显的35条特征谱线作为特征提取的预选谱线, 如表2所示。
| 表2 所选特征谱线 Table 2 Selected characteristic spectral lines |
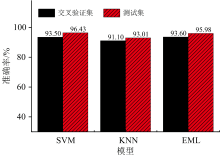
对八种产地山药饮片24565维的原始光谱进行产地溯源, 使用SVM, KNN和EML三种模式识别方法的分类结果如图4所示。 图中显示, 三种模型的交叉验证集和测试集准确率均在90%以上, SVM模型的分类效果最好, 交叉验证集准确率为93.50%, 测试集准确率为96.43%。 由于山药饮片产地种类多、 光谱维度高, 原始信号数据量大且包含噪声和冗余特征, 致使分类耗时长且分类准确率低, 因此有必要对原始光谱进行数据预处理和特征提取。
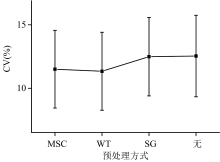
首先, 使用三种预处理方法对八种产地山药饮片的原始光谱进行处理。 在SG卷积平滑法中, 选用5, 7, 9和11的窗口宽度进行参数优化, 通过比较识别准确率, 确定本研究中所用窗口宽度为5。 在WT预处理过程中, 小波函数采用常用的db2, db4, db8, sym2, sym5和sym8, 分解层数采用1~9层, 最终确定选用db2函数2层小波分解为WT的最优参数。 经过预处理后, 各类光谱的CV均值和标准差如图5所示。 图5显示, 无预处理时光谱的波动性最大, 各类光谱的CV均值为12.54%, 经过三种预处理后CV均值明显下降, 光谱的稳定性明显提升。 MSC和WT的CV均值较为接近且小于SG卷积平滑法的CV均值, 表明MSC和WT对光谱稳定性的提升效果好于SG卷积平滑法。 WT的CV均值小于MSC, 但WT各类光谱CV的标准差较大, 即各类光谱的波动差距较大, 而MSC各类光谱的波动差距较小, MSC在减小光谱波动性方面整体表现较好。
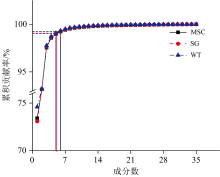
对预处理后的光谱数据进行特征提取。 按表2选出35条特征谱线后, 分别使用PCA和IGA进行特征提取。 不同光谱预处理下PCA选择的主成分数量与累积贡献率的关系如图6所示。 当累积贡献率达到99%以上时, MSC、 SG卷积平滑、 WT三种预处理方式下各选择6, 5和5个主成分, 其累积贡献率分别为99.23%(黑线)、 99.03%(红线)、 99.07%(蓝线)。 针对不同预处理和模式识别方法处理的光谱, 使用IGA提取的特征数量如表3所示。
| 表3 在各方法下IGA提取的特征数量 Table 3 Number of features extracted by IGA under each method |
使用SVM, KNN及EML三种模式识别方法对预处理和特征提取后的信号进行分类, 各模型分类结果排序后如表4所示。
| 表4 各模型分类结果排序 Table 4 Ranking of classification results by model |
表4显示, 排名前十的模型中接近一半的模型使用了MSC预处理方法, 接近一半的模型使用了SVM模式识别方法。 结果表明, MSC预处理方法和SVM模式识别方法有助于准确识别山药饮片产地。 测试集准确率排名前五的模型均使用了IGA进行特征提取, 在识别种类多、 识别难度大的情况下, IGA比PCA更能清楚辨别光谱中的非线性关系, 同时受噪声的影响更小。 MSC-IGA-SVM模型的产地溯源效果最好。 MSC-IGA-SVM模型的交叉验证集准确率为96.9%, 测试集的准确率为97.32%, 与直接使用原信号建立的最好模型SVM(96.43%)相比, 测试集准确率提高了0.87%。 同时, MSC-IGA-SVM模型将输入变量的维度减少了99.93%。
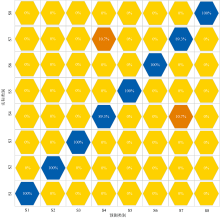
MSC-IGA-SVM模型对八种山药饮片产地溯源的混淆矩阵如图7所示, 除了S4和S7有错误分类现象外, 其他类别均能正确识别。 S1和S2虽然产地相同, 但两批山药饮片炮制工艺不同, 价格相差较远, 因此品质存在差异, 能进行精确分类。 而对于S4和S7, 两种产地山药产地虽然不同, 但存在错分现象。 10.7%的S4错分为S7, 同时10.7%的S7错分为S10。
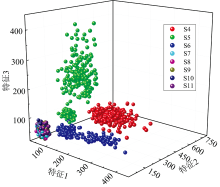
为进一步探究错误分类原因, 对IGA选取的特征进行三维成像, 如图8所示, S4与S7存在重叠区域, 其数据存在相似性。 S4为广西南宁无硫高温烘焙的价值为31元/500 g的山药饮片, S7为安徽池州无硫低温烘焙的价值为18.5元/500 g的山药饮片。 两者产地相差较远, 且炮制工艺不同, 难以辨别的原因推测是山药种植品种、 种植条件等方面存在共性。
针对山药饮片的产地溯源问题, 使用LIBS技术结合MSC-IGA-SVM模型对产地识别的准确度进行了改善。 使用KNN、 SVM和EML分类器对八类光谱直接使用原信号进行分类, SVM模型具有较强鲁棒性, 效果最好, 其准确率为96.43%。 使用预处理(MSC、 SG卷积平滑、 WT)、 特征提取(PCA和IGA)的方法对模型改进, MSC-IGA-SVM模型的识别效果最好, 其能有效降低光谱波动性, 同时输入变量维度降低了99.93%, 将测试集准确率提升为97.30%。 结果表明, LIBS技术结合MSC-IGA-SVM模型能够准确且快速对山药饮片进行产地溯源。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|