作者简介: 黄 清, 1998年生, 内蒙古农业大学计算机与信息工程学院硕士研究生 e-mail: 1905550194@qq.com
脂肪作为牛奶中的重要营养成分, 是评价牛奶质量的一项重要指标。 高光谱图像技术能够提供几十到数千波长的数据, 能够反映牛奶中不同组成成分细微的光谱差异; 另一方面, 相邻波段之间往往具有很强的相关性, 不仅增加了计算量, 而且容易造成维数灾难等问题, 因此对高光谱数据进行波段选择非常重要。 工作中提出了PLS-ACO特征波段选择方法, 并与遗传算法结合, 组合成了PLS-ACO-GA的特征波段选择新方法。 提出的两种方法以蚁群算法为基础, PLS回归模型回归系数的绝对值作为评价波长重要性的主要依据, 以此作为蚁群算法的启发式信息, 利用蚁群算法进行智能搜索, 结合遗传算法, 产生更多优秀的特征波段组合, 避免PLS-ACO算法得到的只是局部最优解, 得到的最优波段组合能够更好的反映牛奶中脂肪成分的信息; 通过计算波长贡献率, 筛选出最优波段组合, 并与遗传算法, CARS算法和基本蚁群算法光谱特征选择方法比较, 最后比较不同特征选择方法下的PLS回归模型预测效果。 PLS-ACO, PLS-ACO-GA, CARS, GA和ACO分别筛选了牛奶样品光谱中的18, 16, 40, 43和42个特征波段。 其中PLS-ACO-GA筛选波段后的PLS预测模型效果最好, 预测集R2P和RMSEP分别为0.997 6和0.062 2, PLS-ACO次之, 预测集R2P和RMSEP分别为0.997 0和0.077 8。 PLS-ACO和PLS-ACO-GA不仅减少了特征波段数量, 而且提高了模型的精度。 对PLS-ACO-GA进行特征波段选择后的数据, 建立MLR, RFR和PLS回归预测模型。 MLR预测模型的R2P和RMSEP分别为0.997 6和0.062 3。 RFR回归模型R2P和RMSEP分别为0.999 9和0.003 0, PLS回归模型的R2P和RMSEP分别为0.997 6和0.062 2。 RFR模型在三种回归预测模型中表现最好。 研究结果表明PLS-ACO和PLS-ACO-GA这两种方法可以实现光谱数据特征波段选择, 高光谱技术可以实现牛奶中脂肪含量的检测, 为牛奶脂肪含量检测提供了一种新的、 快速无损的方法。
As an important nutritional component in milk, fat is an important index to evaluate milk quality. Hyperspectral image technology can provide tens to thousands of bands of data and can reflect the subtle spectral differences of different components in milk. On the other hand, there is often a strong correlation between adjacent bands, which increases the amount of calculation and easily causes problems such as dimension disaster. Therefore, it is very important to select bands for hyperspectral data. This paper proposes a PLS-ACO feature band selection method combined with a genetic algorithm to form a new feature band selection method of PLS-ACO-GA. The two methods proposed in this paper are based on ant colony optimization. The absolute value of the regression coefficient of the PLS regression model is the main basis for evaluating the importance of wavelength, which is used as the heuristic information of ant colony optimization. Ant colony optimization is used for intelligent search, combined with genetic algorithm to produce more excellent characteristic band combinations. To avoid that pls-aco algorithm only obtains the optimal local solution. The optimal band combination can better reflect the information of fat composition in milk. By calculating the wavelength contribution rate, the optimal band combination is selected and compared with the spectral feature selection methods of genetic algorithm, cars algorithm and basic ant colony optimization. Finally, the prediction effects of the PLS regression model under different feature selection methods are compared. PLS-ACO, PLS-ACO-GA, CARS, GA and ACO screened 18, 16, 40, 43 and 42 characteristic bands in the spectrum of milk samples, respectively. The PLS prediction model after the PLS-GA-ACO screening band has the best effect. The prediction sets R2P and RMSEP are 0.997 6 and 0.062 2 respectively, followed by PLS-ACO, and the prediction sets R2P and RMSEP are 0.997 0 and 0.077 8 respectively. PLS-ACO and PLS-ACO-GA reduce the number of characteristic bands and improve the accuracy of the model. MLR, RFR and PLS regression prediction models are established based on PLS-ACO-GA data after characteristic band selection. The R2P and RMSEP of the MLR prediction model are 0.997 6 and 0.062 3 respectively. R2P and RMSEP of the RFR regression model were 0.999 9 and 0.003 0 respectively, and R2P and RMSEP of the PLS regression model were 0.997 6 and 0.062 2 respectively. RFR model performs best among the three regression prediction models. The results show that hyperspectral technology can detect the fat content in milk, which provides a new, rapid and non-destructive method for the detection of fat content in milk.
随着日常生活水平的提高, 牛奶已经逐渐成为了人们生活中必不可少的一种营养品, 牛奶中含有丰富的乳脂肪, 蛋白质等营养物质, 其中, 乳脂肪是特别优质的一种脂肪, 其组成成分是一分子的甘油和三分子的脂肪酸。 全脂牛奶的脂肪含量约为3%, 低脂牛奶的脂肪含量为0.5%~2%, 脱脂牛奶中脂肪含量低于0.5%。 不同人群对牛奶脂肪含量有着不同的需求, 儿童、 青少年因成长需要可以选择全脂牛奶。 而一些患有高血脂、 高血压等疾病的人群, 应当选择脱脂牛奶。 牛奶中对脂肪含量有着及其严格的把控标准, 对牛奶脂肪含量的精准检测显得愈发重要。 随着光谱技术的发展, 光谱分析法凭借着其快速, 无损等优点, 逐渐应用到牛奶品质检测中来[1]。 高光谱图像具有极高的光谱分辨率, 高光谱数据能够反映牛奶中不同组成成分的光谱信息, 然而高光谱图像的众多波段之间, 尤其是相邻或相近波段存在严重的信息冗余。 如何提取高光谱数据的有效信息, 去除冗余信息是决定高光谱数据应用效果的关键要素之一[2]。 波段选择是进行光谱数据分析时常用的一种技术手段, 与其他方法相比, 波段选择只是从全光谱波段中选取与目标信息相关的波段, 并不改变光谱的物理信息。 偏最小二乘(partialleast squares, PLS)是目前应用最广泛的线性建模方法, Li等提出了竞争性自适应重加权算法(competitive adaptive reweighted sampling, CARS), 以PLS回归系数绝对值大小作为衡量指标, 采用竞争性自适应加权采样法对光谱数据进行变量筛选[3], 但是CARS算法随机性强, 稳定性差, 需要经过运行多次才可能选出相对较好的特征波段组合。 有研究提出将变量投影重要性系数(variable importance in the projection, VIP)加PLS回归系数结合蚁群算法(ant colony optimization, ACO)的波长筛选新方法。 张小鸣等提出了一种基于变量有效性精英蚁群系统, 将PLS回归模型系数作为蚁群的初始信息素, 在信息素更新过程中引入VIP系数[4]。 在张小鸣发明的方法中, 均以蚁群算法为基础, 而在蚁群算法中, 以正反馈机制为基础, 这两种方法都容易陷入局部最优, 且计算VIP系数增加了算法的复杂度, 针对这些问题, 本工作将蚁群算法与PLS回归系数相结合(简称为PLS-ACO), 以PLS模型回归系数单独作为蚁群算法的启发式信息, 预测集相关系数(correlation coefficient of prediction,
在天猫、 京东等平台购买了6种不同品牌的牛奶, 其中, 特仑苏脂肪含量为4.4 g· (100 mL)-1, QQ星和蒙牛高钙脂肪含量为3.7 g· (100 mL)-1, The land脂肪含量为3.3 g· (100 mL)-1, 伊利甄浓脂肪含量为4.6 g· (100 mL)-1, 伊利脱脂脂肪含量为0 g· (100 mL)-1。
高光谱采集系统由高光谱成像仪、 高精度扫描云台、 石英卤钨灯照明电源、 等部件组成。 高光谱测量仪为美国Headwall公司2010年生产的型号为1003B— 10141的高光谱成像仪, 光谱范围400~1 000 nm, 共125个波段, 采集到的光谱图像分辨率为777× 1 004像素。
在实验的过程中, 尽量保持牛奶样本与摄像头的距离约20 cm, 为保证不受其他反射光源的影响, 在牛奶样本盛器下垫一块黑色绒布。 在光谱采集过程中, 光谱摄像头中的传感器中会产生暗电流, 每次采集到的光谱数据会伴随着一定的噪声, 影响高光谱图像的质量, 为了避免客观因素影响, 在每次采集光谱图像之前, 需要对该系统进行黑白校正处理, 在一定程度上提高图像的质量[5]。 实验中, 首先盖上相机盖, 点击暗电流, 采集全黑的光谱校正图像IB; 然后取下相机盖, 用白板采集全白校正图像IW; 最后, 采集牛奶样本的光谱图像, 每个样本平行采集三次, 最后根据公式IC=(IR-IB)/(IW-IB)对采集到的牛奶光谱图像进行黑白校正。 其中, IC为校正后的牛奶光谱图像, IR为实验过程中采集到的牛奶光谱图像。 利用ENVI软件从三张图片中选取效果最好的光谱图像, 从中选取40个光线清晰均匀的感兴趣区域, 导出每个区域的光谱反射率数据作为该品牌牛奶的40个样本进行实验。 一共分别采集了6种不同品牌的牛奶样品, 合计240个样本。 图1(a— f)是六个不同品牌牛奶的原始光谱数据反射曲线。 受采集环境和仪器设备的影响, 虽然原始高光谱图像经过了黑白校正处理, 但是数据中仍然存在一些无用的信息和噪声, 通过图1可以看出, 伊利甄浓和QQ星的光谱反射图像在采集的过程中受到的干扰较大。 为了降低散射光和噪声的干扰, 需要对采集到的光谱数据进行数据预处理。
光谱分析中常用的预处理方法包括导数校正法(其中包括一阶导数 (first derivative, 1st Der), 二阶导数(second derivative, 2st Der), 和多阶导数等), 多元散射校正(multiplicative scatter correction, MSC), 标准正态变换(standard normoal variate transformation, SNV), 卷积平滑(savitzky-golay, S-G)等。 其中导数校正法和S-G平滑可以有效消除基线平滑和平移引起的噪声, MSC和SNV则可以减少因光的散射等现象造成的噪声。
使用上述不同的方法对原始光谱数据进行数据预处理, 对预处理后的数据建立PLS回归模型, 将校正集相关系数(correlation coefficient of calibration
| 表1 不同预处理方法的牛奶脂肪含量PLS回归模型预测结果 Table 1 The prediction results of the PLS regression model for the milk fat content of different pretreatment methods |
蚂蚁在寻找食物的过程中, 会根据路径的长短, 判断该路径的好坏, 留下不同浓度的信息, 方便自己的同伴判断觅食的路径, 随着时间的延长, 较优的路径上积累的信息元素比例越来越高, 越来越多的蚂蚁选择此路径, 慢慢的形成了一种正反馈机制。 蚁群算法就是模拟蚂蚁觅食这一行为, 来寻找最优特征波段组合。 在基本蚁群算法中, 初始信息素浓度都是人为设定的(大多数都设定为1), 初始信息素的匮乏, 使得算法收敛时间过长, 执行效率低下[7]。 受到CARS算法的启发, 将PLS回归模型系数的绝对值作为评价波段好坏的标准, 首先在全波段数据下建立PLS回归模型, 每个波段对应的回归系数绝对值作为信息素的初始值。 避免了在第一次迭代中, 蚂蚁随机寻找特征波段的缺点, 加快了算法的收敛速度。 将蚁群算法与PLS回归系数相结合, 虽然解决了传统蚁群算法收敛速度慢, 模型复杂的缺点, 但是仍然无法解决蚁群算法由于正反馈机制容易陷入局部最优的缺点, 因此将PLS-ACO算法与遗传算法相结合, 经过遗传算法迭代产生更多优秀的个体, 为计算波段的贡献率, 提供更多的样本, 以提高算法的稳定性和通用性。 研究提出了全局阈值(threshol)的概念, 传统的遗传算法和蚁群算法, 都是选取某一次迭代最好的个体或几个个体来进入贡献矩阵中, 但是在前几次的迭代中的最优个体, 很有可能不及最后几代里的最差个体。 通过全局阈值, 对产生的所有个体来进行筛选。 大于全局阈值的个体将进入到贡献矩阵中, 计算每个波段的贡献率, 也就是贡献矩阵里每个波段组合中每个波段出现的次数, 依次剔除贡献率最小的波段, 对筛选后的波段组合建立PLS回归预测模型, 选择适应度值最大的波段组合作为最终的最优波段组合。
2.2.1 PLS-ACO
(1) 初始化算法的参数: 用全光谱波段数据建立PLS回归模型, 得到对应波段的回归系数(β )的绝对值作为蚁群的初始信息素矩阵(init-pheromone)。 最大迭代次数(max-iters), 蚂蚁个数(max-ants), 每只蚂蚁选取最大特征个数(max-features), 以及信息素启发因子(Q), 信息素挥发因子(ρ ), 入选特征路径矩阵(has), 未选路径矩阵(have), 全局阈值, 贡献矩阵(contribution)。
(2) 蚂蚁选择路径: 在每一次迭代中, 每只蚂蚁随机选择一个特征波段作为路径起始点, 并将其储存在has矩阵, have矩阵择则删除该特征波段, init-pheromone矩阵删除对应特征波段的信息素。 由init-pheromone矩阵, 计算每一个特征波段的被选概率, 用轮盘赌算法选择下一个特征波段, 直到所选特征波段达到max-features。
(3) 选取最优蚂蚁: 对所有蚂蚁选择的特征波段建立PLS回归模型, 计算出预测集真实值和预测值之间的
(4) 信息素更新: 仿造生物界蚂蚁觅食行为更新信息素, 选取每一次迭代中, 适应度值最高的蚂蚁进行信息素的更新。 将最优蚂蚁所选择的特征波段对应的回归系数绝对值, 以及适应度值作为信息素更新的依据, 对应波段的信息素按照信息素更新公式得到加强。 没有被选择的特征波段, 信息素会因为挥发, 浓度慢慢变小。 具体信息素更新公式如式(1)所示。
重复步骤2— 步骤4, 直至达到设置的最大迭代次数, 通过设置threshol, 得到最终contribution矩阵。 剔除contribution矩阵中, 选取的特征波段完全相同的特征波段组合。 计算每个波段的贡献率, 依次剔除贡献率最小的波段, 对选择的特征波段组合建立PLS回归模型, 选择适应度值最大的波段组合作为最终所选择的最优特征波段组合。
2.2.2 PLS-ACO-GA
具体步骤:
在PLS-ACO的基础下, 结合遗传算法[8], 进行如下步骤: (1) 初始化算法的参数: 遗传算法交叉概率(pc), 变异概率(pm)。
(2) 初始种群生成: 在PLS-ACO算法中, 将每一次迭代中, 每一只蚂蚁所选择的特征波段组合作为遗传算法中的个体, 所有蚂蚁选择的特征波段组合构成了遗传算法中的初始种群。
(3) 选择运算: 用轮盘赌算法淘汰那些适应度值低的个体。 为避免最优个体的遗失, 每一次轮盘赌选择过后, 如果最优个体被过滤了, 则将最优个体代替适应度值最低的个体, 重新保留在种群中, 以便于通过交叉产生更多优秀的个体。
(4) 交叉运算: 随机生成一个0到1之间的数, 如果该数大于pc, 随机选择一个个体作为母代, 进行交叉操作。 本研究使用单点交叉操作, 即随机生成一个交叉点, 交换该点前后的特征波段。 结果生成两个新的个体, 将其建立PLS回归模型, 计算其适应度值, 并与父代母代进行比较, 若子代比父代母代更加优秀, 则进行替换, 对应的父代或者母代淘汰。
(5) 变异运算: 本工作使用单点随机变异, 随机生成一个0~1之间的数, 若该数大于变异概率pm, 随机选择一个特征, 改变其是否被选择的状态。
(6) 个体选取: 如果该个体的适应度值大于全局阈值, 则将该个体的路径存入贡献矩阵中。
重复步骤(1)— 步骤(6), 直至达到设置的遗传最大迭代次数, 将通过全局阈值的特征波段组合存入贡献矩阵中, 进入下一次PLS-ACO算法迭代中。
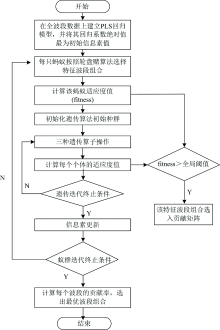
PLS-ACO-GA算法流程如图2所示。
2.2.3 特征波段选择方法结果与比较
通过多次反复实验, 根据经验将蚁群算法中最大迭代次数设置为50, 蚂蚁个数设置为80, ρ 设置为0.4, Q设置为0.2, 遗传迭代次数为30, pc设置为0.5, pm设置为0.01, threshol最小设置为9.564 5(全波段PLS建模下
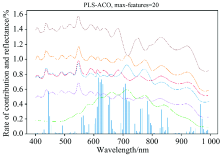
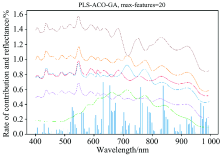
表2和表3分别是指设置不同max-features时, PLS-ACO和PLS-ACO-GA所选特征波段组合建立PLS回归模型时得到的结果。
| 表2 PLS-ACO, max-features分别设置为10, 20, 30的PLS回归模型结果 Table 2 PLS regression model results with PLS-ACO and max-features set to 10, 20 and 30 respectively |
| 表3 PLS-ACO-GA, max-features分别设置为10, 20, 30的PLS回归模型结果 Table 3 PLS regression model results with PLS-ACO-GA and max-features set to 10, 20 and 30 respectively |
从实验结果可知, 当max-features大小设置相同时, PLS-ACO和PLS-ACO-GA相比全波段PLS回归建模结果, 在提高了模型精度的同时, 只需要更少的特征波段。 PLS-ACO-GA方法相比PLS-ACO具有有更加优异的表现, 分析认为加入遗传算法后, 种群将变得更加的丰富, 为贡献矩阵提供了更多优秀的特征组合, 最后按照波段贡献率来选择最终的特征波段组合, 很多优秀随机特征波段组合共同构成了一个稳定的特征波段组合。 加入遗传算法后, 在一定的程度上避免了蚁群算法陷入局部最优; 另一方面, 选出的波段组合也更加具有代表性。 从表3数据可知, 在PLS-ACO中, 虽然
为了证明PLS-ACO和PLS-ACO-GA算法的优越性, 用CARS算法, 遗传算法[6]和传统蚁群算法对牛奶光谱数据进行特征波段选择来进行对比, 对最终选择的波段组合建立PLS回归模型, 以
| 表4 不同特征选择方法下的PLS回归模型结果 Table 4 PLS regression model result sunder different feature selection methods |
在使用PLS-ACO和PLS-ACO-GA算法选择特征波段组合的过程中, 一直选用PLS回归模型的精度作为评价特征波段组合的好坏标准, 很容易陷入此特征波段选择方法只针对PLS回归模型有效果的情况。 用多元线性回归(multiple linear regression, MLR)模型和随机森林回归(random forest regression, RFR)模型再次对PLS-ACO和PLS-ACO-GA这两种算法的适用性进行验证。 PLS-ACO和PLS-ACO-GA算法选取特征波段组合的数据进行MLR和RFR模型结果如表5和表6所示。 实验数据表明, PLS-ACO和PLS-ACO-GA算法在其他模型上也取得了不错的效果。 在MLR模型中, 对比全波段光谱数据, 虽然
| 表5 PLS-ACO和PLS-ACO-GA多元线性回归模型结果 Table 5 PLS-ACO and PLS-ACO-GA MLR results |
| 表6 PLS-ACO和PLS-ACO-GA随机森林回归模型结果 Table 6 PLS-ACO and PLS-ACO-GA RFR results |
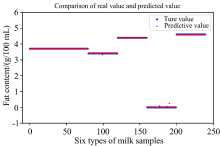
通过对比不同的预处理方法, 特征波段选择方法, 和不同的回归模型, 选择MSC作为预处理的方法, 用PLS-ACO-GA来进行特征波段选择, 最后建立RFR模型得到的牛奶脂肪含量预测最为精准。 将通过MSC-(PLS-ACO-GA)-RFR建模后得到的脂肪含量预测值与真实值做图对比, 以便更加直观的感觉到预测效果, 结果如图5所示。
从图5中可以看出, 只有个别样本真实值和预测值偏差较大, 大多数样本真实值和预测值基本重合, 预测结果表现优异。
利用高光谱技术进行牛奶脂肪含量检测研究, 使用不同预处理方法处理原始光谱数据, 结果MSC取得了最优异的结果; 进行特征波段选择, 提出了PLS-ACO和PLS-ACO-GA这两种方法, 通过与CARS算法, 遗传算法和基本蚁群算法进行对比, 在选取数目更少的基础上, 进一步提高了模型的精度, 最后比较不同预测模型方法, 在MLR, RFR和PLS回归模型中, RFR预测模型取得了最好的效果,
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|