作者简介: 张 杰, 1997年生,西安科技大学测绘科学与技术学院硕士研究生 e-mail: zhangj0216@163.com
利用高光谱遥感技术在水稻收获前对籽粒品质相关的蛋白质含量进行监测, 一方面可以及时调整栽培管理方式, 指导合理追肥, 另一方面, 有助于提前掌握籽粒品质信息, 明确市场定位。 该研究以广东省典型优质籼稻为研究目标, 基于2019年和2020年两年氮肥梯度实验, 以水稻分化期和抽穗期冠层尺度高光谱数据、 水稻氮素参数, 包括叶片氮素含量(LNC)、 叶片氮素积累量(LNA)、 植株氮素含量(PNC)、 植株氮素积累量(PNA)及籽粒蛋白含量数据为基础, 利用四种个体机器学习算法partial least square regression (PLSR)、 K-nearest neighbor (KNN)、 Bayesian ridge regression (BRR)、 support vector regression (SVR), 三种集成学习算法random forest (RF)、 adaboost、 bagging, 针对水稻不同生育期氮素状况进行监测建模, 在此基础上构建基于水稻冠层光谱信息、 光谱信息结合水稻农学氮素参数的籽粒蛋白含量的监测模型, 并对模型进行精度对比。 研究结果表明, 在水稻氮素营养监测方面, 利用水稻冠层454~950 nm波段信息, 采用RF及Adaboost算法, 在水稻分化期、 抽穗期及全生育期LNC、 LNA、 PNC及PNA模型 R2均达到0.90以上, 同时也具有较低的RMSE和MAE。 在水稻籽粒蛋白品质监测方面, 采用全波段光谱信息进行籽粒蛋白含量监测时, RF具有最高的精确度与稳定性, 两生育期的RF模型对籽粒蛋白含量的监测结果 R2分别为0.935和0.941, RMSE分别为0.235和0.226, MAE分别为0.189和0.152; 两生育期以全波段光谱信息结合长势参数进行籽粒蛋白监测时, Adaboost模型具有最高的精确度和稳定性, 其中分化期全波段光谱信息结合PNA作为输入参数, Adaboost模型 R2为0.960, RMSE为0.175, MAE为0.150, 以抽穗期全波段光谱信息结合PNC作为输入参数, R2为0.963, RMSE为0.170, MAE为0.137。 研究结果表明, 与PLSR, KNN, BRR和SVR几种个体学习器算法相比, 集成算法RF, Adaboost和Bagging具备良好的处理多重共线性的能力, 适合用于高光谱数据的分析与处理, 在作物氮素营养监测及水稻品质的早期遥感监测方面具有明显优势。
The use of hyperspectral remote sensing technology to monitor the protein content related to grain quality before rice matures is important. It can promptly adjust cultivation management methods and guide reasonable fertilization and help to grasp rice grain quality information in advance and clarify market positioning. This study took typical high-quality Indica rice in Guangdong Province as the research goal. Two-year nitrogen gradient experiments were carried on in 2019 and 2020. The canopy level hyperspectral data and rice nitrogen parameters, including leaf nitrogen content (LNC), leaf nitrogen accumulation (LNA), plant nitrogen content (PNC), and plant nitrogen accumulation (PNA), were collected at the rice panicle initiation stage and heading stage. Then, four individual machine learning algorithms, Partial Least Square Regression (PLSR), K-Nearest Neighbor (KNN), Bayesian Ridge Regression (BRR), Support Vector Regression (SVR), and three ensemble learning algorithms, Random forest (RF), Adaboost, Bagging were used for monitoring and modeling the nitrogen status of rice at different growth stages. After that, the rice grain protein content estimation models based on rice canopy spectral information, and spectral information combined with rice nitrogen parameterswere constructed by different machine learning algorithms. The rice nitrogen and grain protein content estimation models' accuracy were evaluated and compared. The study results showed that for rice nitrogen nutrition monitoring, using the rice canopy spectral information from 454~950 nm, the R2 of LNC, LNA, PNC and PNA estimation models based on RF and Adaboost algorithms achieved above 0.90 at the rice, heading stage, with low RMSE and MAE. Panicle initiation stage When using full-band spectral information to estimaterice grain protein content, RF had the highest accuracy and stability, with R2 of 0.935 and 0.941, RMSE of 0.235 and 0.226, and MAE of 0.189 and 0.152 at rice panicle initiation and heading stage, respectively. Adaboost model has the highest accuracy and stability for seed protein monitoring with full-band spectral information combined with growth parameters at both fertility stages, at the panicle initiation stage, the Adaboost algorithm with full-band spectral and PNA data can reach the bestfor rice grain proteinestimation, the R2, RMSE and MAE was 0.960, 0.175, and 0.150. While at heading stage, the R2,RMSE and MAE was 0.963, 0.170, 0.137,when using Adaboost algorithm with the full-band spectral and PNC data as input parameters. The results showed that the ensemble algorithms RF, Adaboost and Bagging have good ability to deal with multiple covariance compared with several individual learner algorithms PLSR, KNN, BRR and SVR.And they are suitable for the analysis and processing of hyperspectral data, which have obvious advantages in crop nitrogen nutrition monitoring and rice quality early monitoring through remote sensing.
水稻是我国种植面积最大、 覆盖范围最广的粮食作物, 长期以来我国注重水稻产量发展, 以解决人民生活温饱为目标, 近年来, 随着人民生活消费水平不断提高, 对稻米的需求也从过去的“高产”向“品质-食味”转变, 获取高产、 高品质的水稻是我国实施精准农业的新要求[1]。 稻米蛋白质含量是决定水稻营养品质的重要指标, 氮素是水稻生长发育的关键因素, 既影响水稻营养代谢、 物质积累, 也影响稻米最终营养及食味品质。 高光谱遥感技术是实现对水稻氮素营养及品质进行绿色无损监测的重要途径, 建立水稻氮素营养及籽粒蛋白含量监测模型, 是精准制定田间管理措施、 快速评估水稻品质的可靠依据[2]。 一些学者已利用高光谱遥感对水稻氮素状况及籽粒蛋白含量等品质相关参数进行了深入研究[3, 4]。 张浩等采用主成分分析(principal component analysis, PCA)对200~1 100 nm之间的光谱波段进行降维, 选用贡献率最高的两个主成分作为模型的输入变量, 对水稻叶片氮素含量及籽粒蛋白含量进行预测, 模型决定系数R2达到0.847以上[5]。 孙雪梅等探讨了不同氮素水平下的水稻光谱曲线特征, 利用统计相关分析, 研究了9个植被指数和8个微分参数与叶片叶绿素、 全氮含量的相关关系, 建立了叶绿素和全氮含量的监测模型, 并通过叶绿素监测模型间接对水稻籽粒蛋白含量进行预测[6]。 刘芸等分析了米粉光谱与蛋白质含量、 直链淀粉含量的关系, 通过提取敏感波段的特征参数, 利用多元逐步回归构建的模型决定系数R2达到0.7以上, 检验精度也达到了80%以上[7]。
目前对高光谱数据的处理大多通过主成分分析等方法实现对数据的特征降维, 以较少的特征参数参与模型的构建, 在降低模型构建难度的同时可能会丢失部分有效信息, 而机器学习模型具备强大的处理高维数据与冗余数据的能力, 基于更高效益的数学方法与数据处理方式实现对数据中有效信息的提取[8, 9]。 Bao等在小麦品种快速分类模型的构建中, 使用连续投影算法(successive projections algorithm, SPA)、 主成分分析算法(PCA)和随机蛙跳(random frog, RF)三种特征提取方法, 从数百个光谱波段中筛选可用于建立分类模型的光谱变量, 使用线性判别分析(linear discriminant analysis, LDA)支持向量机(support vector machine, SVM)、 极限学习机(extreme learning machine, ELM)三种机器学习算法分别以全波段和经过特征筛选的波段作为输入变量进行小麦品种分类模型构建, 以全波段作为输入变量的ELM算法分类精度最优[10]。 上述研究大多使用个体学习器的机器学习方法进行建模, 相比于个体学习器, 集成学习往往具备更好的稳定性和更高的精度。 Chan等使用RF和Adaboost对航空高光谱图像进行了生态区分类研究, 并评估了分类精度, 表明RF和Adaboost的精度均优于神经网络分类器[11]。 Gislason等对RF, Boosting和Bagging三种集成方法进行了土地覆盖分类的精度对比研究, 表明RF在与其他集成学习方法精度相当的同时, 拥有更快的训练速度并且不会过拟合[12]。 Pham等在边坡稳定性问题上应用集成学习建立了分类模型, 并与八种传统机器学习模型进行了对比, 模型的平均F1分数、 准确度与ROC曲线下面积(AUC)分别提高2.17%, 1.66%和6.27%, 认为集成学习中的极端梯度增强集成分类器(XGB-CM)更适用于滑坡风险评估问题[13]。
基于遥感的植被理化参数的监测有助于适时、 精准地获取作物氮素营养信息, 是作物肥水诊断及管理决策的基础。 而水稻品质形成与其生长过程中氮素营养代谢的合成与转运直接相关, 结合作物光谱响应机理与碳氮代谢转运机制进行作物蛋白质含量预测具有可行性[14]。 本文在前人研究的基础上, 选择水稻关键生育期冠层全波段光谱数据, 分别基于四种个体学习器算法(PLSR, KNN, BRR, SVR)及三种集成学习算法(RF, Adaboost, Bagging), 开展: (1)水稻氮素营养遥感监测; (2)水稻籽粒蛋白含量遥感监测, 将集成学习应用于水稻生长参数及品质的遥感监测, 通过研究, 探索其适用性, 分析不同算法在水稻氮素营养监测中的优劣, 同时筛选水稻籽粒蛋白含量遥感建模的最优算法及最优变量因子, 为水稻品质的监测应用提供依据。
2019年—2020年在广东省广州市白云区钟落潭试验基地(23° 23'24″N—23° 23'59″N, 113° 25'48″E—113° 26'24″E)开展水稻变量施肥的小区实验。 试验基地内, 2019年的试验品种为美香占2号(V1), 插秧时间2019年8月8日, 插秧密度为20 cm×20 cm, 试验共设计15个小区采样测试; 每个小区插秧规格为16穴×16穴。 2020年的试验品种为美香占2号(V1)和五丰优615(V2), 插秧时间为2020年8月8日, 共30个小区, 插秧密度为20 cm×20 cm, 根据插秧规格, 每个小区16穴×20穴。
2019年及2020年试验共设计5个氮素水平(N0, N1, N2, N3, N4), 分别为0, 60, 120, 180, 240 kg N· ha-1, 设3次重复; 其中基肥、 分蘖肥、 穗肥的施用比例为5:2:3; 磷、 钾肥用量分别为54和144 kg· ha-1。 分化期(2019.9.13, 2020.9.10)和抽穗期(2019.10.11, 2020.10.9)进行田间植株取样, 获取水稻叶片及植株氮素含量数据, 水稻品质数据分区于2019年及2020年水稻成熟期获取, 其中2019年每时期获取15个样本, 2020年每时期获取30个样本, 数据获取情况见表1。
| 表1 试验数据获取情况 Table 1 Test data acquisition |
水稻冠层高光谱数据采集使用的是美国ASD Filed Spec Pro 2500背挂式野外光谱仪, 仪器采集的光谱范围为350~2 500 nm。 根据前人的研究, 作物光谱的可见光与近红外范围已能够反映作物的生长状况, 因此本次实验采用454~950 nm的冠层光谱数据, 重采样后间隔为4 nm[15, 16]。 测量时间为北京时间10:00—14:00, 期间天气晴朗, 在每一个采样点测量前后均用标准白板对冠层辐亮度数据进行校正。 冠层光谱数据采集时距离冠层高度约1 m, 探头垂直向下, 探头角度为25° , 每个采样点取10次测量平均值作为该样方的冠层辐亮度值。 同一年的试验中, 记录采样点的位置, 保证不同生育期同一小区在相同位置采集数据。 对测定的冠层辐亮度和白板辐亮度利用式(1)计算目标的光谱反射率。
式(1)中, R为冠层反射率, Ltarget为冠层辐亮度(μW· cm-2· nm-1· sr-1), Lboard为白板辐亮度(μW· cm-2· nm-1· sr-1), Rboard为白板反射率。
光谱测量完成后, 在小区内随机选择6穴水稻植株样本, 去根并逐丛计数茎蘖数, 分化期茎叶分离, 抽穗期将茎叶和穗分离, 分开放入105 ℃烘箱杀青30 min, 并在80 ℃下干燥至恒重, 分别称重并记录。 然后使用凯氏定氮仪分别测量茎、 叶、 穗的氮含量。 计算公式如式(2)
式(2)中, NC为氮含量(%), V为盐酸体积(mL), M为样品质量(g)。
根据采样点种植密度和水稻样本的干重计算单位面积叶片和植株的生物量和氮积累量, 计算公式如式(3)
式(3)和式(4)中, LNA为叶片氮积累量(kg· m-2); PNA为植株氮积累量(kg· m-2); LAGB, SAGB和EAGB分别为测试样本中叶片、 茎、 穗的生物量(g· m-2); LNC, SNC和ENC分别为叶片、 茎、 穗的氮浓度(%)。 在分化期时, 由于水稻的穗还未发育, 只计算叶片和茎的相关参量即可。
于成熟期逐小区实收125丛稻株(5 m2), 水稻植株脱粒, 籽粒晒干3个月后, 脱壳碾磨成精米, 然后磨细成粉, 采用半微量凯氏定氮法测定籽粒氮素含量, 籽粒蛋白质含量(%)=籽粒氮素含量×5.95。
机器学习中根据算法的构建形式, 可以将其分为个体学习器与集成学习器, 其中个体学习器各自遵循独立的学习策略对目标进行预测, 而集成学习则是将多个已有的个体学习器通过某种策略结合起来, 建立一个新的学习器, 最终以多个基学习器的预测结果的均值或加权均值作为最终预测结果。 本文选择了四种基于不同理论的个体机器学习算法, 三种基于不同构建思想的集成学习算法, 研究两类算法在水稻氮素营养与籽粒蛋白品质监测上的优缺点。
个体学习器算法包括PLSR, KNN, BRR和SVR, 其中PLSR通过最小化误差的平方和, 寻找一组新的潜在变量来解释自变量X与因变量Y之间存在的统计关系, 是一种常见的对数据进行降维处理、 解决数据多重共线性问题、 简化建模过程的方式[17]。 KNN是通过k个最接近的邻居计算与预测因子之间空间相似性关系进行预测, 常被用来分类问题, 后来逐渐应用于参数估计[18]。 BRR是基于贝叶斯方法与最小二乘法的改进而提出的, 通过对线性贝叶斯回归模型加入L2正则化, 结合相关参数的先验信息形成先验分布并给出预估数值[19]。 SVR的基本思想是通过寻找最优划分超平面, 忽略小于偏差ε 的的样本, 对其他样本进行回归, 偏差ε 的引入是SVR区别于传统回归模型的地方, 即以预测y值为中心, 与真实y值之间存在一个宽度为2ε 的区域, 在此区域内, 预测y值与真实y值的差别认为是0。 其回归模型为f(x)=wTx+b, w和b为模型待确定参数[20]。
集成学习算法包括Bagging, RF和Adaboost, 其中Bagging的个体学习器的训练集通过自助采样得到, 每个个体学习器采用的训练集不同, 但个体学习器权重相同, 在每个个体训练完之后进行平均, 从而得到更高的准确性; RF是Bagging以决策树为基学习器的拓展变体, 并进一步引入属性的随机选择, 抗噪性能和泛化性能有所提高, RF和Bagging均属于并行化集成; Adaboost是每个个体使用相同的训练集, 但每轮训练中样本权重不同, 并且后一个学习器的运行依赖前一个学习器的结果, 运行过程中不断优化和提升, 最终将一族弱学习器提升为损失函数极小的强学习器, 属于序列化集成[21, 23]。
利用PLSR, KNN, BRR, SVR, RF, Adaboost和Bagging七种算法构建水稻氮素参数及籽粒蛋白含量预测模型时, 采用k-fold交叉验证方法(k=5)进行建模。 采用决定系数(R2), 均方根误差(root mean square error, RMSE), 平均绝对误差(mean absolute error, MAE)三个指标联合验证模型预测精度, R2越大, 代表模型拟合度越高, RMSE和MAE越小, 模型稳定性越好。
2019年与2020年不同生育期水稻氮素参量与籽粒蛋白含量进行相关性分析, 分化期LNC, LNA, PNC, PNA与籽粒蛋白含量的相关性系数分别为0.452, 0.794, 0.499和0.804, 抽穗期LNC, LNA, PNC, PNA与籽粒蛋白含量的相关性系数分别为0.787, 0.774, 0.824和0.756, LNC与PNC在分化期与籽粒蛋白含量的相关性低于抽穗期, PNC较分化期提高LNA与PNA在分化期与抽穗期均具有较好的相关性, 且除分化期的LNC外, 其余氮素参量均达到了0.001水平显著。 同时, 四种氮素参量之间相关性为0.7左右, LNC和LNA代表叶片尺度的氮素含量与积累情况, PNC和PNA表示植株地上部分整体的氮素含量与积累情况, 由于水稻籽粒蛋白的形成是一个动态的生物学过程, 同一时期, 不同部位与不同形式的氮素参量可能对籽粒蛋白的形成具备不同的转运与作用机理, 故建模过程中分别加入LNC, LNA, PNC和PNA四个参量探究其对水稻籽粒蛋白形成的影响。
2.2.1 冠层光谱与水稻氮素参数的相关性分析
图1是两年试验不同生育期水稻冠层光谱与氮素相关参数的相关系数图。 整体来看, 所有长势参数在两生育期相关系数有所不同, 但均具有相似的变化趋势, 近红外部分均保持在某一值持平, 整体变化幅度很小且以正相关为主, 可见光部分则以负相关为主。 所有长势参数在550 nm附近出现相关性“低谷”, 相关系数低于其他可见光部分。 在可见光区域与近红外区域的交界处, 光谱反射率受叶片内细胞间隙折射率不同的影响, 反射率急剧增加, 相关系数迅速由负转正, 有明显的降低后再抬升的趋势。
 | 图1 2019年与2020年水稻不同生育期冠层光谱与氮素参数相关性(n=45)Fig.1 Correlation between canopy spectrum and nitrogen parameters at different growth stages of rice in 2019 and 2020 |
LNC随着生育期的推进, 与冠层反射率相关性在全波段均有所提高, 分化期最大相关系数为0.450(950 nm), 抽穗期最大相关系数达到0.585(942 nm)。 LNA在可见光部分与冠层光谱的相关性分化期高于抽穗期, 而在近红外部分则相反, 两生育期最大相关系数分别为-0.602(662 nm), 0.662(950 nm)。 PNC与LNC具有相似的趋势, 在分化期与冠层反射率相关性普遍较低, 最大相关系数仅为0.431(950 nm), 抽穗期PNC与冠层反射率的相关性在可见光部分与近红外部分均有较大提升, 特别是近红外部分, 在750~950 nm区间, 最大相关系数达到0.710(940 nm)。 分化期PNA在可见光部分的658 nm附近, 与冠层反射率存在较好的相关性, 最大相关系数为-0.641(666 nm), 随着生育期的推进, 在抽穗期的近红外部分, PNA与冠层反射率的相关性优于分化期, 最大相关系数为0.663(922 nm)。
2.2.2 基于光谱信息的水稻氮素含量监测
基于2019年与2020年数据, 以全波段光谱作为输入参数, 分别采用PLSR, KNN, BRR, SVR, RF, Adaboost和Bagging七种不同算法构建分化期、 抽穗期及全生育期水稻氮素参数LNC, LNA, PNC和PNA监测模型。 结果如表2、 表3、 表4所示, 七种算法均能利用全波段光谱信息实现对各个氮素参数不同程度的表达。 其中, 分化期、 抽穗期及全生育期LNC的最优建模精度R2分别为0.927, 0.954和0.922, RMSE为0.110, 0.241和0.135, MAE为0.187, 0.185和0.107, LNA的最优建模精度R2分别为0.944, 0.948和0.943, RMSE为0.272, 0.591和0.440, MAE为0.230, 0.436和0.325, PNC的最优建模精度R2分别为0.930, 0.951和0.925, RMSE为0.084, 0.073和0.115, MAE为0.079, 0.057和0.091, PNA的最优建模精度R2为0.938, 0.920和0.952, RMSE为0.399, 1.346和0.978, MAE为0.353, 1.042和0.716, LNC在分化期、 抽穗期的最优监测模型为Adaboost, 全生育期最优监测模型为RF, 在进行LNA, PNC和PNA监测时, 均为RF模型表现最优, 表明RF方法在氮素监测时具有良好的适应性与精度。 这些参数和籽粒蛋白含量的相关性显著, 故利用作物生长前期的光谱数据进行籽粒蛋白含量监测是可行的。
| 表2 基于水稻分化期冠层光谱数据的氮素参数模型精度(n=45) Table 2 Model accuracy of nitrogen parameters based on canopy spectral data of rice at Panicle Initiation stage |
| 表3 基于水稻抽穗期冠层光谱数据的氮素参数模型精度(n=45) Table 3 Model accuracy of nitrogen parameters based on canopy spectral data of rice heading stage |
| 表4 基于水稻全生育期冠层光谱数据的氮素参数模型精度(n=90) Table 4 Model accuracy of nitrogen parameters based on canopy spectral data of rice whole growth period |
利用PLSR, KNN, BRR, SVR, RF, Adaboost和Bagging七种不同算法, 以水稻不同生育期冠层光谱数据, 以及冠层光谱数据结合氮素参量为输入参数, 构建水稻蛋白品质监测模型, 并分析对比模型精度。
2.3.1 基于水稻冠层光谱信息的籽粒蛋白含量预测
图2是基于不同算法, 利用水稻分化期与抽穗期冠层全波段光谱信息所建模型的精度对比结果。 由于采用分化期与抽穗期全波段光谱信息进行建模, 多重共线性可能是一个问题, 而不同算法处理多重共线性的能力不同, 各个算法在水稻籽粒蛋白含量的监测上表现差距较大。 从图2中可以看出, 分化期KNN、 PLSR的预测结果R2仅分别为0.538和0.580, 而RF、 Adaboost、 Bagging监测结果R2则分别达到了0.935, 0.916和0.874, 同时也具有更低的RMSE和MAE, 各个算法的监测能力依据R2排名为RF>Adaboost>Bagging>SVR>BRR>PLSR>KNN; 利用抽穗期数据进行预测时, 各个算法的监测精度均有提高, 监测能力依据R2及RMSE排名为RF>Adaboost>Bagging>SVR>BRR>KNN>PLSR。 三种集成算法(RF、 Adaboost、 Bagging)在处理多重共线性问题上表现出更为良好的性能。
 | 图2 基于水稻不同生育期冠层光谱数据籽粒蛋白含量模型的R2, RMSE和MAEFig.2 R2, RMSE and MAE based on the canopy spectral data seed protein content model of rice at different fertility stages |
2.3.2 基于水稻冠层光谱信息氮素参量的籽粒蛋白含量监测
利用PLSR, KNN, BRR, SVR, RF, Adaboost和Bagging七种不同算法, 分别以水稻不同生育期冠层光谱数据与四个不同实测氮素参量为输入参数, 构建水稻蛋白品质监测模型, 分析对比了模型验证精度, 图3显示了七种算法在两个不同生育期, 采用不同参数组合所构建模型的R2, RMSE和MAE变化的统计图, 以光谱数据结合不同氮素参数作为输入参数的模型, 与仅采用光谱信息所建立的模型相比, 大部分算法的监测精度和稳定性均得到了提升, 即在不同运行规则下的大部分算法认为氮素参数是监测籽粒蛋白含量的有效参数, 其含量的高低受到植株氮素的影响。
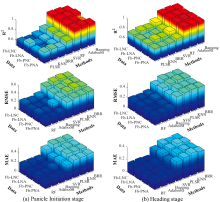 | 图3 分化期和抽穗期不同参数组合下7种算法的R2、 RMSE和MAEFig.3 R2, RMSE and MAE of seven algorithms under different parameter combinations at Panicle Initiation and Heading stage |
通过综合对比, 在分化期, 光谱信息结合PNA作为输入参数时, 各个算法的精度提升最明显, 较以光谱信息作为输入参数的模型, 各个算法R2分别提高0.131, 0.182, 0.013, 0.041, 0.020, 0.044和0.063; 在抽穗期, 以光谱信息结合PNC作为输入参数时, 各个算法的精度提升最明显, 较以光谱信息作为输入参数的模型, 各个算法R2分别提高0.073, 0.054, 0.028, 0.043, 0.013, 0.022和0.019。 在这两组输入参数下, 两时期均为Adaboost表现最优, RF和Bagging方法稍低于Adaboost, 但也表现极好, PLSR, KNN, BRR和SVR在氮素参数影响下, 模型精度提升更为明显, 但仍未能超过RF, Adaboost和Bagging。
图4给出了基于不同机器学习算法以分化期水稻冠层全波段光谱信息及水稻植株氮素累积量PNA作为输入参数的水稻蛋白品质模型预测值与实测值的散点图。
 | 图4 分化期以全波段光谱信息和PNA为输入的七种算法的R2, RMSE和MAEFig.4 R2, RMSE and MAE of seven algorithms with full band spectral information and PNA as input in Panicle Initiation stage |
图5给出了抽穗期以全波段光谱信息结合水稻PNC作为输入参数的水稻蛋白品质模型预测值与实测值的散点图。
 | 图5 抽穗期以全波段光谱信息和PNC为输入的七种算法的R2, RMSE和MAEFig.5 R2, RMSE and MAE of seven algorithms with full band spectral information and PNC as input at Heading stage |
从图4和图5中可以看出, 三种集成算法更有利于在相同输入参数下获取更高精度, PLSR, KNN, BRR和SVR算法预测实测值散点图围绕1:1线仍然存在一定离散点, 相比于单学习器的机器学习回归算法, 三种基于集成学习器的算法RF, Adaboost和Bagging的准确度及稳定性均明显提高, 且不同算法对输入参数具备不同的“适应性”, 例如, RF和Bagging算法获得最优精度时是抽穗期以光谱信息和PNA作为输入参数的, R2分别为0.958和0.943, Adaboost算法在水稻分化期、 抽穗期的其中结合作物冠层光谱数据及氮素信息(PNC及PNA)蛋白品质模型R2均达到0.96以上。
水稻品质形成与其生长过程中氮素营养代谢的合成与转运直接相关, 本研究获取水稻不同生育期冠层光谱及水稻氮素营养参数, 采用多种机器学习算法, 进行作物氮素营养监测及水稻籽粒蛋白质含量监测研究。 研究表明:
(1)基于水稻冠层光谱信息的水稻氮素参量遥感监测表明: 冠层光谱信息能够较好的表达不同生育期水稻的氮素营养状况, 其中基于不同算法的监测结果差异明显, 传统回归方法和部分机器学习方法并不能够较好利用光谱信息对水稻氮素营养状况的监测, 而RF和Adaboost对两时期的氮素参数监测结果R2能够达到0.90以上, 表明冠层光谱中与植株氮素营养相关的信息被较好的利用。 由于水稻关键生育期的氮素含量与其籽粒蛋白含量相关性显著, 因此利用分化期与抽穗期冠层光谱信息监测水稻籽粒蛋白品质是可行的。
(2)基于冠层光谱数据的水稻籽粒蛋白含量监测结果表明: 在利用作物冠层全波段信息可能存在强共线性的情况下, 七种算法的准确度与稳定性不同, 三种集成算法在水稻蛋白品质监测上具有明显优势, 其中RF算法两个不同生育期蛋白品质监测模型的R2分别为0.935, 0.941, Adaboost算法两个不同生育期蛋白品质监测模型的R2分别为0.916, 0.941; Bagging算法两个不同生育期蛋白品质监测模型的R2分别0.874, 0.920, RF, Adaboost和Bagging三种集成算法几乎不受多重共线性的影响, 对比单学习器的机器学习算法, 利用多个基学习器进行训练的算法能够解读更多籽粒蛋白含量与各种参数间尚未明确的关系, 为最终的监测目标挖掘更多的决策信息。
(3)将氮素参量及水稻冠层光谱信息作为模型输入因子, 进行水稻籽粒蛋白含量监测预测时, 模型精度得到进一步改善, 表明氮素参量对籽粒蛋白含量存在一定程度的影响, 其中在分化期以光谱信息和PNA作为输入参数时, 模型精度提升更明显(R2从 0.935提高到0.960), 在抽穗期以光谱信息和PNC作为输入参数时, 模型精度提升更明显(R2从0.941提高到0.963)。 利用抽穗期数据进行监测时, 获取的监测精度比分化期更高, 可能是抽穗期水稻株体发育趋于完善, 籽粒蛋白质的产生与植株氮素的关系更加明确, 故利用抽穗期数据进行水稻籽粒蛋白含量能够获取更高的精度。
实验结果表明, 全波段光谱携带的信息能够较好的对水稻氮素营养参数进行监测, 也表明利用分化期与抽穗期冠层光谱监测当季水稻籽粒蛋白含量是可行的, 七种方法中, 仅输入全波段光谱的时候, RF最优, 最大R2为0.941, 加入氮素参数后, Adaboost表现最优, 最大R2为0.963, Bagging也取得了较好的监测结果。 集成学习方法能够解决数据存在的多重共线性问题, k-fold交叉验证方法也在一定程度上避免了模型的过拟合, 利用所有信息进行回归建模, 较大程度的保留了与水稻氮素营养和籽粒蛋白含量相关的信息, 且简化了数据处理流程, 在实际农业监测中更有利于推广与应用。 论文在将不同参数组合作为自变量进行输入时只对各类参数简单拼接, 未考虑不同参数的权重分布, 不同参数与籽粒蛋白含量是否存在最优映射的关系有待下一步研究。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|


