作者简介: 刘美辰, 1997年生, 内蒙古农业大学计算机与信息工程学院硕士研究生 e-mail: lmcdrr@163.com
牛奶中包含着很多人体需要的营养元素, 如脂肪、 蛋白质、 钙等; 对牛奶营养元素进行分析是牛奶安全检测关键的一部分。 高光谱技术可以有效地结合图像和光谱数据识别牛奶种营养元素。 为了实现对牛奶中蛋白质含量快速、 精确的预测, 采用竞争性自适应重加权(CARS)算法选取特征波长, 并提出一种基于麻雀搜索算法(SSA)优化支持向量机(SVM)实现对牛奶蛋白质含量预测。 利用高光谱仪获取牛奶反射光谱(400~1 000 nm)。 通过选取归一化(N)、 标准化(Standardization)和多元散射校正(MSC)对原始的牛奶数据进行光谱降噪处理提高光谱利用率; 利用竞争性自适应重加权算法和连续投影算法(SPA)对经过处理的牛奶光谱数据提取特征波长, 求取蛋白质和光谱间的相关系数并进行重要性排序, 获取重要的特征波段; 最后, 通过遗传算法(GA)优化SVM, 粒子群算法(PSO)优化SVM和偏最小二乘法(PLS)算法对牛奶蛋白质进行预测并比较预测结果, 为了提高蛋白质预测的精度和模型稳定性, 提出利用SSA对SVM的核函数 g和惩罚参数 c进行优化, 以均方根误差(RMSE)作为适应度函数, 通过迭代选择最优的回归参数训练模型。 牛奶数据预测结果表明最优组合模型为: MSC-CARS-SSA-SVM。 模型测试集的决定系数 R2为0.999 6, 均方根误差RMSE为0.001 1, 耗时4.112 1 s。 结果表明: 使用CARS算法能实现特征波段的提取和冗余信息的剔除, 从而提高模型效率, 简化了算法的复杂度; SSA算法优化SVM的参数, 通过迭代更新麻雀最优位置, 可以快速得到全局最优解, 与SVM, GA-SVM, PSO-SVM和PLS相比, 牛奶蛋白质的预测准确度和模型稳定性都得到了明显提高, 满足了对乳品检测的精确度要求, 是快速检测牛奶蛋白质的一个可行新方法。 为光谱模型的优化及预测模型精度的提高提供参考。
Milk contains many nutritional elements needed by the human body, such as fat, protein, calcium, etc. Therefore, analysing nutritional elements in milk is a key part of milk safety detection. Hyperspectral technology can effectively identify nutritional elements in milk by combining image and spectral data. In order to quickly and accurately predict protein content in milk, the Competitive Adaptive Reweighted Sampling (CARS) algorithm was used to select characteristic wavelengths. A method based on Sparrow Search Algorithm (SSA) to optimize Support Vector Machine (SVM) was proposed to predict milk protein content. The reflectance spectra of milk (400~1 000 nm) extracted by the hyperspectral spectrometer were used for the experiment. During Normalization (N), Standardization and Multiplicative Scatter Correction (MSC), the original milk data are used for spectral noise reduction to improve spectral utilization. The successive projections algorithm (SPA) and the competitive adaptive re-weighting algorithm were used to extract the feature wavelengths from the processed milk spectral data. The correlation coefficients between proteins and the spectrum were calculated and ranked by importance to obtain the important feature wavelengths. In the end, through SVM, the Genetic Algorithm (GA)-SVM, Particle Swarm Optimization (PSO)-SVM and Partial Least-Regression (PLS) algorithm was used to predict milk proteins and compare the prediction results. In order to improve the accuracy of protein prediction and model stability, SSA was proposed to optimize the kernel function G and penalty parameter C of SVM. Root Mean Squared Error (RMSE) was used as the fitness function, and the optimal regression parameters were selected through iteration to train the model. The results of milk data prediction showed that the optimal combination model was MSC-CARS-SSA-SVM. The determination coefficient R2 of the model test set was 0.999 6, the root means square error RMSE was 0.001 1, and the time was 4.112 1 seconds. The results show that the CARS algorithm can extract the characteristic bands and eliminate redundant information, thus improving the efficiency of the model and simplifying the algorithm's complexity. The SSA algorithm optimizes SVM's parameters and can quickly obtain the global optimal solution by iteratively updating the optimal position. Compared with SVM, GA-SVM, PSO-SVM and PLS, the prediction accuracy and model stability are significantly improved, which meets the accuracy requirements of milk detection, and is a feasible new method for fast detection of milk protein. It provides a theoretical reference for the optimization of spectral models and the improvement of prediction model accuracy.
牛奶作为天然乳液, 包含着很多人体需要的营养元素。 近些年, 对牛奶中蛋白质的含量检测日益得到社会的重视。 最常见的乳品检测方法为化学分析法, 虽然准确度高, 但检验过程复杂且成本大。 高光谱利用400~1 000 nm内的光谱波段对牛奶进行检测, 不仅操作简单、 预测快速无损, 而且效果好。
国内外很多学者对牛奶中的营养元素光谱检测做了研究。 Laverroux等使用液相色谱结合荧光法对牛奶中的维生素B2进行分析, 展示了在浓度区间内对营养元素预测的一种新方法[1]; 范睿等基于近红外光谱, 建立主成分回归模型对牛奶中的掺假蛋白质检测[2]; Lin等基于近红外光谱检测蛋白质, 通过运用支持向量机(support vector machine, SVM)和BP神经网络(back-propagation artificial neural network, BP-ANN)以及偏最小二乘法(partial least-regression, PLS)建模, 结合多种预处理方法, 最终得分最高的模型DOSC-KPLS的R2达到了0.974[3]。 使用高光谱对牛奶进行分析的研究也有报道, 近年, Munir等将光谱处理的奶粉图像空间属性与化学属性相结合, 使用PLS建立回归模型实时显示质量, 证明高光谱技术可以用于乳制品检测[4]。 赵紫竹等为了对牛奶中脂肪含量进行检测使用了NPLS算法[5]; 张倩倩等采用主成分回归和最小二乘支持向量机两个方法对牛奶中的蛋白质进行定量分析。 目前基于高光谱技术的牛奶定量分析中, 由于牛奶成分复杂, 原始光谱数据中变量间的相关性低, 模型的预测精度仍需要提高, 分析方法也需要提升。
综上, 针对牛奶分析精度和方法的需求, 实验以五种蛋白质含量不同的牛奶为研究样本, 测量其400~1 000 nm范围的高光谱反射数据, 结合蛋白质含量数据, 建立SSA-SVM模型, 通过综合考察多种预处理和特征波长提取方法对建模的作用。 并采用常应用于牛奶分析中的偏最小二乘法回归(PLS)算法、 具有强非线性拟合能力的支持向量机回归(SVM)算法以及两个基于SVM的优化算法GA-SVM和PSO-SVM作为对比, 找出测量牛奶蛋白质含量的最优方法, 为牛奶营养元素含量的定量检验提供参考。
所用的样品是从市面上购买的五种蛋白质含量不同的牛奶, 蛋白质含量分别为3.0, 3.2, 3.3, 3.4和3.6 g· (100 mL)-1。 培养皿的直径为90 mm高度为8 mm。 分别取不同批次的五种牛奶置于培养皿中, 用玻璃棒搅拌。 共250个样本, 其中训练集175个, 测试集75个。
利用图1所示HyperSpec VNIR高光谱仪采集样本的牛奶反射光谱。 光谱扫描范围在400~1 000 nm区间内 , 光谱的波段数为125个, 平均间隔为0.8 nm, 曝光时间选择10 ms, 像元混合次数为6次, 分辨率为4.8 nm, 采集的光谱图像像素为777× 1 004。 采集条件: 室温(23~25 ℃) , 牛奶样本置于光谱仪探头垂直下方的吸光黑色绒布上, 于暗室中测量。 卤素灯至样本40 cm的距离垂直照射, 扫描探头到样本的垂直距离为15 cm。 测量前, 对光谱图像标定, 分别采集白板和全黑的标定图像进行校正以消除因光源和暗电流存在导致的噪声[6]。
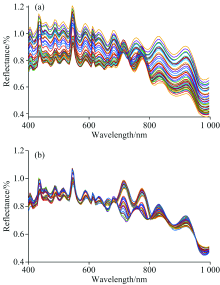
图2(a)为高光谱仪采集图像后经ENVI导出的牛奶样本原始高光谱曲线。
因测量时室内光线、 角度、 温度等因素可能对光谱数据造成误差, 光谱的噪声很大, 所以需对低质量的数据进行优化, 减小对目标结果的影响。 采取的预处理方法有: 归一化(normalization, N)、 标准化(standardization)和多元散射校正(multiplicative scatter correction, MSC)。
1.4.1 竞争性自适应重加权算法采样算法
通过竞争性自适应重加权算法(competitive adaptive reweighted sampling, CARS)选取特征波段, 算法基于蒙特卡洛采样结合偏最小二乘法(PLS), 由衰减指数法(EDF)来决定波长个数[7], 通过自适应重加权采样(> adaptive reweighted sampling, ARS)保留波长回归系数的权重值大的集合, 创建PLS模型, 引入交叉验证, 不断优化计算均方根误差(root mean squard error, RMSE), 选择RMSE最小的子集, 即模型精度最高的特征波长组合[8]。
1.4.2 基于优化算法的SVM参数优化
SVM作为一个经典的预测算法具有着较强的非线性拟合能力和鲁棒性, 并且因预测精度高和复杂度低的优势常被应用于牛奶定量分析的研究中, 但据预测结果表明其对于参数选取的敏感度高, 若选取不当会使性能降低[9]。 故需要选取合适的优化算法对SVM优化提高计算精度。 目前已有的对其改进的优化算法均可实现参数选取的要求, 但由于大多欠缺于局部性能, 所以计算精度仍需改进。
1.4.3 基于GA的SVM参数优化
遗传算法(genetic algorithm, GA)是一种常被应用于预测模型的全局优化算法。 其原理是通过生物作用机制, 对当前研究的种群进行筛选, 逐步选出适应度最高的个体[10]。 实验最大迭代次数和设置为200, 基因位置为参数值, 将参数带入SVM模型中, 对牛奶数据进行训练并计算个体的适应度值, 如达最大迭代次数, 则停止搜索。 输出全局最优的参数值, 实现对SVM的参数优化。
1.4.4 基于PSO的SVM参数优化
粒子群算法(particle swarm optimization, PSO)的原理是利用种群中的各粒子通过学习不断调整位置和速度来实现优化[11]。 实验最大迭代次数为200, SVM参数取值范围一定, 利用SVM对牛奶数据做出预测, 计算粒子的适应度值并更新其速度和位置, 当达到最大迭代次数时, 跳出循环, 输出全局的最佳参数并训练SVM模型, 实现PSO对SVM的参数优化。
1.4.5 基于SSA的SVM参数优化
麻雀搜索算法(sparrow search algorithm, SSA)是通过麻雀的捕食、 追随、 侦查三种行为为启发而创建[12]。 算法将麻雀种群分为发现者和跟随者, 麻雀属性为位置, 对应优化的解, 适应度值对应觅食位置。 其中发现者发现者和跟随者的位置是动态变化的, 凡是发现者会选择最好的位置觅食。
追随者会在发现者周围觅食或与发现者争夺食物, 当追随者发现更好觅食位置则更新发现者的解, 否则不变。 一旦发现危险, 边缘麻雀会迁移到安全区躲避危险, 同时就处于最佳位置的麻雀会在周围位置走动。 SSA基于以上步骤循环搜索最优解。 其对支持向量机算法进行优化的主要思路为用麻雀位置表示SVM的参数c和g, 通过对全局的适应度值排序, 求最优值和最优位置, 得知最优参数[13]。
SSA优化SVM的流程图见图3。
主要步骤如下:
(1) 参数初始化。 对麻雀种群初始化, 最大迭代参数itermax, 麻雀总数、 发现者和跟随者比例以及参数c和g的取值范围。
(2) 适应度设置。 适应度函数设置为SVM训练后的MSE误差
对全局的适应度值排序, 求最优值和最优位置。
(3) 更新发现者位置。
式(2)中, 迭代次数为t时, 第i只麻雀的j维位置信息, α 为(0, 1]的随机数, itermax为最大迭代次数, R2是[0, 1]中的一个随机数, 代表安全值, ST为警戒阈值, Q为一个标准正态分布随机数。 ST为警戒阈值。
(4) 更新追随者位置。
式(3)中, xworst为最坏位置, xb则为最好的位置。 A+=AT(AAT)-1, 其中, A为一个大小为1× D的矩阵, 当i> n/2时, 其值会收敛于0, 第i只获取食物少的麻雀需要更新位置获取食物。 当i≤ n/2时, 值收敛于最优位置。
(5) 更新侦查者位置。
式(4)中, xbest代表最优位置, fg表示全局的最优位置上的麻雀的最佳适应度值, fw为最坏位置上麻雀的适应度值, K是区间[-1, 1]内的一个随机值, ε 为一个为了防止分母为0的极小数。
(6) 计算各适应度值。 对麻雀的位置更新, 直到最大迭代次数。
(7) 如满足最优解, 则输出最优解c和g。 否则。 继续执行步骤(2)— (6)。
1.4.6 评价指标
预测模型的好坏依据以下几个参数评估: 决定系数(determination coefficients, R2)、 均方根误差 (root mean squared error, RMSE)、 耗时。 RMSE越小则表明预测的数据与原始数据重合度越高、 R2越趋近于1回归效果越好。
三类预处理的PLS模型精度如表1所示。 选用多元散射校正(MSC)预处理的模型的拟合程度最高, 预处理后的光谱曲线如图2(b)所示, 可以看出经过MSC处理后, 可以有效的消除一些由散射、 光程突变、 乳液不均匀带来的影响, 消除了牛奶光谱数据的噪声干扰。 从而提高了光谱的灵敏度和实用性。
| 表1 预处理模型精度对比 Table 1 Precision comparison of preprocessing models |
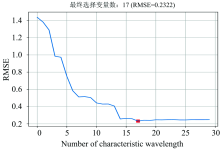
连续投影算法(successive projections algorithm, SPA)在食品检测分析中常被用于特征选择, 能从众多波段中选出冗余信息少的波段, 从而使共线性问题达到最小化, 减小模型变量, 提高运算效率[14]。 以经过MSC处理后的牛奶光谱值为输入变量, 通过反复迭代投影值, 对最终得到的特征变量进行回归分析, 以均方根误差RMSE最小的集合为特征波段集合。 选出的特征波段有17个。
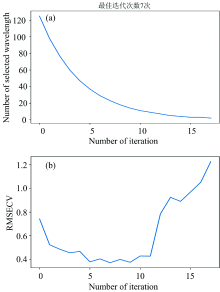
图5为采用CARS提取牛奶的全部波长变量进行特征提取后的运算结果图。 设置蒙特卡罗采样次数为20[15]。 图5(a)显示随着采样次数增加, 由于衰减指数EDF的作用, 开始在CARS的光谱粗选阶段中会用较大的学习率, 选择的波长数量快速下降, 到较优解的时候逐渐降低学习率来训练模型, 即CARS的精选阶段。 结果显示选取的变量数在前7次中下降很快, 之后下降速度减慢至平稳。 同时, 随着采样次数的增加, 部分无关波段被剔除导致RMSECV值整体呈现下降趋势, 图5(b)可知开始迭代时, 由于牛奶数据中大量和蛋白质无关的波段被消除, 故RMSECV值快速减小, 当采样次数到第7次时均方根误差达到最低值, 后由于部分重要信息被剔除, RMSECV值又整体呈上升趋势。 故当迭代次数为7时所选择的波长集合为最优集合。 7次采样得到27个特征波长。
惩罚参数c和核函数g是支持向量机中两个重要参数, 影响着预测模型的精度; 因此, 选取最优的c和g是SVM优化的关键[16], 也是本工作提出用SSA算法优化SVM的初心。 经反复调试, 麻雀算法的参数设置为: 麻雀数量N=20, 最大迭代次数Max_iteration=200; SVM的惩罚参数c和核函数g取值范围均为[2-5, 25]。 最终得到的最佳参数为: c=3.482 5, g=0.031 2。 也就是说, 用此最佳的c和g值, SSA-SVM模型的精度最高。
为了考察SSA-SVM模型的预测性能, 选取PLS和用遗传算法(GA)、 粒子群算法(PSO)优化的SVM模型比较。 将CARS和SPA分别选取出的27和17个特征波段作为几个模型的输入变量; GA-SVM的最佳参数为c=7.221 8, g=0.043 5, PSO-SVM的最佳参数为c=17.187 2, g=0.051 2。 将最佳参数代入模型, 预测牛奶的蛋白质含量, 通过对比各个模型的精度和耗时, 选取出最优的牛奶蛋白质预测模型。
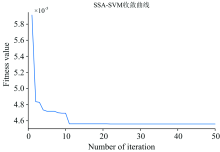
对比蛋白质含量预测精度: 全波段数据作为输入变量时, SSA-SVM算法较PLS算法准确率提高了5.83%, 较SVM算法准确率提高6.03%, 较GA-SVM算法和PSO-SVM算法准确率分别提高了2.68%和5.2%。 表明SSA-SVM为牛奶蛋白质最佳的预测模型, 可以有效的预测牛奶中蛋白质含量。 从图6可以看出SSA-SVM算法的适应度值随适应度迭代次数增加逐渐趋近最优值, 到达最低点后保持稳定。
表2显示将全部125个波段、 CARS提取的27个特征波段和SPA提取的17个特征波段分别作为输入变量时, 利用五种不同的预测模型对牛奶蛋白质预测的精度对比。 当全波段作为输入时, SSA-SVM模型对蛋白质含量预测可以满足预测要求, R2=0.988 9, RMSE=0.009 2, 耗时6.056 9 s。 SVM、 PLS两种经典回归模型耗时虽短, 但准确率较低。 GA-SVM和PSO-SVM虽然精度较传统SVM得到提高, 但仍低于本文提出的SSA-SVM模型, 并且耗时过长。 由表2可以更直观的看出不同模型针对牛奶数据的预测效果。 预测效果排序为: SSA-SVM> GA-SVM> PSO-SVM> PLS> SVM。 且在特征波段选择时, CARS的效果好于SPA, 当以SSA-SVM为预测模型时, CARS选择的波段作为输入变量准确度比SPA选择波段高0.12%, 比全波段高1.07%。
| 表2 各模型精度对比 Table 2 Precision comparison of different models |
结果表明, SSA算法较于GA、 PSO算法对SVM的优化更具有优势, 不容易陷入局部最优, 有效提高了牛奶蛋白质预测的准确率, 且迭代时间得到明显缩短。 PLS和SVM算法虽耗时短, 但预测准确率低, 在牛奶蛋白质预测中不具有优势。
表2可知: CARS-SSA-SVM的预测效果最好, R2=0.999 6, RMSE=0.001 1, 耗时为4.112 1 s。 预测结果如图7所示。
以五种蛋白质浓度不同的牛奶作为实验对象建立SVM, PLS, GA-SVM, PSO-SVM和SSA-SVM五种不同的模型, 研究了利用牛奶的高光谱反射率对蛋白质含量进行预测, 结论如下:
(1) MSC算法可以有效的对原始牛奶光谱数据预处理, 降低了噪声干扰, 消除了光谱差异, 增强了光谱和数据的相关性, 有效降低了牛奶蛋白质预测模型的误差, 提高了准确率。
(2) 使用CARS和SPA算法对牛奶数据进行特征提取, CARS算法较SPA算法更能增强了光谱和蛋白质含量之间的相关性, 消除了冗余信息, 大大降低了模型输入变量多导致的复杂性, 同时模型的预测效率也得到了有效的提高。
(3) 采用SVM, PLS, GA-SVM, PSO-SVM和SSA-SVM五种回归算法对比实验。 其中利用SSA-SVM算法选择最佳参数的模型具有较高的精度。 在CARS的特征波长作为输入变量时, 验证集R2=0.999 6, RMSE=0.001 1, 相较于另外四个模型, 稳定性和准确性得到明显提高, 满足了预测要求。
(4) SSA-SVM牛奶营养含量分析方法精度高、 实验过程简便、 耗时小。 是牛奶中蛋白质含量预测的新方法。 下一步目标是有关牛奶营养元素的含量建立更加理想化的预测模型, 对有效波长的选择进行更深入的研究分析。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|