作者简介: 谭 阳, 2000年生, 江苏大学卓越学院本科生 e-mail: TaNvYaN@126.com
红外光谱分析是基于分子振动与跃迁理论的鉴别物质化学组成的技术。 得到的光谱数据常常具有较高的维数和重叠度, 这给后续的数据处理带来困难。 为此提出一种GK可能C均值聚类算法(GKIPCM), 引入了GK聚类算法的马氏距离测度与改进的可能C均值聚类算法(IPCM)的模糊隶属度与聚类中心更新方程, 使样本的距离测度具有自适应性且避免了聚类中心的一致性。 GKIPCM算法具有分类精度更高, 分类准确率对参数敏感性低的优点。 将四组洗净白菜作为光谱分析对象, 分别施加三种农药(高效氯氟氰菊酯)配比, 采用安捷伦Cary 630 FTIR光谱仪采集白菜的傅里叶中红外光谱(FT-MIR)。 首先对样本进行预处理, 使用多元散射矫正(MSC)对光谱数据降噪, 消除数据偏移量; 其次, 由于采集到的数据波数范围为4 300~590 cm-1, 数据维数达到了971维, 故使用主成分分析(PCA)对数据实现降维, 降维后的数据维度减小到了23, 且23个主成分的累积贡献率高达99.60%; 但各类光谱的特征信息依然混杂在一起, 故使用线性判别分析(LDA)提取特征鉴别信息, 进一步将数据降至3维; 最终, 运行模糊C-均值聚类算法(FCM)得到较优初始聚类中心, 使用GKIPCM算法对四类降维后的光谱数据进行聚类分析, 并与GK聚类算法与IPCM聚类算法的运行结果作对比。 GKIPCM算法的总迭代时长为0.218 8 s, 分类准确率达到了97.22%。 相较之下, GK算法与IPCM算法的准确率分别为63.89%和91.67%, 运行的总时长为0.093 8与0.062 5 s。 从实验结果可看出, GKIPCM算法可以通过分析光谱数据从而完成对不同程度农药残留进行定性分析的任务。
Infrared spectroscopy is a technology used to identify the chemical composition of substances based on molecular vibration and quantum-jump theory. Due to the unique absorbance of different functional groups, the spectral data related to the absorbance and the wavelength (or wavenumber) can be obtained when the infrared beam irradiates the molecular. However, the spectral data from experiments always have high dimensions and overlap, making it difficult to process the data. Thus, this paper proposed an improved Gustafson-Kessel possibilistic c-means clustering (GKIPCM), introducing the Mahalanobis distance from GK clustering and the iterative equations of fuzzy membership values and cluster centers from improved possibilistic c-means clustering (IPCM). GKIPCM makes the data adapt to different mathematical distance measures and avoids identical cluster centers. Furthermore, GKIPCM has higher classification accuracy, which is less sensitive to parameters. In the experiments, four groups of washed Chinese cabbage were the objects of spectral analysis and different concentrations of lambda-cyhalothrin pesticide were sprayed on the Chinese cabbages. Spectral data of Chinese cabbages were collected with Agilent Cary 630 FTIR spectrometer. Firstly, multiplicative scatter correction (MSC) was applied to reduce the noise and eliminate data offset when pre-processing the data. Secondly, principal component analysis (PCA) was utilized to reduce dimensions due to the wide wavenumber range (4 300~590 cm-1) and the high data dimensions (971). After conducting PAC, the dimensionality of data was reduced to 23, and the total contribution of 23 principal components reached 99.60%. Nonetheless, the feature information was still mixed. So the linear discriminant analysis (LDA) was used to extract features of the spectral data, and the LDA algorithm reduced the dimensionality of the spectral data to 3. Finally, the fuzzy c-means clustering (FCM) was employed to obtain the optimal initial cluster centers. Then, the GKIPCM algorithm was applied to cluster four different groups of spectral data. Comparisons were made among the clustering results of GKIPCM, GK and IPCM. The running time and accuracy of GKIPCM were 0.218 8 seconds and 97.22%, and those of GK and IPCM were 0.093 8 seconds and 63.89%, 0.062 5 seconds and 91.67%, respectively. According to the results of the experiments, the GKIPCM algorithm finished the qualitative analysis of different concentrations of pesticide residues by analyzing the spectral data.
民以食为天, 食以安为先。 我国自古以来就是一个人口数量庞大的农业大国, 对于食品质量的重视程度从未放松。 为了保证粮食的产量与营养价值, 往往需要对作物定期喷洒定量的农药, 以消灭害虫。 然而, 农药的残留对人类健康与自然环境却有所危害[1, 2, 3]。 当农作物流入市场时, 所施加的农药总会或多或少地残留在农作物的表面, 其浓度由于初始浓度, 喷洒周期, 贮存时间等因素而各不相同。 我国有严格的进口食品安全管理体制和进口蔬菜农药标准, 故准确有效的蔬菜农药残留检测分类与国计民生息息相关, 具有重要的研究价值。
国内外学者在农药残留检测方面做了很多的研究, 较为常见的方法有传统的化学检测与红外光谱分析等。 红外光谱分析的独特优势, 推动了农药残留检测方法的进一步发展, 实现了快速无损化; Jiang等借助气相色谱法, 使用近红外高光谱成像系统预测桑叶中农药残留的分布[4]; Zhou等采用偏振光谱检测技术收集偏振光信息, 使用BP神经网络, K近邻和支持向量机建立分类模型, 最后得到不同农药残留的校准识别率为100%, 预测识别率为97.78%[5]; Sun等提出了一种结合化学分子结构和小波变换的方法提取特征波长, 通过提取的特征光谱数据建立支持向量机模型, 校准与预测精度达到100%[6]; Wu等提出深简网络和近红外透射光谱相结合的检测方法, 通过支持向量机、 PLS-DA和K近邻法建立分类模型, 训练与测试的准确率分别达到了98.89%和95.00%[7]; Sun等基于生菜叶片的高光谱数据, 将竞争性自适应重加权采样与连续投影算法应用于最小二乘支持向量回归模型, 对生菜叶片中混合农药残留进行定量检测[8]。
聚类分析是一种无监督的机器学习算法, 广泛应用于模式识别领域[9], 例如K均值聚类算法[10]。 模糊聚类分析将模糊理论[11]与聚类算法结合, 每一个数据样本可同时隶属于多个聚类, 以数据特征相似性度量来识别类群。 例如: 为了解决模糊C-均值聚类算法(fuzzy c-means clustering, FCM)[12]对噪声环境的敏感问题, 引入可能性的概念代替模糊隶属度, 修改目标函数约束条件的可能C均值聚类算法[13]; 基于模糊马氏距离的自适应改进可能C均值聚类算法[14]; 使用马氏距离测度预测非椭球形数据的GK(gustafson-kessel clustering)算法[15]; Wu等提出的结合模糊协方差矩阵的可能性模糊C均值聚类算法[16]。 为了得到更适合含农药残留的白菜的中红外光谱聚类分析算法, 并在已有的算法上进一步提高分类的准确率且保持算法的优良特性, 本文结合GK算法与改进的可能C均值聚类算法(improved possibilistic c-means clustering, IPCM), 提出一种GK可能C均值聚类算法(gustafson-kesselimproved possibilistic c-means clustering, GKIPCM)。
首先使用中红外光谱仪采集白菜的中红外光谱数据, 其次依次使用多元散射矫正, 主成分分析, 线性判别分析对数据进行预处理, 然后应用GKIPCM对数据进行聚类分析, 最后对比GK聚类与IPCM聚类的分类准确率得出结论: GKIPCM算法可以完成对不同浓度农药残留的定性分析。
1.1.1 材料
本实验对象为采购于菜市场的新鲜小白菜, 将其以45 ℃温水洗净后作为施以农药的原材料。 选取高效氯氟氰菊酯为实验农药, 使用喷雾方式确保农药喷洒均匀。 将实验原材料分为四组, 第一组不喷洒农药, 作为对照实验组; 第二、 三、 四组的农药配比分别为1:500, 1:100, 1:20。 阴干后将白菜分解成小块, 便于进行中红外光谱数据的收集。
1.1.2 光谱仪器与分析软件
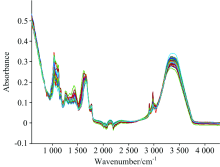
仪器选用安捷伦Cary 630 FTIR光谱仪, 调至ATR衰减全反射光谱采集模式。 扫描背景与样本各64次, 分辨率调整为8 cm-1。 配合Microlab PC、 Resolutions Pro软件, 每组样品采集40组数据, 共计160组, 每组数据有971维, 波数变化范围为4 300~590 cm-1。 白菜傅里叶中红外光谱(Fourier transform mid-infrared spectroscopy, FT-MIR)如图1所示, 采用Matlab R2016b对该光谱数据进行定性分析。
(1)参数初始化
设置权重指数m(m> 1), 测试样本的维数d, 循环迭代次数的初始值r=0, 最大允许迭代次数rmax=100, 迭代的最大误差参数ε =0.000 01; 运行FCM算法并将最终模糊隶属度与类中心作为GKIPCM算法的初始模糊隶属度
(2)计算第r次迭代时的模糊散射矩阵Sfi, r
式(1)中, uik, r-1为第r-1次迭代后第k个测试样本对第i类的模糊隶属度; n为样本总量; xk为第k个白菜中红外光谱测试样本; vi, r-1为第r-1次迭代后第i类的类中心(i=1, 2, 3, 4); Sfi, r是第r次迭代后第i类样本的模糊散射矩阵。
(3)计算第r次迭代后第i个聚类中心的范数矩阵Ai, r
(4)由式(3)计算第r次迭代后测试样本xk到类中心vi, r-1的距离范数Dik, r
(5)计算第r次迭代后第k个测试样本隶属于第i类的典型值tik, r
(6)计算第r次迭代后的模糊隶属度矩阵Ur, 其各元素值计算公式为:
式(5)中, c为样本类别数。
(7)计算第r次迭代后第i类的类中心vi, r
(8)计算相邻两次迭代后两聚类中心的距离范数Dr
(9)对已计算数据进行如下判断:
若Dr< ε 或r≥ rmax, 则停止运行, 记录迭代结束时模糊聚类中心Vend, 模糊隶属度矩阵Uend以及典型值矩阵Tend; 否则令vi, r-1=vi, r, uik, r-1=uik, r, 回到步骤(2)继续迭代计算。
(10)根据模糊隶属度矩阵与典型值矩阵对样本进行分类:
对于模糊隶属度, 若uij为uj中的最大值, 则判断第j个样本属于第i类; 对于典型值, 若tij为tj中的最大值, 则认为第j个样本有更大的可能性属于第i类。
将样本数据分为训练集与测试集。 其中训练集共4类, 每类22个样本; 测试集共4类, 每类18个样本。 在光谱数据采集的过程中, 散射水平的差异往往造成白菜表面实际农药的残留量与光谱波长的数据相关性的下降。 为了减小此类差异且提高信噪比, 首先使用多元散射矫正(multiplicative scattering correction, MSC)对光谱数据进行预处理。 图2为使用多元散射矫正后傅里叶中红外光谱数据图。
为观察不同农药残留程度的光谱数据的吸光度有所差异, 计算了每一类光谱数据的平均值并截取了波数介于800与1 500之间的部分如图3所示。
不同农药浓度样品的中红外光谱吸光度有着明显差值, 直接保证了不同类光谱数据的可分性。
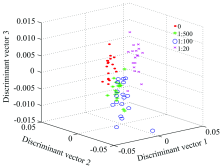
尽管MSC将基线平移并且消除了偏移量, 但数据依然达到了971维。 为减小运算量, 采用主成分分析(principal component analysis, PCA)对数据实现降维并提高分类准确率。 通过训练数据集的协方差矩阵得到前23个主成分, 累计贡献率高达99.60%。 故通过该23个主成分将光谱数据缩小至23维。 为了提取样本数据的相关特征信息并进一步降维, 使用线性判别分析(linear discriminant analysis, LDA)处理训练样本。 此处求取了3个判别向量与特征值。 其中特征值的计算结果为: λ 1=7.371 3, λ 2=3.932 0, λ 3=1.931 7。 将PCA处理后的训练数据投影到判别向量构成的特征空间, 得到线性判别分析得分图, 见图4。
图中, “ · ” , “ * ” , “ ○” , “ × ” 分别表示农药配比为无农药, 1:500, 1:100, 1:20的光谱数据。 无农药的样本与配比为1:20的样本区分度最高, 相比之下农药配比为1:100和1:500的两类样本区分度稍弱。 尽管如此, 四类样本整体的区分度较强, 说明线性判别分析的特征提取结果是可取的。 于是将PCA的23组线性变换(主成分)与LDA的3个鉴别向量应用于测试集, 为模糊聚类做准备。
2.2.1 模糊聚类相关参数的初始化
设置权重指数m=2.0(m≥ 1), 测试样本的维数d=3; 由于FCM, GK, IPCM, GKICPM算法都属于迭代算法, 设置迭代次数初始值r=1, 最大迭代次数rmax=100; 当迭代误差小于ε =0.000 01或r≥ rmax时停止迭代。 FCM的初始聚类中心如式(8)
对于GK, IPCM, GKIPCM, 将FCM的最终聚类中心与模糊隶属度作为算法的初始值。
2.2.2 模糊聚类算法的迭代时长
运行GK, IPCM, GKIPCM, 对比迭代次数与收敛时间: GK迭代了100次, 运行时间为0.093 8 s; IPCM迭代了13次, 运行时间为0.062 5 s; GKIPCM迭代了87次, 运行时间为0.218 8 s。 处理器: Inter(R) Core(TM) i5-8300H CPU @2.30GHz(8 CPUs); 内存: 8192MB RAM。
2.2.3 模糊隶属度分类结果
参照2.2.1将各参数初始化后, 分别运行GK, IPCM与GKIPCM。 得到GK, IPCM, GKIPCM对应的模糊隶属度分别为uik, GK, uik, IPCM, uik, GKIPCM, 表示第k个测试样本对第i类的模糊隶属度; 同时, IPCM与GKIPCM还具有典型值tik, IPCM, tik, GKIPCM, 表示第k个测试样本属于第i类的可能性, 其中GKIPCM算法的典型值分布如图5所示。 典型值取消了测试样本xk对各类的隶属度之和为1的约束条件, 减弱了聚类过程中噪声对分类的影响。
对于模糊隶属度uik与典型值tik, 若uik为uk中的最大值或tik为tk中的最大值则认为第k个测试样本可以被划分到第i个类别当中。 最终根据模糊隶属度与典型值得到GK, IPCM, GKIPCM的分类精度分别为: 63.89%, 91.67%, 97.22%。
2.3.1 权重指数m
在模糊聚类算法中, 权重指数m对算法的准确程度有着重要影响。 对于GKIPCM, 当m≥ 2时, 模糊隶属度矩阵在迭代过程中有着较好的收敛性; 但由于同一样本对不同类的典型值没有和为1这一约束条件, m过大时式(5)的计算结果会受到干扰, 甚至使下一次迭代时式(1)计算的模糊散射矩阵Sfi, r接近奇异值, 影响分类准确度。 此处固定主成分个数为23, 研究权重指数m在区间[2, 6]上时, 各算法的聚类准确率, 结果如表1所示。
| 表1 不同参数m的GK, GKIPCM, IPCM算法聚类准确率 Table 1 Clustering accuracies of GK, GKIPCM, IPCM with different values of m |
由表1可见, m在有限范围内, GKIPCM聚类准确率高于GK聚类与IPCM聚类。
2.3.2 主成分个数
在PCA降维过程中, 通常使得主成分的累积贡献率达到一个较高的百分比(80%~90%), 否则样本数据维度降低的同时也会损失大量的特征鉴别信息。 此处固定m=2, 通过调整主成分的个数从而调整累计贡献率, 观察对聚类准确率的影响, 结果如表2所示。
| 表2 不同主成分的个数下GK, GKIPCM, IPCM算法的准确率 Table 2 Clustering accuracies of GK, GKIPCM, IPCM with different numbers of principal components |
由表2知: 随着主成分个数的增加, GK, GKIPCM, IPCM的准确率都呈现上升趋势, GK的准确率相较其他算法对参数敏感一些, GKIPCM聚类的准确率在主成分个数为23时高达到97.22%。 综合表1、 表2, 该GKIPCM算法适合处理农药残留程度的定性分析问题。
将GK的马氏距离测度与IPCM的模糊隶属度和聚类中心更新方程结合, 提出了一种新型GKIPCM算法。 实验证明, 对农药残留的白菜傅里叶中红外光谱数据采用GKIPCM算法进行分析, 准确率达到了97.22%(例如: m=2, 主成分个数等于23), 分类效果优于GK与IPCM聚类算法, 具有较高的实际应用价值。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|