作者简介: 赵 勇, 1978年生, 燕山大学电气工程学院副教授 e-mail: zhaoyong@ysu.edu.cn
肺炎支原体是造成人类呼吸系统疾病的主要原因。 临床中, 患者感染不同肺炎支原体症状极为相似, 很难根据症状判别肺炎支原体类型并对症给药。 因此, 准确判别肺炎支原体菌株类型对于发病机理和疾病流行病学研究以及临床精准治疗具有重要意义。 拉曼光谱具有快速、 高效、 无污染等优点, 在生物医学领域逐渐得到越来越多研究者们的关注。 一维卷积神经网络(1D-CNN)是一类包含卷积运算且具有深度结构的前反馈网络, 在语音信号和振动信号分析等方面取得成功应用。 提出一维卷积神经网络与拉曼光谱技术结合, 针对肺炎支原体主要基因型M129型和FH型样本的拉曼光谱数据集, 实现肺炎支原体菌株分类。 利用光谱数据增强方法扩充原光谱数据集作为模型输入, 训练一维卷积神经网络模型, 解决由于小样本导致卷积神经网络数据饥渴问题; 为了得到最好的肺炎支原体分类效果并加速学习过程, 优化模型结构并确定最佳模型参数; 拉曼光谱测量时常混有高斯噪声、 泊松噪声和乘性噪声, 为优化模型抗噪能力, 将原光谱分别叠加高斯噪声、 泊松噪声和乘性噪声, 训练一维卷积神经网络模型并和LDA, KNN和SVM等传统算法进行比较。 实验结果表明基于1D-CNN方法, 对于叠加高斯噪声的光谱数据所建模型分类正确率为98.0%, 叠加泊松噪声的光谱数据分类正确率为97.0%, 叠加乘性噪声的光谱数据分类正确率为97.0%, 分类正确率远高于基于LDA, KNN和SVM等传统算法所建模型分类正确率; 同时构造叠加5, 15, 25, 35, 45和55 dBW不同强度噪声的光谱数据集, 当噪声达到55 dBW时, 1D-CNN模型仍能取得92.5%的分类正确率。 因此, 一维卷积神经网络结合拉曼光谱技术应用于肺炎支原体菌株类型分类是可行的, 具有抗噪声能力强和分类正确率高的优点, 该研究为肺炎支原体肺炎快速诊断提供新思路。
Mycoplasma pneumoniae is the main cause of human respiratory diseases. Clinically, the symptoms of patients infected with different mycoplasma pneumoniae are very similar, so it is difficult to distinguish the type of mycoplasma pneumoniae according to the symptoms and give medication. Therefore, the accurate identification of mycoplasma pneumoniae strain type is of great significance for the pathogenesis and epidemiological research of the disease, and accurate clinical treatment. Raman spectrum has been paid more and more attention because of its advantages of fast-speed, high efficiency, pollution-free and non-destructive analysis. One-dimensional Convolution Neural Network (1D-CNN) is a kind of pre-feedback network with a deep structure, including Convolution operation. It has been successfully applied in the analysis of speech and vibration signals. The combination of the One-Dimensional Convolution Neural Network and the Raman spectral data of the main genotypes of mycoplasma pneumoniae M129 and FH were used as the research objects to realize mycoplasma classification pneumoniae strains. The spectral data enhancement method expands the original spectral data set, and the one-dimensional convolution neural network model was trained, and the problem of data hunger of convolutional neural network caused by small samples was solved. In order to obtain the best classification effect of mycoplasma pneumoniae and accelerate the learning process, the model structure was optimized, and the best model parameters were determined. Gaussian noise, Poisson noise and multiplicative noise are often mixed in Raman spectral measurement. Gaussian noise, Poisson noise and multiplicative noise are often mixed in Raman spectral measurement. In order to optimize the anti-noise ability of the model, Gaussian noise, Poisson noise and multiplicative noise were superimposed on the original spectrum respectively, and the 1D-CNN model was trained and compared with the models built by traditional algorithms such as LDA, KNN and SVM. The experimental results show that for the Raman spectra superimposed with Gaussian noise, Poisson noise and multiplicative noise, the classification accuracy of the models based on 1D-CNN method has achieved 98.0%, 97.0% and 97.0%, respectively, which are all much higher than those of the models based on LDA, KNN and SVM algorithms. At the same time, the 1D-CNN model can achieve 92.5% classification accuracy when the noise reacheds the 55 dBW interference factor, aiming at the noise with different intensities of 5, 15, 25, 35, 45 and 55 dBW.Therefore, it is feasible to apply a one-dimensional convolutional neural network combined with Raman spectrum technology to the classification of mycoplasma pneumoniae strain types, which has the advantages of strong anti-noise ability and high classification accuracy. This study provides a new idea for the rapid diagnosis of mycoplasma pneumoniae pneumonia.
肺炎支原体(mycoplasma pneumoniae, MP)感染是社区获得性肺炎最常见的病因之一[1]。 它是引起人类呼吸道感染的常见病原体, 如支气管炎、 肺炎等, 严重可诱发哮喘等疾病[2]。 目前, 肺炎支原体菌株传统实验室诊断方法主要包括培养法、 血清学法和聚合酶链反应(polymerase chain reaction, PCR)分析[3]。 然而培养实验的培养周期长, 出结果慢; 血清学分析使用抗体作为生物标志物, 缺乏对疾病发作的敏感性[1]; PCR技术具有较高灵敏度和特异性, 但需要熟练的操作人员、 昂贵的仪器和复杂的样品预处理, 不能广泛应用于早期即时检测[4]。 目前各种检测方法都存在耗时长, 对实验条件、 环境和人员要求高, 培养过程中易受细菌和真菌干扰, 诊断敏感性和特异性低等问题, 限制了其在临床上的广泛应用[5, 6]。 因此, 快速、 灵敏、 特异的肺炎支原体菌株检测方法研究具有重要临床意义。
拉曼光谱是一种非弹性散射的电磁辐射, 是分子振动和辐射之间能量交换的结果。 拉曼光谱技术具有所需样品少、 无需复杂预处理、 不破坏样品、 检测速度快且灵敏度高等特点, 广泛应用于乙肝[7]、 肺癌、 胃癌、 肾病[8]等疾病的诊断。 感染肺炎支原体的患者血液分子结构发生变化并反映在其拉曼光谱中, 为基于拉曼光谱法判断肺炎支原体菌株类型提供理论依据。
随着深度学习在图像处理、 语音识别等许多领域都取得成功应用, 卷积神经网络获得广泛关注和极大发展, 但目前应用于疾病诊断领域的光谱识别算法大多采用传统机器学习方法。 相对于传统拉曼光谱分类算法而言, 深度学习方法可以省去特征提取环节, 简化光谱分类过程, 提高识别准确率。 一维卷积神经网络可以从包含各种特征的光谱中提取与目标分析物相关的信息。 Liu等[9]采用包括特征提取的金字塔形卷积层和用于分类的2个全连接层的LetNet变体的深度卷积神经网络对拉曼光谱数据分类的方法, 在RRUFF矿物拉曼光谱数据库上取得很好的分类效果。 Shao等[10]使用2个卷积和1个全连接的卷积神经网络结合拉曼数据筛选前列腺癌骨转移的能力, 使用五倍交叉验证方法对模型进行训练和测试, 模型平均检测正确率81.70%。 李庆旭等[11]将可见-近红外透射光谱技术与3个卷积层和1个全连接层的卷积神经网络相结合, 用于入孵前种鸭蛋受精信息的无损鉴别, 测试集分类正确率97.41%, 高于逻辑回归、 SVM等传统方法。
本文提出将一维卷积神经网络(one-dimensional convolution neural network, 1D-CNN)模型应用到肺炎支原体菌株拉曼光谱识别问题, 优化卷积核大小和数目等模型参数, 针对M129型和FH型两类肺炎支原体光谱, 模拟高斯噪声、 泊松噪声和乘性噪声等测量拉曼光谱数据时的常见噪声, 验证模型抗噪能力, 通过和传统机器学习算法所建模型分类结果进行比较, 证明所提出方法的有效性, 为肺炎支原体肺炎快速诊断提供一个新的思路。
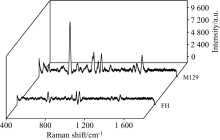
本文研究的肺炎支原体菌株拉曼光谱数据来源于Dryad光谱数据集(https://doi.org/10.5061/dryad.s5h20)[12], 具体选择两种主要的肺炎支原体菌株基因型M129型和FH型菌株的各25条拉曼光谱作为基础光谱样本模板。 原始数据库中采集两类菌株拉曼光谱时, 激光器光源功率为28 mW, 积分时间为10 s, 光谱采集范围400~1 800 cm-1, 两类肺炎支原体菌株的基线校正后的平均拉曼光谱图如图1所示。
卷积神经网络的建立通常要求基于大规模数据库作为训练数据, 充足的训练数据可以使神经网络模型充分学习到数据类别内部特征和类别间的区别, 增强模型的鲁棒性, 尽可能避免过拟合现象。 然而在实际应用领域中, 由于临床样本的限制往往无法获得足够的拉曼光谱来训练深度学习模型。
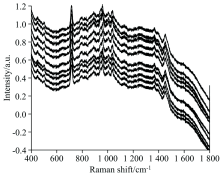
数据增强是一种从有限的标记样本中扩大样本数量来训练神经网络, 从而提高模型鲁棒性的技术。 对于光谱数据, 本文通过给拉曼光谱加入随机基线偏移量, 设定样本光谱的不同基线斜率和随机乘性扩大光谱幅值的方法进行光谱数据增强。 随机偏移量设定为样本± 0.10倍光谱标准差; 在0.95~1.05之间随机设定基线的斜率; 按照样本光谱的1± 0.10倍标准差进行幅值乘性扩大。 一个光谱样本进行10倍增强后的光谱如图2所示。
实际光谱数据采集过程中, 由于受传感器材料属性、 工作环境、 电子元器件和结构等影响, 会引入各种噪声, 如电阻引起的热噪声、 光子噪声、 暗电流噪声、 光响应的非均匀性、 环境噪声等。 噪声以无用的信息形式出现, 扰乱光谱的可观测信息, 呈现与物质不相连的谱峰。 传统算法很难完全去除噪声影响, 因此模型的抗噪性能显得尤为重要。
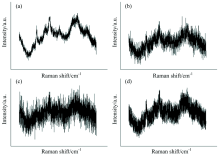
高斯噪声是最主要的随机噪声类型, 主要是由于传感器亮度不均匀, 长期工作温度过高引起; 由于光具有量子特效, 到达检测器表面的量子数目存在统计涨落, 因此对光谱细节信息遮盖, 这种由于光量子而造成的测量不确定性称为泊松噪声; 乘性噪声往往由于信道不理想引起, 噪声部分随信号变化而变化, 且与信号是相乘关系。 根据以上对噪声的分析, 本文在M129型和FH型肺炎支原体菌株的原始光谱中分别叠加均值为0, 方差为0.000 1的高斯噪声、 泊松噪声以及方差为0.002的乘性噪声, 叠加噪声后的光谱如图3所示。
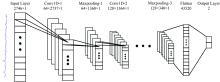
根据光谱数据特点, 本文构造1D-CNN模型进行光谱分类。 整个1D-CNN包含3个卷积层和3个池化层, 其网络结构如图4所示。
光谱数据以2 746× 1的矩阵形式输入卷积层, 在卷积运算层中, 使用线性修正单元变体LeakyReLU作为激活函数, 表示式如式(1)所示
为了降低运算复杂度, 卷积神经网络每层内的神经元权值共享, 通过大量仿真优化比较, 卷积核尺寸参数分别设定为10× 1, 5× 1和2× 1, 卷积核个数分别设定为64, 64和128。 卷积可提取前一层信息的不同特征, 这些不同特征共同作为下一层网络的输入数据。 一维信号卷积运算公式如式(2)所示
式(2)中, * 表示卷积运算, yj为第j个输出特征图, xj为第j个输入特征图, kij为本层卷积运算所使用的卷积核, bj为第j个特征图的偏置。
每个卷积运算层后对应一个池化层, 对卷积运算生成的特征图采样, 池化层运算并未减少特征图个数, 而是减小每个特征图的维度, 缩减数据量, 提升运算速度, 本文采用最大池化(Max-pooling)对信号进行降采样, 其表达式如式(3)所示。
式(3)是最大池化对信号降采样计算方法, 对卷积层运算所得的一个特征映射将其划分多个不重叠Xk, k=1, 2, 3, ..., K, 区域大小为s。
卷积及池化层获取光谱数据特征后, 将其展开并输入全连接层进行分类, 使用tanh激活函数, 并在全连接层后加入应用比例为0.5的随机失活层, 避免过拟合, 加快收敛速度, 提高神经网络鲁棒性。 设置批处理样本数目参数为90, 采用交叉熵损失函数, 计算公式如式(4)所示
式(4)中, xn是训练光谱数据, yn, k是第n个样本预测第k个数据的标签, pn, k是第n个样本预测第k个标签值的概率, N是总共的样本数, k为总标签类数。
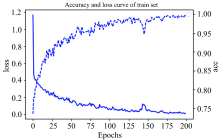
原始光谱数据经过增强和三类噪声叠加构成建模所需的光谱数据集, 将数据集随机划分为3部分: 70%光谱数据作为训练集, 10%光谱数据作为验证集, 用于在反向传播训练过程中调整神经元权重参数; 20%光谱数据作为测试集, 用于测试已训练后的网络模型性能。 模型经过200个epoch训练之后的正确率和损失值曲线如图5所示, 可以看出网络基本收敛。
本文提出的1D-CNN方法与LDA, KNN和SVM三种传统方法进行比较, 结果如表1所示。 从表1中可以看出, CNN对加入高斯噪声的M129和FH肺炎支原体菌株光谱数据所建模型分类正确率为98%, 泊松噪声的M129和FH肺炎支原体菌株光谱数据所建模型的分类正确率为97%, 乘性噪声的M129和FH肺炎支原体光谱菌株数据所建模型的分类正确率为97%, 均高于传统算法所建模型的分类正确率。
| 表1 不同算法分类结果对比 Table 1 Comparison of classification results with different algorithms |
为了进一步比较1D-CNN模型和传统算法对不同种类噪声的抗噪能力, 分别得到KNN, SVM, LDA和CNN模型的混淆矩阵, 如图6所示。 从图6中可以看出, 基于1D-CNN方法所建模型的误判个数最少, 同时1D-CNN模型对肺炎支原体菌株类型的灵敏度和特异性均高于其他算法所建模型, 结果表明1D-CNN模型相比传统算法模型在抗噪声方面具有明显优势。
对于肺炎支原体菌株类型的定性分析, ROC曲线下面积AUC(area under the curve)与准确率呈正相关, AUC值越大, 模型准确率越高。 不同算法针对不同种类噪声的ROC曲线对比结果如图7所示。 由图7(a)可知, 加入高斯噪声后, CNN模型的AUC值为0.982, 传统分类算法LDA, KNN和SVM模型的AUC值分别为0.612, 0.624和0.658; 由图7(b)和(c)可知, 加入泊松噪声和乘性噪声后, CNN模型的AUC值也均高于其他算法。
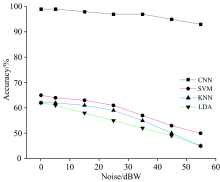
噪声强度对拉曼光谱定性分析模型提出更高要求, 因此在肺炎支原体菌株原始拉曼光谱中分别叠加5, 15, 25, 35, 45和55 dBW六种不同强度的高斯白噪声, 以测试模型的抗噪声性能, 四种算法在不同噪声强度下的分类结果对比如图8所示。
 | 图8 不同算法针对不同强度噪声分类结果Fig.8 Classification results with different algorithms for different intensity of noise |
从图8可以看出, 随着噪声强度的不断增大, LDA, KNN和SVM算法所建模型分类正确率下降幅度较大, 而1D-CNN 方法所建模型的分类正确率变化幅度较小, 当添加噪声强度达到55 dBW时, 1D-CNN模型依然能够提取到拉曼光谱特征, 获得92.5%的分类正确率。 因此, 1D-CNN 所建模型抗噪性能远优于传统算法所建模型性能。
为实现肺炎支原体菌株类型的准确分类, 提出1D-CNN拉曼光谱分类方法。 针对小样本肺炎支原体菌株的拉曼光谱, 提出适用于拉曼光谱数据的数据增强方法, 扩充光谱数据以满足建模样本需求。 同时模拟光谱采集时不同种类噪声影响, 验证模型的抗噪能力。 结果表明利用拉曼光谱结合1D-CNN, 无需光谱预处理可以有效筛选信息, 同时能够更好地挖掘出光谱特征, 从而减少计算量和缩短计算时间。 相比传统算法能得到更高的分类正确率, 并具有很好地抗噪声能力, 具有明显的优势和重要的实际应用价值。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|