作者简介: 冀荣华, 1973年生, 中国农业大学信息与电气工程学院副教授 e-mail: jessic1212@cau.edu.cn
基于光谱的土壤氮含量预测模型泛化能力弱是制约其推广应用的瓶颈。 鉴于特征提取及非线性表达能力方面的优势, 深度学习模型具有较强的泛化能力。 提出一种融合自动编码器和卷积神经网络(Encoder-CNN)的土壤氮含量光谱预测模型, 探索模型结构和参数对模型性能的影响。 根据以往研究成果和相关性分析, 获得180个与氮含量强相关的波长, 将其作为Encoder-CNN模型输入, 而将土壤氮含量作为模型输出。 Encoder-CNN模型利用自动编码器的编码部分进行光谱数据降维, 然后输入到卷积神经网络进行土壤氮含量预测。 设计2种网络结构, 每种网络结构包含2种不同参数设置, 共4个模型, 用以探索Encoder-CNN土壤氮含量光谱预测模型结构和参数对模型性能的影响。 利用公开数据集LUCAS对模型进行训练。 按3σ原则对公开数据集LUCAS进行异常值检测与处理, 获得20 791个数据, 其中18 711个样本作为训练集, 2 080个样本作为测试集, 对Encoder-CNN模型进行训练。 结果表明: 对于自动编码器, 在相同隐含层数下, 最后的隐含层神经元个数为30时, 复现效果最优。 增加隐含层数, 会提升复现效果。 增加卷积核数量, 特别是尺寸为1×1卷积核, 能够提高模型的预测性能与可靠性。 增加池化层的网络结构, 模型预测精度提升至0.90以上。 增加全连接层神经元数量也会提升模型性能。 利用自采集的黑龙江黑土实时光谱数据集进行模型迁移, 观察模型泛化能力。 当模型迭代100次后, 在黑龙江数据集上的预测精度即可达到0.90以上; 当迭代次数为900时, 模型在训练集和测试集上的预测精度可以达到0.98。 结果表明, 所构建的Encoder-CNN土壤氮含量光谱预测模型具有较好的泛化能力。
Poor generalization ability of soil nitrogen content prediction models based on spectroscopy is the bottleneck of its actual application in agriculture production. However, the deep learning model shows strong potential for generalization because of its automatic feature extraction and excellent nonlinear expression. In this paper, a spectral prediction model of soil nitrogen content combining the auto-encoder and convolutional neural network (Encoder-CNN) was proposed, the influence of model structure and parameters on model performance was explored, and its generalization ability was investigated. After researching the previous references and analyzing the correlation between wavelengths and soil nitrogen content, 180 wavelengths with the highest correlation were selected and taken as the input of the Encoder-CNN model. The output of the Encoder-CNN model was the soil nitrogen content. The Encoder-CNN model first used the auto-encoder to reduce the dimension of 180 wavelengths and then predicted the soil nitrogen content by its convolutional neural network. Two network structures were designed. Each network structure had two different parameter settings. Therefore, four models were used to explore the effects of structure and parameters of the Encoder-CNN soil nitrogen content spectral prediction model on modeling performance. Encoder CNN models were trained by the LUCAS data set. According to the 3 σ principle, 20 791 data were obtained from LUCAS and then divided into a training set (18 711) and test set (2 080). The results were analyzed and discussed, and several conclusions were achieved in this research. The reproduction effect of the automatic encoder reached the best when the number of neurons in the last hidden layer was set to 30 with the same number of hidden layers as the others; the more hidden layers, the better the reproduction effect. As for the prediction part based on CNN, increasing the number of convolution kernels, especially 1×1 convolution kernels, could improve the prediction performance and reliability. By adding pooling layer in CNN, the model's prediction accuracy was improved to more than 0.90. The model's performance could also be improved by increasing the number of neurons in the full junction layer. The Encoder-CNN model built by the LUCAS data set was migrated to the Heilongjiang data set, and the generalization ability of the model was observed and evaluated. The prediction accuracy of the model, which was trained 100 times by the Heilongjiang data set, could reach more than 0.90. When the number of iterations was set to 900, the model's prediction accuracy could be as high as 0.98. The results showed that the proposed Encoder-CNN spectral prediction model of soil nitrogen content had good generalization ability, and it could be used to detect soil nitrogen content after the model migration process.
精准获取土壤中的氮含量是实施各类农田水肥管理技术的基础。 传统土壤氮含量化学测定方法, 很难客观、 全面地反映农田土壤养分含量实际分布状况。
利用光谱分析技术能够快速、 高效检测土壤氮含量[1]。 基于光谱的土壤氮含量预测相关研究主要集中在土壤光谱预处理、 特征波长选取和预测模型构建三个方面。 研究表明, 对光谱数据进行预处理可使模型精度得到显著提高[3]。 周鹏等[4]提出利用灰度关联方法进行特征提取, 提高土壤氮含量预测精度。 Marcelo de Souza[5]针对多类型土壤构建多元回归模型进行土壤有机碳测定。 Li等[6]发现LS-SVM和PLSR模型具有一定稳定性。 Xu等[7]利用不同数据集建立土壤氮含量光谱预测模型, 发现模型泛化能力有待提高。 利用传统方法构建的预测模型泛化能力较弱, 原因在于数据量有限, 且模型非线性表达能力较弱。 深度学习在特征自动提取和优秀的非线性表达方面的优势, 使研究人员开始探索将深度学习算法应用于土壤养分预测[8]。 有研究设计五种深度不同的CNN, 发现7个卷积层的CNN网络对土壤有机碳的预测能力最强。 Zhang等[9]利用端到端深度学习方法进行土壤养分含量预测, 发现模型可以从原始数据中学习到更为有效的特征。 Ng等[10]讨论训练样本大小对深度学习模型精度影响。 Tsakiridis等[11]建立一维卷积神经网络(CNN), 引入自适应纠错机制改进模型结构, 提高模型预测精度。 Wang等[12]利用公共土壤光谱数据集(LUCAS)通过对比分析发现深度学习方法比传统的机器学习方法更有效、 实用。
深度学习模型在特征自动提取和非线性表达方面的优势使其在土壤氮含量预测性能方面表现出色。 但针对模型泛化能力方面的相关研究还有待加强。 本工作通过融合多种深度学习模型, 从模型结构设计、 参数设置方面开展研究, 提高模型泛化能力。
首先利用公开数据集构建土壤氮含量光谱预测模型, 再利用自采集数据集对模型迁移修订。 其中公开数据集来自欧盟范围内开展的大型土壤数据集采集项目— — 土地利用及覆盖面积框架调查(land use and cover area frame survey, LUCAS)。 LUCAS在2020年11月公布采自28个欧盟成员国的21 782个表土样本(0~20 cm)的吸光光谱数据。 样本采自农田、 林地、 灌木地、 草地和荒地等地, 涵盖灰化土、 棕壤、 荒漠土、 草炭土和栗钙土等欧洲主要土壤类型。 土壤样品经过40 ℃风干、 去除杂质、 研磨和过筛(孔径< 2 mm)处理后, 利用FOSS XDS光谱分析仪对其正反向扫描各一次, 取两次扫描结果平均值作为样本的光谱数据。 光谱波长范围400~2 500 nm, 间隔0.5 nm, 共4 200个波长。 采用改进的凯氏定氮法测定样品氮含量, 测定方法参见国际标准ISO 11261— 1995。 自采集数据集在中国黑龙江省胜利农场进行土壤样本采集及其吸光光谱和氮含量测定。 胜利农场位于东经133° 45', 北纬47° 24', 占地45万亩, 土壤类型为草炭土和黑土。 在农场随机选取300个土壤采样点, 用方形土壤采样器进行土壤样本采集。 在每个土壤采样点垂直剖面深度为5, 10, 15和20 cm的位置处分别取2 cm厚度土壤样品, 并混合装入一个取样袋, 作为该采样点处的土壤样品。 采用密闭避光包装, 标记, 带回实验室。 将土样烘干研磨后进行20目过筛处理, 利用自动定氮仪测定含氮量, 测定方法参见农业部标准NY/T1121.24— 2012。 使用傅里叶变换近红外光谱分析仪(FTS, MATRIX_I型, 布鲁克公司, 德国)测定光谱。 光谱测量范围为834~2 503 nm, 间隔0.5~4.8 nm, 每个样本光谱测量3次, 每次扫描30 s, 取平均值作为最终结果。 每个土壤样本测得1 037个波长吸光度光谱数据。
按3σ 原则对数据进行异常值检测与处理。 LUCAS数据集共20 791个数据样本, 黑龙江数据集共300个数据样本。 LUCAS数据集中氮含量范围为0~14.10 g· kg-1, 平均值2.39 g· kg-1; 黑龙江数据集的氮含量范围为1~27.43 g· kg-1, 平均值9.52 g· kg-1。
为消除量纲对模型的影响, 对光谱数据和氮含量进行归一化处理, 计算公式如式(1)所示
式(1)中, x和y分别为归一化前、 后数据值; min和max分别为样本对同一波长的吸光度的最小(大)值或氮含量的最小(大)值。
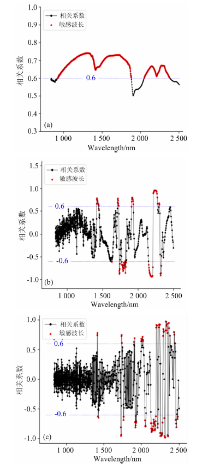
以特征波长为模型输入。 首先计算土壤样本原始光谱、 一阶微分光谱和二阶微分光谱与氮含量的相关系数。 图1(a)— (c)分别展示了自采集光谱数据相关分析结果。
选取相关系数绝对值大于0.6的波段为强相关波段, 按两数据集强相关波段的最大交集选出强相关波段, 统计结果如表1所示。
| 表1 光谱强相关波段 Table 1 List of strongly correlated wavebands for spectral data |
按照式(2)初步筛选出强相关波段作为敏感波段S。
式(2)中, A, A'和A″分别为利用原始光谱、 一阶微分光谱和二阶微分光谱筛选出的强相关波段。
按强相关波段和文献中强相关波段[3, 4]的最大交集筛选出四个波段作为特征波段, 选取180个波长(见表2)作为模型输入。
| 表2 特征波段及模型输入波长选择 Table 2 Characteristic bands and model input wavelength selection |
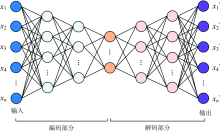
在保证模型精度的前提下, 降低模型复杂度, 利用自动编码器对特征波长进行降维处理。 自动编码器由编码和解码两部分组成, 结构如图2所示。
其中编码部分用于提取输入数据特征; 解码部分用于复现数据。
融合自动编码器和卷积神经网络优势, 提出基于Encoder-CNN的土壤氮含量光谱预测模型。 模型以特征波长为输入, 经过自动编码器进行波长降维, 将编码输出作为卷积神经网络的输入, 利用卷积神经网络进行土壤氮含量预测。 卷积神经网络由卷积层、 池化层和全连接层组成, 网络结构示意如图3所示。
网络中加入批量归一化用于约束数据分布, Dropout处理用于防止模型过拟合。
LUCAS数据集中, 训练集和测试集分别为18 711和2 080个样本。 设置训练批次为256, 迭代次数为80, 损失函数为均方误差, 激活函数为ReLU函数。 初始学习率为0.001, 每30 epoch(完整训练)下降为原来的1/10。 采用均方根误差(RMSE)、 决定系数(R2)和相对分析误差(RPD)评价模型性能, 计算公式如式(3)— 式(5)
其中, yi是真实值,
设计8种不同结构自动编码器用于土壤光谱数据降维。 自动编码器复现效果好表明编码输出能够有效表达输入, 复现效果如表3所示。
| 表3 不同自动编码器结构下光谱复现结果 Table 3 Spectral reconstruction effect of different automatic encoder structures |
研究中发现, 隐含层数越多复现效果越好。 选择AutoEnc7编码部分用于光谱数据降维。
设计两种网络结构, 每种网络结构设置两种不同参数, 共4个模型, 探讨网络结构、 参数对网络性能的影响, 设置如表4所示。
| 表4 卷积层参数设置 Table 4 The parameters setting of convolution layers |
模型利用相同数据集和参数(见1.5节)训练和测试, 结果如表5所示。
| 表5 四种模型在不同数据集上的预测结果(RMSE单位: g· kg-1) Table 5 The prediction performance of four models on different datasets(unit of RMSE: g· kg-1) |
可以看出, 针对网络结构1而言, 模型CNN-2预测性能、 拟合效果和可靠性均较模型CNN-1有所提升。 R2提高0.03, RMSE降低约0.1 g· kg-1, RPD提高约0.4。 依据CNN-2设置, 增加两个池化层形成网络结构2。 结构2模型预测精度均在0.90以上, 即增加池化层可高模型性能。 对比CNN-3和CNN-4, 发现增加全连接层神经元数量可改善模型性能。
利用自采集黑龙江黑土光谱数据集验证所建基于Encoder-CNN土壤氮含量光谱预测模型泛化能力, 结果如表5所示。 发现3个模型预测精度大于0.70, 即模型具有一定的泛化能力。 利用自采集数据集(270个样本作为训练集, 30个样本作为测试集)对模型CNN-3进行迁移学习。 迭代次数从100变化到1 000, 步长设为100, 结果分别如图4(a)— (c)所示。 观察图4(a)可以发现, 当模型迭代100次后, 预测精度可达到0.90以上。 当迭代900次模型的预测精度可以达到0.98, 其预测效果如图5所示。
 | 图4 模型CNN-3评价指标变化 (a): 决定系数; (b): 均方根误差; (c): 相对分析误差Fig.4 Changes of evaluation indexes of CNN-3 model (a): Coefficient of determination; (b): Root-mean-square error; (c): Relative percent deviation |
 | 图5 模型黑龙江数据集上的预测效果(迭代900次)Fig.5 The model prediction performance on Heilongjiang data set (900 iterations) |
结果表明, 基于Encoder-CNN的土壤氮含量光谱预测模型具有一定泛化能力。 模型通过小样本数据迁移学习, 即可适用于黑龙江黑土的氮含量预测, 精度较高。
快速检测土壤氮含量是农田水肥管理技术实施的重要基础。 提出一种融合自动编码器和卷积神经网络(Encoder-CNN)的土壤氮含量光谱预测模型。 探索网络结构及参数设置对模型性能的影响, 并利用自采集土壤光谱数据集对模型进行泛化能力验证。 结论如下:
(1)自动编码器实现光谱数据降维, 增加隐含层数会提升降维效果;
(2)网络结构和参数对基于Encoder-CNN土壤氮含量光谱预测模型性能影响较大。 1× 1卷积核个数、 池化层数和全连接层神经元个数等均可改变模型性能;
(3)利用样本丰富且数据量大的LUCAS数据集训练模型, 具有一定泛化能力。 在不改变网络结构, 仅需要少量样本, 迁移模型, 即可获得精度较高的模型。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|