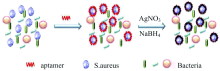
作者简介: 伏秋月, 1996年生, 广东医科大学医学技术学院硕士研究生 e-mail: fuqiuy@163.com
随着抗菌药物广泛应用于临床, 细菌耐药日益严重。 实现快速、 高灵敏、 准确的细菌及其药物敏感性检测是缓解细菌耐药的关键环节。 表面增强拉曼光谱(SERS)具有快速、 灵敏、 无损等优点, 可直接获取分子指纹信息, 它已成为一种有效的细菌及其耐药性检测技术。 不同种类细菌的分子组成和结构存在差异、 抗生素处理前后细菌的特征拉曼信号会发生变化, 这为表面增强拉曼光谱技术在致病菌及其耐药性检测中的应用提供了依据。 基于分子组成与结构的差异, 结合传统多分类数据分析以及机器学习算法, 表面增强拉曼光谱技术可以提供客观的诊断信息。 这篇综述回顾了近年来表面增强拉曼光谱技术对于致病菌及其耐药性检测的研究进展, 阐述了当前表面增强拉曼光谱技术应用于致病菌检测面临的问题。 首先, 讨论了致病菌及其耐药性检测中常用SERS基底的材料和结构: 金纳米粒子、 银纳米粒子、 银包金纳米粒子以及新型纳米材料与纳米粒子结合形成的复合SERS基底。 然后, 概述了SERS检测中捕获细菌的方法, 主要介绍了基于核酸适配体、 免疫磁性分离、 微流控系统以及静电结合的捕获方法, 包括上述捕获方法的原理以及捕获方式, 综述了以上捕获方法的研究进展。 最后, 总结了致病菌SERS光谱的各种数据分析方法, 通过光谱预处理, 特征提取与分类识别, 以及构建致病菌SERS光谱诊断模型, 实现致病菌及其耐药性的检测; 比较了传统的数据分析方法以及机器学习分析方法, 重点介绍了深度学习算法在致病菌及其耐药性SERS检测中的优势与应用。 文章也对表面增强拉曼光谱应用于致病菌及其耐药性检测的关键问题进行了讨论, 并对基于表面增强拉曼技术的致病菌及其耐药性检测方法进行了展望, 以促进表面增强拉曼光谱技术在临床检测中的应用。
With antimicrobial drugs widely used in the clinic, the drug resistance of pathogenic bacteria is becoming more and more serious. Rapid, highly sensitive, and accurate detection of bacteria and their drug susceptibility is the key to alleviating bacterial resistance. Surface-Enhanced Raman Scattering (SERS), can be used to obtain molecular fingerprint information directly and has become an effective detection technology for bacteria and their drug resistance. The molecular composition and structure of different species of bacteria are different, and the characteristic Raman signal of bacteria will also change before and after antibiotic treatment, which provides the basis for the application of SERS for the detection of pathogenic bacteria and their drug resistance. Based on the differences in molecular composition and structure, combined with traditional multi-class data analysis and machine learning algorithms, SERS can provide objective diagnostic information. In this review, the research progress of SERS for the detection of pathogenic bacteria and their drug resistance in recent years is reviewed, and the current problems existing in the application of SERS in the detection of pathogenic bacteria are also described. Firstly, the materials and structures of SERS substrates commonly used to detect pathogenic bacteria and their drug resistance were discussed, including gold nanoparticles, silver nanoparticles, silver-coated gold nanoparticles and composite SERS substrates formed by the combination of new nanomaterials and nanoparticles. Then, the methods of capturing bacteria in SERS detection were summarized, mainly based on nucleic acid aptamers, immunomagnetic separation, microfluidic systems and electrostatic binding. The principles and methods of the above methods were described, and the research progress of the above methods was summarized. Finally, various data analysis methods of SERS spectra of pathogenic bacteria were summarized. Through spectral preprocessing, feature extraction, classification and recognition, and the establishment of a diagnosis model of pathogenic bacteria, the detection of pathogenic bacteria and their drug resistance was realized. The traditional data analysis methods and machine learning methods were compared, and the advantages and applications of deep learning algorithms in the SERS detection of pathogenic bacteria and their drug resistance were introduced. In this paper, the key issues in the application of SERS in the detection of pathogenic bacteria and their drug resistance were also discussed, and the detection methods of pathogenic bacteria and their drug resistance based on SERS prospected, to promote the application of surface-enhanced Raman spectroscopy in clinical detection.
细菌耐药已经成为临床抗感染治疗中一个非常棘手的问题, 全球每年因细菌感染造成的死亡高达670万人, 消耗的治疗费用在数百亿美元以上[1]。 随着抗菌药物的大量使用, 这使得致病菌及其耐药性的快速检测变得迫切需要, 及时选择合适的抗生素治疗, 从而改善患者的预后, 同时避免不适当的治疗, 以及不必要的广谱抗生素的使用[2]。 基于传统培养方法的检测是临床检测中细菌以及耐药性鉴定的金标准。 传统的致病菌检测方法是将分离培养后的可疑菌落做涂片染色实验(革兰染色)、 生化反应实验、 溶血实验、 协同溶血实验、 动物实验及典型运动等鉴定方法, 例如利用显色培养基检测大肠杆菌。 这些方法费时费力, 而且一般一次实验只能检测单一细菌, 不能满足临床需要[3]。
为了克服传统检测方法的局限性, 人们对其进行了改进。 目前临床检验最广泛使用的方法有纸片扩散法、 梯度扩散法、 肉汤稀释法或商用半自动系统。 虽然这些方法既经济又准确, 但是细菌培养和药敏试验至少要24小时才能得出结果, 不能满足临床快速检测的需求[4]。 近年来, 随着分子生物学、 微生物生理学等学科的飞速发展, 一些新型的药敏检测技术不断涌现, 如聚合酶链反应, 分子杂交技术、 流式细胞荧光法[5]、 质谱、 表面等离子体共振、 拉曼光谱、 激光镊子、 和阻抗方法等, 这些方法各有优缺点, 有的不能检测多重耐药菌, 有的需要昂贵的试剂, 有的样品预处理复杂耗时等[6]。 虽然总体上这些方法加快了检测速度, 一般能在几个小时内得到结果, 但在稳定性及可操作性上还需继续研究。
在以上新型检测方法中, 拉曼光谱技术显示出了快速鉴定致病菌及其耐药性的巨大潜力。 拉曼光谱是基于拉曼散射效应的分子振动光谱[7], 它能够提供关于分子结构和构型的“ 指纹” 信息。 其谱峰窄, 分辨率高, 可以灵敏显示致病菌间的细微差别[8]。 普通拉曼散射的拉曼散射信号弱、 效率低, 难以有效测量生物材料的拉曼光谱[9], 往往需要高光谱受激拉曼[10, 11]进行检测或者借助标记手段来检测, 例如重水标记[12]。 普通拉曼检测方法提供了微生物细胞的可重复拉曼光谱。 这种光谱再现性对于在不同实验室使用拉曼光谱进行微生物鉴定非常重要。 当样品与某些金属如银、 金等粗糙表面接触时, 其拉曼散射信号会急剧增强, 最高可达10个数量级以上[13], 这种现象称为表面增强拉曼散射相应的光谱称为表面增强拉曼光谱(surface enhanced Raman scattering, SERS)。 SERS检测致病菌所需样品量少, 可以实现无标记、 非接触、 它不需要任何有毒的免疫化学染色[10]。 免标记检测直接反应病原菌本身的信息, 免去了繁琐耗时的标记程序, SERS可以提供更精细更丰富的致病菌生化特征和对抗生素的生化反应[14]。 但是, 利用SERS技术检测致病菌还存在不足。 第一, 由于SERS“ 热点” 难以控制, SERS信号重复性欠佳。 第二, 致病菌体积小、 在溶液中处于运动状态、 难以聚焦测量。 第三, SERS光谱维度高且致病菌间光谱轮廓高度相似, 普通的数据分析方法不易区分。 针对上述SERS检测中面临的一系列困难, 各研究团队从SERS基底的设计、 致病菌的捕获技术以及数据分析方法三个方面提出了各种解决办法。
SERS基底的材料和结构对于致病菌检测至关重要, 不同的合成方法、 形貌和表面结构直接影响分析结果。 SERS基底的材料主要有银和金。 银的增强效果优于金, 但是银在空气中极易氧化。 SERS基底的结构直接影响基底的整体性能[15]。 SERS基底结构主要分为粒子型和平面阵列型。 平面阵列型SERS基底一般采用周期性点阵结构, 其SERS“ 热点” 分布均匀, SERS光谱重复性较好且方便直接测量。 粒子型种类繁多, 主要有球形、 杆状以及复合粒子。 银纳米粒子制备过程中粒子大小不一, 均匀性不佳, 在空气中极易氧化, 而金纳米粒子尺寸均一, 稳定性优于银纳米粒子。 因此, 采用先制备金纳米粒子再包裹一层银的方法制备的Au@AgNPs复合粒子, 粒子大小可控、 形状均匀、 稳定性极佳[16]。 粒子型SERS基底制备简便, 对其加以修饰可方便其附着在细菌表面。
Liu等[17]用电化学镀银的方法产生平均长度为60 nm的银纳米粒子阵列。 对甲氧西林敏感的金黄色葡萄球菌和野生型大肠杆菌以及临床分离株的抗生素敏感性试验(antibiotic susceptibility testing, AST)和最低抑菌浓度(minimum inhibitory concentration, MIC)快速检测, 其SERS光谱中的特定生物标记物的强度在两小时内明显下降。 表明其在补充现有耗时的方法以及帮助缓解临床细菌耐药方面具有很高的潜力。 Yuan等[18]研发了一种基于大小可调的Au@Ag纳米粒子包覆贻贝壳的复合SERS阵列基底[图1(a)— (d)]快速鉴别大肠埃希菌、 金黄色葡萄球菌和铜绿假单胞菌。 贻贝壳中CaCO3的高含量导致了分析物的超高疏水性, 交叉的纳米板和纳米通道提供了丰富的SERS热点。 对罗丹明的SERS检出限可低至10-9 mol· L-1。 这种新颖的Au@Ag自组装贻贝壳基底在基于SERS的生物传感器中作为低成本、 耐用和可复制的基底中具有相当大的应用前景。
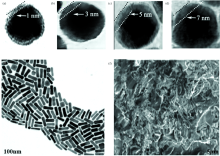 | 图1 (a)— (d)Au@AgNPs的高倍透射电镜图, Ag壳层厚度分别为1, 3, 5和7 nm[18]; (e) Au@AgNR的透射电镜图[16]; (f) B-g-PAMDAC的透射电镜图[20]Fig.1 (a)— (d) High-magnification TEM images of Au@AgNPs with Ag shell thicknesses of 1, 3, 5, 7 nm respectively[18]; (e) TEM images of Au@AgNR[16]; (f) SEM of B-g-PAMDAC[20] |
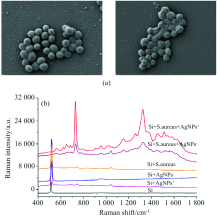
Wei等[19]以银纳米粒子为基底, 利用SERS检测5种食源性致病菌, 在10 min内实现了大肠杆菌O157:H7、 鼠伤寒沙门菌、 福氏志贺菌、 布鲁菌S2株以及金黄色葡萄球菌的快速鉴别, 且金属基底增强稳定性高, 重复性较好。 Bi等[16]开发了一个具有SERS活性的Au@Ag核壳纳米棒[图1(e)]用于超敏感细菌检测和抗生素敏感性试验(AST)。 纳米棒和细菌之间的等离子体效应提高了SERS检测限。 大肠杆菌的检测范围为107~102 CFU· mL-1。 通过分析细菌拉曼强度变化可以获得快速的抗生素耐药性检测(< 3.5 h), 该方法可以实现致病菌及耐药性的快速检测。 Zhang等[20]合成了氧化石墨烯(GO)和4-巯基苯基硼酸(4-MPBA)修饰的Au@AgNPs[图1(f)]。 细菌聚集在其周围导致大量的“ 热点” 并产生强烈的拉曼信号。 即使在低细菌浓度为103 CFU· mL-1时, 也具有较高的灵敏度、 准确性和稳定性。
在溶液中直接检测致病菌, 可以实时观测药物分子与致病菌的作用过程, 有利于进一步理解细菌与药物的相互作用, 是实现快速AST检测的有效途径。 但是, 致病菌在溶液中处于布朗运动状态, 不易对其直接测量。 目前, 人们采用了各种捕获并固定致病菌的方法: 核酸适配体、 免疫磁性分离、 微流控、 静电吸引等。
适体是一组单链DNA或RNA分子, 可以通过范德华力、 氢键、 疏水和静电相互作用结合到靶向细菌的表面。 适体的体积小, 易于合成和标记, 缺乏免疫原性, 以及靶标结合亲和力和特异性高。 一种细菌可以结合多种适体, SERS信号在原位被放大, 可实现高灵敏度的检测。 研究人员已经通过基于SERS的适体和拉曼标记成功检测出金黄色葡萄球菌和鼠伤寒沙门氏菌。 Zhang[21]、 Pang等[22]合成了适体-Fe3O4@Au磁性纳米粒子(图2)作为特异性细菌富集和SERS活化底物, 万古霉素-SERS标签用于致病菌的灵敏定量。 在50 min内可以达到3个细胞/毫升的检测限。 然而, 这些策略需要繁琐的修改过程或依赖于拉曼标记。 Gao等[23]依靠适体在细菌表面原位合成了细菌适体@AgNPs, 细菌SERS信号被其特异性识别的适体显著增强。 该方法无须标记, 检测限低至1.5 CFU· mL-1。
免疫磁性分离技术采用初级抗体修饰的磁性纳米结构来浓缩细菌, 其将夹心型测定与磁分离相结合。 磁性纳米材料的表面积与体积比具有较高的捕获效率。 采用链霉素-亲和素级联放大系统大大提高了致病菌的检测限, 致病菌与磁性纳米粒子采用抗原抗体特异性结合所需样品量少、 检测时间短。 使用大肠杆菌抗体结合磁性金纳米粒子(Fe3O4@Au)对大肠杆菌进行选择性分离和富集[24]以及计数, 该方法快速、 灵敏, 总分析时间少于70 min。 Kearns等[25]使用凝集素功能化的磁性纳米颗粒捕获细菌[图3(a)], 然后使用菌株特异性抗体功能化SERS活性纳米颗粒特异性检测细菌[图3(b)和(c)]。 成功分离并检测出三种致病菌: 大肠杆菌、 鼠伤寒沙门氏菌和耐甲氧西林金黄色葡萄球菌[图3(d)], 每种菌株的最低检测浓度仅为101 CFU· mL-1。 Yuan等[26]利用抗菌肽功能化的磁性纳米粒子以及4-巯基苯硼酸标记的Au@Ag-GO纳米复合材料成功分离大肠杆菌、 金黄色葡萄球菌和绿脓杆菌[图3(e)— (f)]。 此外腺苷酸修饰的Fe3O4纳米粒子具有高抗菌活性, 可以同时分离、 识别和杀死细菌。
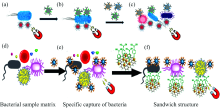 | 图3 (a)凝集素(Con A)功能化的镀银磁性纳米颗粒(Ag@MNPs)与细菌结合, 磁体的存在下从样品基质中分离细菌; (b)抗体和SERS标签添加; (c)将3个AgNP缀合物与3种细菌病原体和Con A(结合所有三种细菌)一起加入, 利用532 nm激光进行分析[25]; (d)腺苷酸修饰的与样品基质(包括细菌、 血细胞或其他干扰物)一起培养; (e)Fe3O4NPs @细菌复合物与样品基质磁性分离; (f)4-巯基苯硼酸修饰的Au@Ag-GO与Fe3O4NPs @细菌复合物一起培养以形成夹层结构[26]Fig.3 (a) Agglutinin (Con A) functionalized silver-plated magnetic nanoparticles (Ag@MNPs) are combined with bacteria, and the bacteria are separated from the sample matrix in the presence of a magnet; (b) Antibody and SERS tag addition; (c) 3 AgNP conjugates are added together with 3 bacterial pathogens and Con A (combined with all three bacteria), and analyzed by 532 nm laser[25]; (d) AMP modified and The sample matrix (including bacteria, blood cells or other interfering substances) are cultured together; (e) Fe3O4NPs@bacterial complex is magnetically separated from the sample matrix; (f) 4-MPBA modified Au@Ag-GO is cultured with Fe3O4NPs@bacterial complex to form a sandwich structure[26] |
微流控是在微米尺度下对微量的流体进行精准操控的一种技术, 多种微流控芯片可实现致病菌分离。 电泳可以通过施加电场使微通道中的分子和粒子产生偏移从而达到富集或分离的目的。 多电极微流控芯片施加电流时, 不同的电极组合方式会产生不同的电场分布, 实现致病菌分离。 该技术具有低成本、 便携性、 易控制、 低剂量、 响应时间短、 精度和灵敏度高等优异的特征。 Chang等[27]开发了一种集成膜过滤和SERS 检测的微流体装置[图4(a)], 用于细菌富集、 代谢物提取和原位SERS测量。 封闭的微环境可以保证代谢物在SERS底物上稳定、 均匀的吸附。 通过比较抗生素处理前后细菌分泌的代谢物的特征拉曼信号变化可以在2 h内准确确定最低抑菌浓度, 并且不会受到细菌聚集和不均匀分布问题的影响。 Madiyar等[28]报告了通过在微流控芯片的底部嵌入垂直排列的碳纳米纤维制成的纳米电极阵列[图4(b)和(c)], 用于有效地捕获和浓缩大肠杆菌。 然而, 这些器件存在限制了吞吐量和分离效率, 电极污染, 以及无法处理样品碎片等不足。 Honson等[29]发明了一种非接触的介电泳-SERS器件[图4(d)], 电极通过绝缘屏障与样品通道隔离, 防止电极污染, 该器件集成了阻抗分析可以分离和捕捉细菌, 并且识别碎片。 该装置已成功地从3 μ m聚苯乙烯微球中分离出了分枝杆菌, 准确率为100%。
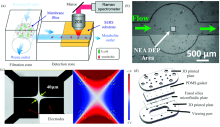 | 图4 (a)微流控系统及器件原理图[27]; (b) 在4倍放大下拍摄的光学显微镜图像, 显示微流体通道和中心的活性正方形[28]; (c)四个具有多项式边界的电极对称排列以及电势场分布和电泳力的曲线图[30]; (d)介电泳-SERS装置的示意图[29]2.4 静电结合检测Fig.4 (a) Schematic diagram of the microfluidic system and device[27]; (b) Optical microscope image taken under 4X magnification showing the microfluidic channel and the active square at the center[28]; (c) The symmetrical arrangement of four electrodes with polynomial boundaries and the graphs of electric potential field distribution and electrophoretic force[30] ; (d) Schematic diagram of DEP-SERS device[29] |
革兰阳性菌表面的肽聚糖和革兰阴性菌表面的脂多糖都带负电荷, 负电荷脂多糖是几种革兰氏阴性细菌的主要内毒素和外膜成分, 带负电荷的细菌表面物质成为致病菌SERS检测的生物标志物。 因此, 带正电荷的纳米粒子和带负电荷的细菌结合后, 纳米粒子在致病菌表面形成大量的SERS “ 热点” , 这种检测方法大大增加了致病菌及其耐药性的检测限。 Fang等[31]发现带正电荷的银纳米粒子通过静电聚集在细菌壁表面, 增强的SERS信号优于标准基底负电荷银纳米粒子。 Chen等[32]使用带正电的银纳米粒子鉴定耐甲氧西林金黄色葡萄球菌(图5)。 为了进一步证明其在临床样本鉴定中的适用性, 检测52株金黄色葡萄球菌和215株耐甲氧西林金黄色葡萄球菌, 该方法成功检测出耐甲氧西林金黄色葡萄球菌。 Tadesse[33]使用十六烷基三甲基溴化铵(CTAB)合成了带正电的金纳米棒并表征了水中革兰氏阴性大肠杆菌和粘质沙雷氏菌以及革兰氏阳性金黄色葡萄球菌和表皮葡萄球菌的SERS特征。 金纳米棒和细菌之间的等离子体和静电相互作用使SERS均匀增强。 具有较高表面电荷密度的细菌表现出明显较高的SERS信号。 Qiu等[34]制备的巯基-聚乙二醇-NH2功能化的Au@Ag纳米棒柱状阵列可以有效地捕获带负电的致病菌。 实现了三种代表性食源性革兰氏阳性菌, 即木糖葡萄球菌、 单核细胞增生李斯特菌和屎肠球菌的高灵敏度检测。
拉曼信号由细菌的多种成分产生, 且光谱维度高、 光谱复杂[35]; 不同种类细菌的基本成分非常相似, 导致相似的SERS信号。 因此, 需要高效的数据分析方法来处理致病菌的拉曼光谱。 经典的数据分析方法为多变量统计分析如: 主成分分析、 聚类分析、 判别分析等。 机器学习方法有支持向量机、 深度学习等等。 在这些方法中, 深度学习技术表现出了优于传统方法的光谱分类和识别能力。
多变量统计分析方法是一种传统的分析高维数据的方法, 一般通过特定的算法提取原始数据中的有效信息降低数据维度, 从而有利于后续数据分析。 主成分分析(principal component analysis, PCA)是最常用的多变量分析方法。 主成分分析通过正交变换将原始变量转换成一系列不相关的变量, 转换后的一系列变量称为主成分。 由于少量头部主成分包含了绝大部分原始数据的有效信息, 一般在分析时只截取前几个主成分, 从而降低数据维度。 Hadjigorgiou等[36]使用SERS对肺炎克雷伯氏菌、 变形杆菌和大肠杆菌进行AST, 使用PCA结合线性判别分析和留一交叉验证区分耐药菌、 中间菌和敏感菌, 分类正确率达94%。 Premasiri等[37]用SERS研究了两株细菌, 通过偏最小二乘判别分析区分耐药菌和敏感菌。 判别的敏感性> 95%, 特异性> 99%。 Ayala等[38]利用SERS结合PCA和判别分析用于区分不同的种类的致病菌。 Lin等[39]建立了一种基于SERS和层次聚类分析的大肠杆菌、 鲍曼不动杆菌和铜绿假单胞菌耐药性快速检测方法。 对123株临床分离菌(42株大肠杆菌、 41株鲍曼不动杆菌和40株铜绿假单胞菌)进行了检测。 与微量肉汤稀释法相比, 检测灵敏度和特异性分别为90.9%和91.1%。 筛查易于进行, 可在1.5 h内完成。
机器学习通过大规模的学习训练数据集, 从而提取数据的有效特征, 建立预测模型实现高灵敏度判别分析。 传统的机器学习方法有支持向量机、 决策树、 K近邻分类算法等。 Cheong等[15]采用多元统计学方法PCA和支持向量机来快速鉴定喹诺酮类耐药肺炎克雷伯菌(ATCC70063, ST11和ST15)。 该方法具有高重现性(相对标准偏差为7.4%)和灵敏的检测限(100 pmol· L-1, R6G), 相关系数为0.98。 Walter等[40]比较了支持向量机和线性判别分析区分两种不同激发波长(532和244 nm)的致病菌SERS光谱的能力。 分别实现了大约92%和90%的识别率。 Ciloglu等[41]使用SERS结合机器学习技术快速鉴定耐甲氧西林金黄色葡萄球菌和嗜肺军团菌。 K近邻分析方法在支持向量机、 决策树和朴素贝叶斯等传统分类器中具有97.8%的分类准确率。 SERS和机器学习为抗生素耐药菌的鉴别提供了一个可靠的工具。
深度学习是最先进的机器学习算法, 它从大规模的原始数据集中学习特征并直接构建预测模型。 深度学习算法有很多, 包括卷积神经网络, 完全连接的网络, 残差神经网络等。 其在挖掘数据的局部特征和提取全局训练特征方面具有良好的性能[42], 数据分类识别的能力远超传统多变量统计分析算法[43]。 Ho等[44]通过训练一个卷积神经网络准确识别30种常见的细菌病原体, 即使在低信噪比谱上, 也能达到超过82%的准确率并且抗生素治疗识别准确率高达97.0%± 0.3%。 对50名患者的临床分离株进行了验证, 仅使用来自每个患者分离物的10个细菌谱, 实现了99.7%的抗生素治疗识别准确率。 Thrift等[45]利用卷积神经网络联合SERS技术在抗生素暴露后10 min内将大肠杆菌和铜绿假单胞菌对抗生素的反应与未处理的细菌分离, 准确率大于99%。 这种无需对细菌进行培养的方法显著提高了分类精度, 可在30 min内选择有效的抗生素治疗, 这极大地减少了用常规生长试验验证表型AST所需的时间。
表面增强拉曼光谱是一种分子检测技术, 它能够无损害、 快速、 高灵敏度地提供致病菌与药物相互作用的信息[46]。 通过人们的不断努力, 基于SERS的致病菌及其耐药性检测的准确性已大幅提高, 检测时间也缩短为两小时以内, 但是要实现该方法在临床上的全面应用尚有许多工作需要完善。 首先, SERS基底的重复性有待提高。 其次, 需要探索操作简单、 特异性高的致病菌捕获方式。 希望未来的研究能提高SERS检测致病菌的易用性, 缩短从样本收集到检出结果的周转时间, 构建一个高灵敏低成本的测试平台, 并建立和完善细菌的标准SERS图谱数据库。 使 SERS细菌鉴别技术走出实验室, 有效应对并缓解临床细菌耐药。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|