作者简介: 李 梦, 女, 1992年生, 中国中医科学院中药资源中心助理研究员, 河南中医药大学博士研究生 e-mail: limeng0642@163.com
人参是传统中药材中的贵重品种, 具有较高的经济价值。 人参生长的地域性很强, 不同产地人参有效成分含量存在差异, 人参因“道地”与否, 会导致其质量、 医学效用和经济价值的差异, 因此人参产地识别的意义重大。 目前常通过磨粉提取等制备, 再采用化学或光学等多种手段检验人参产地, 但会造成样本破坏。 而基于外观性状或芦头特征的鉴别, 因主观性差异不能作为标准化的识别方法。 如何用高精度、 无损、 快速检测识别的方法, 对人参的产地进行识别分析, 是该研究的主要立足点。 通过采用高光谱成像技术, 对已知产地信息的人参样本, 通过获取从4002 500 nm的反射光谱, 经过基于白板的绝对和相对辐射校正处理, 构建了高光谱反射率数据集。 采用随机森林的机器学习方法, 构建了基于高光谱数据的全光谱人参产地识别模型, 并对不同尺度的地域划分规则分别开展了产地识别精度验证, 发现不同产地的人参光谱有明显区别。 其中东三省与否的产地识别精度, 可以达到98.2%。 同时利用随机森林基于决策树构建的优势, 获得了人参产地识别的光谱重要性结果, 为专用轻量化仪器研发指明特征光谱。 高光谱人参产地识别研究作为严格的无损检测方式, 将对人参等道地药材的产地识别、 药材图谱指纹认知和挖掘、 药材鉴定和质量评价等提供理论支撑和技术手段。
Ginseng is a valuable variety of traditional Chinese medicine with high economic value. The growth is very regional, and the effective ingredients of ginseng from different origins are different. Whether ginseng is “authentic” or not, it will cause differences in its quality, medical utility and economic value, so the identification of ginseng origin is of great significance. After powder extraction and other preparations, chemical or optical methods are used to test the origin of ginseng, but this will cause damage to the sample. Besides, the identification based on appearance traits or rhizome head characteristics can not be used as a standardized recognition method because of human subjective differences or easy to be falsified. The main standpoint of this article is how to use high-precision, non-destructive, and rapid detection and identification methods to identify and analyze the origin of ginseng. This experiment uses hyperspectral imaging technology, for ginseng samples with known origin information, the hyperspectral reflectance dataset was constructed by obtaining reflectance spectra from 400 to 2 500 nm, after absolute and relative radiometric corrections based on the whiteboard. A full spectrum ginseng origin recognition model based on hyperspectral data was constructed, and the accuracy of origin recognition was verified for different scales of regional division rules. It was found that the ginseng spectra from different origins were significantly different. The accuracy of origin identification of the northeastern provinces or not can reach 98.2%. The spectral importance results of ginseng origin recognition were given, indicating the characteristic spectrum for developing a special lightweight instrument. As a strict non-destructive detection method, hyperspectral ginseng origin identification research will provide theoretical support and technical means for identifying the origin of authentic Chinese medicinal materials such as ginseng, fingerprint recognition and mining of medicinal materials, identification and quality evaluation, etc.
人参是五加科植物人参(Panax ginseng C. A. Meyer)的干燥根和根茎, 是我国名贵中药材, 应用历史悠久。 栽培的俗称“ 园参” , 播种在山林野生状态下自然生长的称“ 林下山参” , 习称“ 籽海” [1]。 人参早在秦汉时期应用已较为普遍, 在《神农本草经》中被列为上品, 记载其具有“ 主补五脏, 安精神, 定魂魄, 止惊悸, 除邪气, 明目, 开心益智。 久服, 轻身延年” 功效。
《中国药材学》记载: “ 野生品称山参、 野山参, 主产于东北长白山区, 大、 小兴安岭, 栽培品称园参, 主产于吉林、 辽宁、 黑龙江; 河北、 山西、 山东、 湖北及北京等地有引种试种” 。 依据历代本草记载, 人参最早出于山西上党(潞州)及辽东等地, 后因资源枯竭, 至明之后, 基本以东北为主产地, 奉为道地。 道地中药材, 是指经过中医临床长期应用优选出来、 产在特定地域, 与其他地区所产同种中药材相比, 品质和疗效更好, 且质量稳定, 具有较高知名度的中药材。 故人参道地药材指产于以东北长白山山脉为中心, 核心区域包括吉林抚松、 集安、 靖宇, 辽宁宽甸、 桓仁及周边地区, 也包括黑龙江大兴安岭、 小兴安岭等地区的人参。
近年来, 通常采用性状观察法、 化学指纹图谱法、 光谱分析、 分子识别等方法[2, 3, 4, 5, 6, 7]进行人参产地的识别, 但上述方法均要求有一定的经验积累或者专业知识, 同时在识别的过程中易造成样品损毁, 故对于经济价值较高的人参药材, 迫切需要研发一种无损的检测方法。 本工作采用高光谱成像技术对人参的产地进行识别分析, 该技术具备快速无损的突出优势, 其电磁波在较短的波长范围内(如4002 500 nm)照射到人参药材上产生反射信号, 测量时间短, 不对人参药材造成损坏, 未涉及到热辐射波段, 不受环境温度影响, 通过对其高光谱数据进行分析来识别人参产地。 本研究以我国黑龙江、 吉林、 辽宁、 山东四省十个地区的人参样品为研究对象, 采用高光谱成像设备获取人参药材的光谱反射率信息, 基于具备部分可解释性的随机森林机器学习模型对人参进行产地识别。
收集黑龙江省(伊春市、 铁力市、 虎林市), 吉林省(抚松县、 靖宇县、 临江市、 长白县、 珲春市)、 辽宁省(宽甸县)、 山东省共十个不同产地的54个人参(园参)样品。 统一进行简单清洗及干燥处理。 随机选取一定数量样本作为机器学习的训练数据集, 剩余的样本作为测试验证数据集。 机器学习方法的训练和验证重复10次, 以测试方法稳定性。
人参的高光谱数据使用NEO公司的两台相机获取: Hyspex VNIR-1024的可见光近红外高光谱相机和Hyspex SWIR-384短波红外高光谱相机。 二者均为线阵扫描方式, 线阵探元个数分别为1 024和384, 覆盖波段分别为4001 000和9402 500 nm, 联合使用可以覆盖4002 500 nm的光谱范围。 使用暗室环境拍摄, 内置稳定人工光源, 保证所有样本的高光谱数据是在同样的光照条件下获取。 人参样本放置在黑色背景中接受扫描。 扫描成像的同时放置具有接近朗伯体反射特性的白板, 用以实现绝对和相对辐射校正。
将每个人参样本的高光谱图像处理成一条光谱曲线。 数据处理方案如下。
(1)为减小采集过程中光源分布不均及镜头中暗电流造成的噪声影响, 对每个波段的图像进行相对和绝对辐射校正。 白板以上的所有像素值(digital number, DN) 按照式(1)进行校正, 校正后得到反射率
式(1)中, λ 为电磁波长, DN(i, j)为校正前的第i行, 第j列的像素值, E(DNwb(:, j))是第j列白板所有像素值的平均值, 此处平均计算目的是消除白板因尘埃污染等造成的空间反射差异。 将DN值除以白板值定义为是归一化到白板反射率为1情况下的人参反射率数值, 通过白板作为参考完成绝对辐射校正。 所有样本中的反射率绝对值具有大小可比性。 其值域范围理论上为从0到无穷大, 实际上处于(0, 2.5)的区间。 从白板亮度可以看出相机扫描的每个探元对应的光照条件并不一致, 呈现中间亮边缘暗的低频相对辐射差异, 探元之间响应能力不同导致固定的高频相对辐射差异, 通过按照每列分别除以白板均值, 可以完成相对辐射校正。
(2)图像分割。 通过统计黑色背景、 白板、 人参在单波段的数值差异, 构建了仅基于单波段反射率阈值的人参目标图像分割方法, 可以确定人参所包含的所有像素, 完成人参目标的图像分割, 存为二值图像掩膜Mask, 1代表人参, 0代表非人参。
(3)获得反射率光谱曲线数据, 计算方法如式(2)所示。
式(2)中, ρ λ 是一个数值, 表示波长为λ 的反射率, 公式中分子表示波长为λ 的图像中所有人参像素反射率之和, 公式右侧分母表示人参像素数量。 通过循环处理高光谱图像的每个波段, 每个样本可以得到一条反射率光谱曲线。
随机森林是一种包含多个决策树的机器学习模型, 大多用于解决分类问题, 随机森林的输出是所有决策树输出的众数。 “ 森林” 中的单个决策树使用部分样本进行训练, 因此每个决策树都是“ 弱分类器” , 最终结果取决于多个弱分类器投票表决。 因使用了决策树, 随机森林可以根据输入特征作为决策依据的重要程度, 给出输入特征的重要性排序, 从而具备部分可解释性。
人参高光谱产地识别系统基于scikit-learn0.23.2版本, 使用python语言开发, 随机森林设置使用默认参数。 产地, 是一个通俗说法, 在研究中需要明确地域尺度大小才能进行识别研究。 共使用三种产地归类尺度, 分别为东北与否二分类、 省域四分类、 县级或地级八分类识别。 通过测试验证数据集预测混淆矩阵给出结果的总体精度进行评价。
将人参样品摆放于移动平台上, 摆放时突出每一样品的特征, 将用于黑白校正的白板摆放在样品后方5 cm处。 通过高光谱设备采集数据, 在高光谱数据收集完成后, 为消除仪器对样品数据的影响, 利用仪器自带RAD校正软件校正原始高光谱图像。 得到单个样品的高光谱图像如图1(a, b)所示。
因不同相机在采集样品高光谱图像的过程中, 可能会受光源分布不均及镜头中暗电流造成的噪声等多重因素影响, 故对4001 000和9402 500 nm两个不同波段范围的高光谱图像分别进行数据处理, 得到每个样品的反射率光谱曲线。
其光谱曲线数据, 反射率绝对值具有大小可比性, 且避免了人参单一位置光谱因杂质等造成噪声、 因选取部位不同造成光谱不可比等问题, 具有较高的信噪比和稳定性。
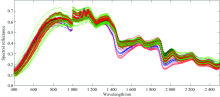
为合并分析4002 500 nm光谱范围内人参高光谱曲线规律, 将两个不同波段范围的人参反射率光谱曲线在1 000 nm处拼接在一起。 可见-近红外波段的相机(visible-near infrared, VNIR)和短波红外(short wavelength infrared, SWIR)两台相机拍摄的灯光照射角度不同, 两个反射率的方向定义差异导致形成曲线断层, 但每个样本的拍摄条件相同, 不影响随机森林方法识别。 通过数据处理后, 得到54个人参样品反射率光谱曲线如图2所示。 其中黑龙江省样品标为红色, 吉林省样品标为绿色, 辽宁省样品标为蓝色, 山东省样品标为黑色。
从图2中可以看出, 仅靠反射率大小很难将不同产地分开, 使用机器学习的方法是一种较好的解决方案。 在当前的54个样本集中, 将东北与否二分类随机森林随机选取20%(11个)用于验证, 共验证识别110次; 考虑到总样本数量有限, 参与训练的样本要保障一定数量, 四省分类和八地分类尺度, 按照随机选取10%(5个)用于验证。 按照三种产地归类尺度, 每种尺度分别使用随机森林训练并验证重复10次, 验证结果如表1所示。
| 表1 三种产地归类尺度下的识别精度(百分比) Table 1 The recognition accuracies under three origin classification scale (100%) |
人参是我国东北三省的“ 三宝” 之一, 东三省是传统的人参产区。 首先按照东三省与否来进行产地区分, 共有2次将东北人参错分为山东人参, 平均总体精度98.2%, 对于人参产地是否属于东三省“ 道地产区” 的识别具有较高的实际应用价值。 受到本批次人参样品数量限制, 四省分类和八地分类已经受到样本数量的影响, 平均总体精度分别为82%和68%。 尤其是八地分类, 随机选取训练和识别样本不同, 导致验证精度从100%可能降到20%, 样本数量少导致的学习不足最为明显。
从三种产地归类尺度均有100%识别精度的情况来看, 可以预期在样本数量增加的情况下, 所有产地归类尺度下的随机森林识别精度将会进一步提升。
将三种产地归类尺度下, 按照(1)超过80%验证精度, (2)各波段重要性累加后, 需要占到全部光谱的重要性的96%以上。 将符合上述两个条件的训练和验证轮次得到的光谱波段重要性进行了统计, 将重要性高的波段视为特征波段, 如表2所示。
| 表2 随机森林统计出的人参产地识别特征波段 Table 2 The feature bands statistics of ginseng origin recognition by random forest |
从表2可以看出, 对于东北人参与否的识别, SWIR具有明显优势, 在训练样本数量够多的情况下, 甚至仅使用SWIR光谱相机即可满足应用需求。 对于四个省份的人参识别, 依然是短波红外占据了主要信息量, 仍然存在仅使用SWIR即可达到较高精度的可能。 对于县级和地市级区分的八地识别, 暂无明确结论。
(1)基于机器学习方法, 可以仅通过光谱信息进行高精度的人参产地识别, 在四省和东北与否两种尺度下, 识别精度分别可达82%和98.2%。 按照纯反射光谱的识别要求开发专用设备, 将具有高精度、 无损、 快速、 普通人可以简易操作的优势。
(2)可见-近红外波段的相机(VNIR)和短波红外(SWIR)相机因探测器不同, 可以认为是两台设备, 在产地识别中, 应重点探索基于SWIR的识别技术和硬件方案。
(3)人参属于贵重中药材, 机器学习方法需要采集购买足够多的样本来提高识别算法的精度。
(4)基于决策树的机器学习方法有利于发现描述产地之间差异的特征光谱, 为进一步建立人参高光谱图谱提供支撑。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|