作者简介: 陈楚汉, 1997年生, 华南农业大学电子工程学院硕士研究生 e-mail: 597748426@qq.com
使用近红外光谱鉴别蚕茧雌雄设备成本较高, 挑选有用特征可以减少成本。 雌雄蚕茧的近红外光谱存在着共线性的关系, 因此提出了一种包裹式的特征选择方法, 基于支持向量机的自助重加权采样(BRS-SVM)的特征选择方法。 使用NirQuest512近红外光谱仪采集了蚕茧的漫透射近红外光谱。 用试验集的全波段建模得到特征重要度热图, 并通过热图得到重要特征波段的范围。 然后在重要特征波段范围内, 分别用BRS-SVM、 基于SVM的特征排序方法(MBR-SVM)、 基于逻辑回归的特征排序方法(MBR-LR)、 递归特征消除法(RFE)、 连续投影算法(SPA) 和遗传算法(GA)挑选单波段特征和连续波段面积特征, 再分别用支持向量机(SVM)和逻辑回归(LR)建立雌雄分类模型。 通过特征重要性热力图发现, 蚕茧雌雄分类重要区域在900~1 399 nm内, 用此波段范围建立SVM模型, 试验集准确率为99.40%。 用BRS-SVM挑选5个单波段特征, 然后再用SVM建模, 验证集准确率为93.88%, 高出其他特征选择方法5%~12%, 测试集准确率为89.56%, 测试集准确率高出其他特征选择方法2%~4%。 用BRS-SVM挑选27个单波段特征, 建立SVM雌雄分类模型测试集准确率为94.97%, 准确率达到生产条件要求。 用BRS-SVM挑选的14个连续波段面积特征, 再用SVM建模, 测试集准确率为94.43%。 在挑选少量特征情况下, 我们提出的BRS-SVM要优于其他方法。 用BRS-SVM挑选少量的特征, 可以建立性能良好的蚕茧雌雄分类模型, 有效减少了成本, 具有重要的现实意义。
The cost of identifying male and female cocoons by NIR is high, and the cost can be reduced by selecting useful features. Since there is a nonlinear relationship between the NIR spectra of female and male cocoons, a wrapper feature selection method, Bootstrapping Re-weighted Sampling Support Vector Machines (BRS-SVM), was proposed. The diffuse transmission NIR spectra of silkworm cocoons were collected by NirQuest512 NIR spectrometer. The heat map of characteristic importance was obtained by modeling the whole band of the test set, and the heat map obtained the range of important characteristic bands. Then, in the range of important characteristic bands, the single band features and continuous band area features were selected by BRS-SVM, Model-based ranking support vector machines (MBR-SVM), Model-based ranking Logistic Regression feature sorting method (MBR-LR), Recursive feature elimination (RFE), successive projections algorithm(SPA), Genetic Algorithm(GA), and then the support vector machines (SVM) and Logistic Regression (LR) sex classification models were established respectively. According to the characteristic importance heat map, it is found that the important area of male and female classification of silkworm cocoon was within 9001 399 nm. We used this band to build the SVM model, and achieved 99.40% accuracy. BRS-SVM was used to select 5 single-band features. The accuracy of the test set is 89.56%, which is 2%4% higher than other feature selection methods. RS-SVM was used to select 27 single-band features, and the accuracy of the test set of the SVM gender classification model was 94.97%, which reached the requirements of production conditions. The accuracy of modeling test set by BRS-SVM was 94.43% for 14 continuous band features. In the case of selecting a small number of features, our proposed BRS-SVM is superior to other methods. Using BRS-SVM to select a small number of features, we can establish a good performance of the female and male cocoon classification model, effectively reduce the cost, has important practical significance.
蚕茧雌雄鉴别是蚕茧杂交育种的重要一步[1]。 从熟蚕上蔟到蚕蛹化蛾共约14 d, 蚕种场一般在第8天进行削茧鉴蛹辨别雌雄, 削茧鉴蛹时间只有4~5 d, 在短时间内, 完成削茧鉴蛹需要大量人工, 劳动成本高。 使用近红外光谱对蚕茧进行雌雄鉴别, 成本比较高, 使用较少的近红外波段可以节约成本。
目前关于蚕茧性别自动鉴定的方法大多都是有损的, 需要人工削茧, 这些方法有荧光蚕茧辨性[2]、 磁共振成像、 X射线成像技术, 高光谱成像技术[3], 计算机视觉方法和近红外光谱分析[4, 5]等。 目前还没有结合化学计量学和近红外光谱的蚕茧性别自动鉴别的研究[6]。 使用全波段光谱进行分析, 仪器成本较高, 无法大规模应用在实际生产中。
数据提取是把之前维度的特征映射到一个更低维度的空间[7], 但数据提取的方法无法减少使用的近红外光谱波段。 在近红外光谱分析中, 用特征选择方法挑选单波段特征[8], 然后用挑选出来特征波长对应的单波发光二极管(LED) 或激光光源代替近红外光谱仪[9], 能节约设备成本。
根据上述需求, 提出了一种基于统计学的包裹式方法, 基于SVM的自助重加权采样(bootstraping re-weighted sampling support vector machines, BRS-SVM)的特征选择方法。 近红外光谱分析依靠不同样品光谱间的微小变化进行分析[10], 连续波段面积能很好反映出不同样本光谱间的微小差异。 用BRS-SVM分别挑选单波段特征和连续波段面积特征, 再用支持向量机(support vector machines, SVM)和逻辑回归(logistic regression, LR)建立雌雄分类模型, 以挑选相同特征个数时模型的准确率对特征选择方法评估, 并和其他特征选择方法比较, 分析实验结果, 以期选择合适数量的窄LED灯代替近红外光谱仪。
基于学习模型的特征排序方法是基于学习器, 通过衡量学习器特征的权重大小, 给特征重要性排序, 去除不重要的特征。 其优势是可以快速去除大量不重要特征, 但是不适合挑选较少特征。 本工作使用基于SVM的特征排序方法(model based ranking support vector machines, MBR-SVM)和逻辑回归LR的特征排序方法(model based ranking logistic regression, MBR-LR)。
特征选择的方法分为过滤试, 包裹式和嵌入式。 包裹式特征选择法的特征选择过程与学习器相关, 使用学习器的性能作为特征选择的评价准则, 选择最有利于学习器性能的特征子集[11]。 递归特征消除(RFE)是一种包裹式特征选择的方法, 该方法类似使用了多次基于学习模型的特征排序方法, 每次迭代消除少量特征。 以SVM-REF为例, 在每一轮训练过程中, 会选择所有特征来进行训练, 继而得到了分类的超平面, SVM-REF会消除较小的权重, 本工作每次迭代消除两个特征。
连续投影算法(successive projections algorithm, SPA) 是前向特征变量选择方法。 SPA利用向量的投影分析, 通过将波长投影到其他波长上, 比较投影向量大小, 以投影向量最大的波长为待选波长, 然后基于矫正模型选择最终的特征波长。 SPA选择的是含有最少冗余信息及最小共线性的变量组合。
遗传算法(genetic algorithm, GA)是模拟达尔文进化论的自然选择和遗传学机理的生物进化过程的计算模型, 是一种模拟自然进化过程搜索最优解的方法, 利用选择, 交叉和突变等进化因子使得种群的适应度不断增强, 从而达到优胜劣汰的目的。 本工作利用SVM给个体适应度评分。
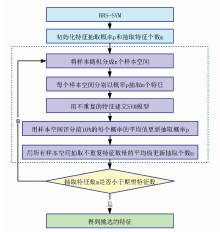
BRS-SVM是一种包裹式法, 该方法通过统计学的方式, 评价不同组合的特征子集的得分, 逐步选取最优的特征子集, 子集搜索策略是启发式搜索策略, 这种搜索策略效率要远优于全局最优搜索; 自助法是一种启发式搜索策略, 在光谱特征选择中有着较好的效果[12]。 BRS-SVM能够快速有效的寻找最优的特征组合。 BRS-SVM大致可以分为子集搜索和子集评价部分, 首先初始化每个特征的权重u和抽取特征的数量, 其中每个特征的初始权重u相等且和为1, 抽取的特征个数等于样本特征个数。 子集搜索部分: (1)首先初始化n个样本空间, 即重复n次将数据随机分成80%的训练集和20%验证集, 样本空间个数n越大, 统计次数就越多; (2)在n个样本空间下, 每个样本空间按权重为u进行随机重复抽样, 抽取出m个特征。 子集评价部分: (1)根据自助法, 排除重复的特征, 剩下约0.632 m个不重复的特征; (2)每个样本空间分别用SVM建模, 然后用验证集准确率评价抽取的特征子集; (3)得分前10%的特征子集有利于学习器的性能, 以得分前10%的特征抽取频率更新特征的权重u; (4)以所有样本空间抽取不重复特征个数的评价值更新抽取个数m。 重复子集搜索和子集评价部分, 直到抽取个数m满足需求, 算法流程图如图1所示。 设置BRS-SVM的样本空间大小为200。
所有实验都重复计算50次, 再求平均值, 其中准确率的定义如式(1)所示
式(1)中, T为数据集分类正确的数量, F为数据集分类错误的数量。
所有的运算都是在个人计算机上(Intel Core i5-4200, 2.8 GHz CPU和12GB内存)用Pycharm(Python版本3.6.5, Tensorflow版本1.14.0, Keras版本2.3.1)进行的。
样本的漫透射光谱采集使用课题组自行研制的种茧自动分选样机完成, 光谱仪为海洋公司的NirQuest512型便携式光纤光谱仪, 检测范围: 900~1 699 nm。 光谱仪设置积分时间为200 ms, 平均次数为4以提高数据的稳定性, 平滑宽度为4以匹配系统的分辨率, 样机如图1所示。 样机工作步骤如下:
(1)将未剥壳的蚕茧放入左边进料口中, 机械臂会抓取蚕茧到转盘中。
(2)转盘再将蚕茧转到光源(100 W的卤灯泡)处, 光源从上向下照射蚕茧, 积分球在蚕茧下面采集蚕茧的漫透射光, 通过600 μ m光纤连接光谱仪。
(3)通过USB线将光谱仪采集的光谱数据传输给电脑, 保存数据。
试验用的家蚕种茧样本来自于广东省蚕业推广中心和广东化州种茧场。 将2019年4月至2020年10月采集的4517个近红外光谱样本作为试验的数据集, 2021年6月采集的1 695个样本作为测试集, 其中数据集信息如表1所示。 9芙× 7湘是9芙和7湘的第一代杂交品质, 它们体型大小十分接近。 试验集和测试集数据的采集时间不同, 但他们品种接近, 用测试集数据能很好验证试验的有效性。 将茧壳削开, 通过观察蚕茧尾部花纹来判断蚕蛹雌雄。
| 表1 试验数据集的详细信息 Table 1 Details of the data sets |
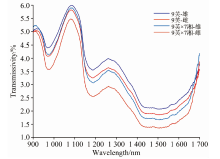
图3为9芙和9芙× 7湘通过NirQuest512型便携式光纤光谱仪采集到的雌雄蚕茧平均光谱, 采集范围为900~1 699 nm。 由图3可以看出, 两种品种的蚕茧雌雄光谱有5个相同的谱峰, 峰值波长分别为918, 970, 1 084, 1 186和1 269 nm。 两种品种雌雄蚕茧的平均近红外光谱的谱峰差别不大, 且它们谱峰都较宽。 通常, 雌蚕蛹的个体要比雄蚕蛹的大, 所以相同品种情况下, 雌蚕茧的平均近红外漫透射率要低于雄蚕茧的。 雌雄蚕茧的漫透射近红外光谱存在交叉, 但其交叉规律较为复杂, 很难观察出雌雄蚕茧光谱差异较大的波长, 因此需要使用相关算法挑选出相应的特征波长。
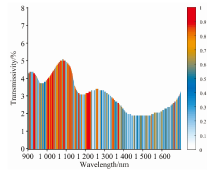
将试验集随机分为80%的训练集和20%的验证集。 使用训练集的全波段光谱数据建立SVM模型, 验证集准确率为99.16%, 以该SVM模型的权重大小为评判标准, 权重越大特征越重要, 将900~1 699 nm波段特征的重要性排序, 并根据排序将重要程度缩放到0~1, 其中重要程度的计算如式(2)所示
式(2)中, S为特征重要性的排序。 得到全波段特征重要性热力图, 如图4所示, 辞雄分类的重要特征都集中在900~1 399 nm, 使用该波段范围的训练集建立SVM模型, 验证集准确率为99.40%, 所以我们认为雌雄分类信息大部分在900~1 399 nm波段内。
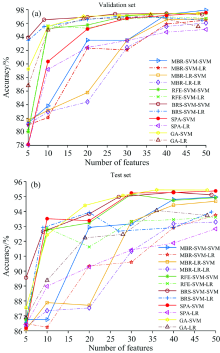
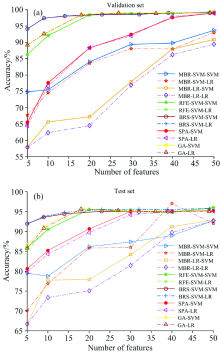
在900~1 399 nm波段内挑选蚕茧雌雄分类的有用信息, 分别使用MBR-SVM, MBR-LR, REF-SVM和SPA挑选5, 10, 20, 30, 40和50个特征, GA和BRS-SVM无法抽取固定的特征个数。 将试验集随机分为80%训练集和20%验证集, 使用挑选出来的特征训练SVM和LR雌雄分类模型, 计算验证集准确率, 重复上述50次, 得到平均验证集准确率如图5所示, 其中MBR-SVM-SVM表示使用MBR-SVM挑选特征, 再使用SVM建模, 同理可得其他图例含意。 使用同种特征选择的方法挑选特征, 再使用SVM模型建模的准确率比LR模型准确率高。 挑选5个特征, BRS-SVM-SVM验证集准确率为93.88%, GA-SVM验证集准确率为89.24%, 而其他特征选择方法只有80%~82%。 BRS-SVM的性能要优于GA-SVM, 而GA-SVM的性能要优于其他算法。
用特征选择方法在试验集中挑选特征, 得到的特征再用测试集建立分类模型, 测试集准确率如图5所示。 用测试集900~1 399 nm波段建立SVM雌雄分类模型准确率为95.70%, 建立LR雌雄分类模型准确率为95.54%。 用BRS-SVM挑选5个特征使用SVM建模准确率为89.56%, 其余准确率大多在86%~87%, SVM建模的准确率比LR的高, 当挑选大于9个特征个数时, RFE-SVM, GA-SVM和BRS-SVM性能接近, 用BRS-SVM挑选27个特征SVM建模准确率为94.97%, 和使用900~1 399 nm波段建模准确率接近。 通过上述实验, 证明挑选单波段特征时我们的方法要优于其他方法, 尤其是挑选特征数量较少的情况下。
计算试验集900~1 399 nm波段内的面积特征, 如900 nm需要计算900, 900~901和900~902 nm等15个连续波段的面积, 1 385~1 399 nm范围向1 400 nm后面的波段计算, 共获取7 500个新的特征, 再使用MBR* SVM, MBR-LR, REF-SVM, SPA, GA和BRS-SVM挑选连续波段的面积特征, 其中MBR-SVM, MBR-LR, REF-SVM和SPA分别挑选5, 10, 20, 30, 40和50个特征, 验证集准确率如图6所示, 测试集准确率如图6所示。 用BRS-SVM挑选5个特征再用SVM建模, 验证集准确率为94.17%, 测试集准确率为91.95%。 用REF-SVM挑选5个特征再用SVM建模, 验证集准确率为86.30%, 测试集准确率为85.91%, 用GA挑选5个特征再用SVM建模, 验证集准确率为89.30%, 测试集准确率为86.66%, 在总特征数量较多且挑选少量特征的情况下, 我们提出的MBR-SVM要优于REF-SVM和GA, 在挑选特征数量大于等于20个时, REF-SVM的性能和MBR-SVM, GA相同。
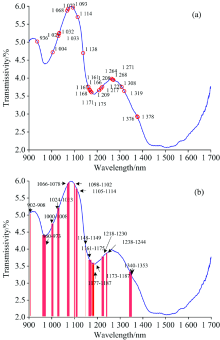
图7(a)为用BRS-SVM挑选的27个单波段特征, 用这些特征建立SVM雌雄分类模型测试集准确率为94.97%。 图7(b)为用BRS-SVM挑选的14个连续波段面积特征, 用SVM建模测试集准确率为94.43%, 可用13个LED灯替代近红外光谱。 可以根据实际生产需求选择合适的特征, 成本较低准确率要求不高, 可选择挑选连续波段面积的特征, 如用BRS-SVM挑选的5个连续波段面积特征, 再用SVM建模测试集准确率为91.95%, 可用5个LED灯替代近红外光谱。
为了进一步验证挑选的特征的有效性, 我们用SW2540型便携式光纤光谱仪采集112个932品种蚕茧的漫透射光谱和77个7xia品种蚕茧的漫透射光谱。 用BRS-SVM挑选的27个单波段特征和14个连续波段面积特征建立SVM雌雄分类模型, 准确率如表2所示。 932品种的分类模型效果差些, 这是因为不同光谱仪或者不同品种的蚕茧采集的近红外光谱存在着差异。
| 表2 932和7xia品种蚕茧的SVM雌雄分类模型准确率 Table 2 Accuracy of SVM sex classification model for silkworm cocoons of 932 and 7xia |
提出了一种包裹式的特征选择方法, 基于支持向量机的自助重加权采样(BRS-SVM)的特征选择方法, 分别对蚕茧近红外光谱单波段特征和连续波段特征进行选择, 建立有效的雌雄分类模型。 BRS-SVM与其他特征选择方法相比性能均有一定优化, 特别是在挑选少量特征时模型精度最高。 在需求为低成本和低精度的情况下, 挑选5个单波段特征, 测试集准确率为89.56%, 在需求为高精度的情况下, 挑选14个连续波段面积特征, 测试集准确率为94.97%。 首次结合化学计量法分析蚕茧的近红外光谱, 为蚕茧的雌雄检测应用提供一种实用的解决方案。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|