作者简介: 孙志兴, 1998年生, 江南大学轻工过程先进控制教育部重点实验室硕士研究生 e-mail: zhixingsun@stu.jiangnan.edu.cn
近红外光谱中包含了物质中有机分子含氢基团的特征信息, 具有维度高、 冗余大等特点。 传统的基于浅层校正模型, 比如主成分回归、 偏最小二乘回归、 人工神经网络、 支持向量回归等, 无法提取近红外光谱数据深层的信息。 提出一种基于堆叠监督自动编码器的近红外光谱建模方法, 不仅可以拟合光谱数据与理化值之间复杂的非线性关系, 还可以提取数据深层的特征信息。 首先通过对比不同的光谱预处理对模型预测结果的影响, 选择最优的预处理方法, 然后再使用相关系数法提取特征波段。 将处理好的近红外光谱数据作为堆叠监督自动编码器的输入信号, 利用理化值对多个监督自动编码器进行有监督的预训练; 将多个经过预训练的监督自动编码器进行堆叠, 得到堆叠监督自动编码器; 将预训练的参数作为堆叠监督自动编码器的初始化参数, 然后再利用理化值对堆叠监督自动编码器进行有监督的微调, 最后得到模型的最优参数。 分别利用玉米含水量和黄酒总酸含量等近红外数据集进行验证, 建立了偏最小二乘回归预测模型、 人工神经网络预测模型、 堆叠自动编码器预测模型和堆叠监督自动编码器预测模型, 验证了堆叠监督自动编码器建模的可行性; 以预测均方根误差和预测相对分析误差两个指标对比分析了偏最小二乘回归、 反向传播人工神经网络、 堆叠自动编码器及堆叠监督自动编码器四种建模方法的评价指标。 分析结果表明, 采用该方法建立的模型, 模型预测效果更好, 玉米含水量数据集的两个评价指标达到了0.060 4和4.313; 黄酒总酸含量数据的两个评价指标达到了0.120和4.227, 均优于另外三种方法。
The near-infrared spectrum contains the characteristic information of the hydrogen-containing groups of organic molecules in the substance, and it has the characteristics of high dimensionality and large redundancy. Traditional near-infrared spectroscopy techniques are based on shallow correction models, such as principal component regression, partial least squares regression, artificial neural networks, support vector regression etc., which cannot extract the deep information of the spectral data. This paper proposes a near-infrared spectroscopy modeling method based on stacked supervised autoencoders, which can fit the complex non-linear relationship between spectral data and target physicochemical values and extract the deep feature information of the data. First, the optimal preprocessing method is selected by comparing the effects of different spectral preprocessing on the model prediction results. Then the correlation coefficient method is used to extract the characteristic bands of the preprocessed spectrum. The method uses the processed near-infrared spectrum data as the input signal. Then use the target physicochemical values to perform supervised pre-training on multiple supervised autoencoders, and stack multiple pre-trained supervised autoencoders. The stacked supervised autoencoder is obtained, the pre-trained parameters are used as the initialization parameters of the stacked supervised autoencoder, and then the target physicochemical values are used to supervise and fine-tune the stacked supervised autoencoder. Finally the optimal parameters of the model are obtained. Established partial least squares regression prediction model, artificial neural networks prediction model, stack auto-encoder prediction model and stack supervised auto-encoder prediction model on the corn water content data and the total acid content data of yellow wine respectively, verifying the feasibility of stack supervised auto-encoder modeling. The root means square error and residual prediction deviation are employed to evaluate model performance. The accuracy of four modeling methods of partial least squares regression, backpropagation- artificial neural networks, stack auto-encoder, and stack supervised auto-encoder are compared and analyzed. The analysis results show that the model established by stack supervised auto-encoder has a good prediction effect. The two evaluation indexes of the corn water content data set reached 0.061 1 and 4.271; the two evaluation indexes of rice wine’s total acid content data reached 0.126 6 and 4.006, excellent for the other three methods.
近红外光是波长介于可见光区与中红外区之间的电磁波, 它的波长范围约为800~2 500 nm。 近红外光谱的谱区与有机分子的含氢基团, 如C— H, N— H, O— H等的合频和不同倍频吸收区一致, 所以近红外光谱中包含了有机分子含氢基团的主要结构信息[1]。 通过对光谱进行分析, 可以得到物质中的特定成分的含量, 能够实现无损检测。 近年来, 在石油化工、 农业、 发酵等领域近红外光谱分析得到广泛的应用。
在使用近红外光谱进行检测时, 首先需要建立光谱信息与目标理化值的校正模型。 常用建模方法有多元线性回归(multiple linear regression, MLR)[2]、 主成分回归(principle component regression, PCR)[3]、 偏最小二乘回归(partial least squares regression, PLSR)[4]、 人工神经网络(artificial neural networks, ANN)[5]、 支持向量回归(support vector regression, SVR)等。 其中, PLSR是应用较广泛的线性建模方法之一。 ANN可以建立光谱的非线性校正模型, 但是易陷入局部最优解。 支持向量回归是一种机器学习的方法, 它有较好的泛化能力。 但是SVR中使用的核函数以及核函数参数没有选择标准。
近年来, 作为一种智能学习算法, 深度学习逐渐在图像处理、 语音处理以及故障检测等领域受到重视[6, 7, 8, 9]。 鉴于在数据非线性强、 维度高、 数据量大的情况下特征提取的良好效果, 目前深度学习也开始被引入到近红外光谱分析中[10]。 Zhang等使用堆栈稀疏自编码器融合核极限学习机对药品的质量进行鉴别[11]; Lu等用堆叠降噪自编码器与随机森林结合进行植物黄龙病鉴别[12]; Yang等使用深度信念网络对药品质量进行无损伤的鉴别[13]。 上述工作中使用的深度学习算法在预训练时都采用无监督训练方式。 无监督的训练没有理化值的指导, 所以提取的特征信息中会包含很多与理化值无关的信息, 这部分信息对理化值的预测是没有帮助的, 甚至会降低模型的预测精度。
堆叠监督自动编码器(stack supervised auto-encoder, SSAE)是深度学习算法的一种, 它具有多个隐含层, 可以通过有监督的学习方式实现复杂非线性函数的逼近。 使用堆叠监督自动编码器建模有以下优点: (1)深层网络的复杂多层结构可以使其更好的拟合变量之间的非线性关系; (2)堆叠监督自动编码器预训练和微调均采用有监督的方式, 能够更好的提取光谱数据中与理化值相关的特征信息并建立预测模型; (3)可以更加快速有效地处理大数据; (4)通过预训练初始化全局网络的参数, 能够有效的避免网络在训练过程中出现梯度消失或者陷入局部最优解的问题。
近红外光谱数据具有维度高, 重叠性高, 信噪比低等问题, 而堆叠监督自动编码器可以在数据维度高, 信噪比差的情况下表现出良好的特征提取和分析能力, 同时还可以处理非线性关系。 故利用堆叠监督自动编码器对近红外光谱数据进行建模, 分别对玉米含水量数据和黄酒发酵过程总酸含量数据建立SSAE预测模型, 并引入预测均方根误差(RMSEP)和预测相对分析误差(RPD)对模型进行评价。 玉米含水量数据集的两个评价指标达到了0.060 4和4.313; 黄酒总酸含量数据的两个评价指标达到了0.120和4.227。 进一步建立了PLSR预测模型、 误差反向传播人工神经网络(back propagation, BP)预测模型、 堆叠自动编码器(stack auto-encoder, SAE)预测模型与SSAE进行对比, 验证该方法的优越性。
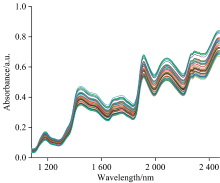
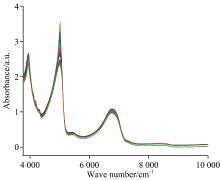
首先选用玉米近红外光谱数据集, 该数据集可在EVRI网站上免费获取http://eigenvector.com/data/Corn, 原始数据如图1所示。 该数据集是由m5, mp5和mp6三种不同的近红外光谱仪对80个不同的玉米样本扫描构成的。 光谱的波长范围为1 100~2 498 nm, 分辨率为2 nm, 每条光谱含有700个数据点。 该数据集给出了玉米的水分、 淀粉、 油脂的含量(百分比)作为目标理化值。 在此采用m5光谱仪的80组光谱数据以及对应的水分含量进行建模。 而在黄酒发酵的应用中, 我们对黄酒发酵周期的12个关键时间点进行取样, 每个样本扫描4次, 并且进行两个批次的黄酒发酵, 每个批次获取48个样本, 总共获得96个样本。 采集的黄酒样本的近红外光谱数据通过美国Thermo Antaris MX傅里叶变换型近红外分析仪扫描获得, 光谱仪的参数为: 光谱波长范围10 000~4 000 cm-1, 分辨率为8 cm-1 [14]。 原始光谱数据如图2所示。
两个数据集的训练集与测试集都采用SPXY(sample set partitioning based on joint x-y distances)法进行划分, SPXY方法能使训练集的数据在X空间和Y空间均匀分布[15]。 本工作按照3∶ 1进行训练集与测试集的划分, 第一个玉米数据集得到60个训练集和20个测试集。 第二个黄酒数据集得到含有72组数据的训练集和24组数据的测试集。
1.2.1 监督自动编码器
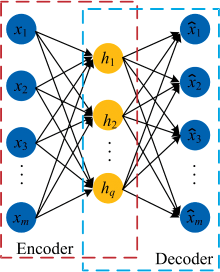
自动编码器是监督自动编码器的基础, 它是一个无监督的三层神经网络, 结构如图3所示。 它由输入层、 隐含层和输出层构成, 包含一个编码器和一个解码器。 通过对自动编码器无监督的训练, 能够实现对输入数据的特征提取和数据降维[16]。 由于近红外光谱数据具有维度高、 冗余大、 样本量多等问题。 而传统的特征提取方法, 如主成分分析, 所提取的特征本质上就是原始变量的线性组合, 所以无法包含非线性特征信息。 而作为神经网络的一种, 自动编码器可以很好的建立非线性特征提取模型, 并且它可以处理维度高、 样本量多的数据, 还可以提取近红外光谱数据中的非线性特征信息。 具体的过程如下:
假设自动编码器输入m维的光谱数据样本, x=[x1, x2, …, xm]T∈ Rm, 由编码器可以得到隐含层的输出, 也就是特征表示为h=[h1, h2, …, hq]T∈ Rq,
式(1)中, We∈ Rq× m为权重矩阵, be∈ Rq为偏置向量, f(· )为激活函数, 这里的激活函数选用的是tanh函数(即tanh(x)=(ex-e-x)/(ex+e-x))。
由编码器获得隐含层特征表示之后, 解码器将隐含层的特征表示进行重构, 即
式(2)中, Wd∈ Rm× q为权重矩阵, bd∈ Rm为偏置向量, g(· )为激活函数, 这里的激活函数选用的是线性激活函数(即g(x)=x),
假设输入光谱数据为X={x1, x2, …, xn}∈ Rm× n其中n为光谱样本个数, m为光谱数据的维数。 重构输出为
式(3)中, xi∈ {x1, x2, …, xn},
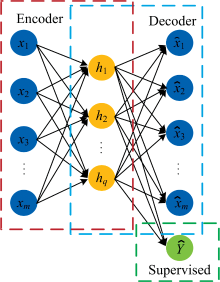
自动编码器通过无监督的训练方式, 可以根据输入与重构输出之间误差来调整参数, 从而得到输入光谱数据的特征表示。 但是在建立近红外光谱定量分析模型时, 无监督的训练机制会使自动编码器的隐含层特征输出包含许多与理化值无关的信息, 使模型的预测性能下降, 不利于模型对理化值的预测。 为了解决这个问题, 我们引入了监督自动编码器用以提取与目标理化值高度相关的特征信息[17]。 监督自动编码器是在普通的自动编码器的基础上加了一个监督层, 监督层是与隐含层全相连接的, 具体的结构如图4所示。 这样可以使隐含层的特征信息不仅仅能精确的重构出输入数据, 还能够很好的预测理化值, 使得特征信息中含有与理化值高度相关的信息。
监督层的值为y=[y1, y2, …, yp]T∈ Rp, 监督层与隐含层之间的关系为
式(4)中,
假设监督层的实际值为Y={y1, y2, …, yn}∈ Rp× n, 输入层的光谱数据为X={x1, x2, …, xn}∈ Rm× n其中n为光谱样本个数, m为变量数, p为监督层的变量数。 重构输出为
式(5)中,
监督自动编码器的损失函数Loss为重构损失和预测损失的加权和
监督自动编码器在原自动编码器的基础上加了一个监督层后, 可以将重构损失与预测损失结合, 使其不仅可以提取数据底层结构, 又可以提供准确的预测性能[17]。 所以监督自动编码器可以有效地解决所提取的光谱特征信息与理化值之间相关性弱的问题, 获得更好的光谱数据特征表示, 提高近红外光谱定量分析的性能。
1.2.2 堆叠监督自动编码器
单个监督自动编码器只是一个简单的、 浅层的神经网络, 无法提取深层次的特征信息。 为了提取近红外光谱深层次的特征信息, 可以将其扩展到深层结构-堆叠监督自动编码器[16]。 结构如图5所示。 图5(a)为堆叠监督自动编码器, 它是由多个监督自动编码器构成。 图5(b)为构成图5(a)的多个监督自动编码器。
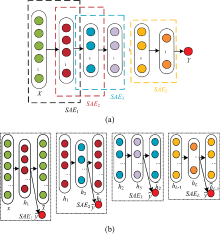 | 图5 堆叠监督自动编码器的结构图 (a): 堆叠监督自动编码器; (b): 监督自动编码器Fig.5 Structure of stack supervised auto-encoder (a): Stack supervised AE; (b): Supervised AE |
堆叠监督自动编码器的训练分为两步: 首先是有监督的预训练, 然后是有监督的微调。 预训练阶段: 首先最小化第一个监督自动编码器的损失函数, 将第一个监督自动编码器获得的隐含层作为下一个监督自动编码器的输入层, 这样一直训练到最后一个监督自动编码器, 用训练得到的权重和偏置来初始化相应的全局网络。 微调阶段: 使用预训练得到的相应的参数进行初始化, 然后使用梯度下降法微调所有层的参数。 训练的过程如图6所示。
1.2.3 基于堆叠监督自动编码器的建模过程
基于堆叠监督自动编码器的近红外光谱数据建模的具体步骤如下:
① 采集近红外光谱数据与对应的理化值;
② 剔除其中的异常值;
③ 将数据分为训练集和测试集, 并分别用同样的方法对光谱进行预处理;
④ 用训练集进行堆叠监督自动编码器的训练;
⑤ 用测试集进行模型的测试和评价。
算法的运行环境为: Intel(R) Core(TM) i5-7500 CPU @3.40 GHz CPU; 计算机内存为8 GB。 所使用的为Pycharm, 系统环境中配置了tensorflow, numpy, pandas等Python运算库。
原始光谱中除了包含对目标理化值预测有用的信息之外, 还有包含许多与目标理化值无关的冗余信息和测量噪声, 如电噪声、 杂散光和漂移等, 所以对原始光谱进行合适的预处理是十分必要的。 预处理的方法有一阶差分、 二阶差分、 Savitzky-Golay平滑(SG)、 多元散射矫正(MSC)、 标准正态变量变换(SNV), 在无预处理和不同方法预处理之后的光谱, 分别采用PLSR, BP, SAE和SSAE建立预测模型, 结果表1所示。
| 表1 基于不同预处理的预测效果 Table 1 Prediction results based on different pretreatment methods |
由表1可知, 光谱预处理方法对模型的精度有明显的影响, 而且不同的光谱数据和不同的建模方法使用的预处理方法也不同。 对于玉米数据集, 四种建模方法与一阶差分预处理的组合均是最好的; 黄酒数据集在使用PLSR和BP建模时, SG平滑处理的效果最好, 而SAE和SSAE建模时, 则是不进行处理的效果最好。 因此玉米数据集的四种建模方法对比时采用一阶差分预处理后的数据进行建模, 而对于黄酒数据集, PLSR和BP建模使用SG平滑预处理光谱, SAE和SSAE建模则使用原始光谱。
特征波段的选择使用的是相关系数法。 它是通过计算每一个波长的吸光度与目标理化值之间的相关系数, 选择相关系数大的波长作为特征波段。
对预处理后的近红外光谱使用相关系数法选择特征波长。 玉米数据集最终选择了425个特征波长; 黄酒数据集最终选择了179个特征波长。
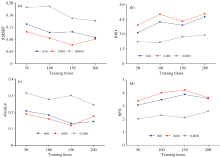
在堆叠监督自动编码器中, 模型参数的选择对预测精度有重要的影响。 在此主要考虑训练次数和学习率。 模型的层数和每层的节点数, 两个数据集分别采用的结构为425-132-52-6-1和179-50-10-1。 训练次数和学习率的选择分别如图7所示, 训练的次数过多会导致模型过拟合, 而训练次数太少模型会有较大的损失, 预测精度较差。 从图7(a)和(b), 可知玉米数据集在学习率为0.001, 训练次数为150次时, 模型的精度最高, RMSE=0.060 4, RPD=4.313; 从图7(c)和(d), 可知黄酒数据集在学习率为0.001, 训练次数为150次时, 模型的精度最高, RMSE=0.120, RPD=4.227。
为验证堆叠监督自动编码器建模方法的可行性, 对玉米数据和黄酒发酵数据仿真建模, 与PLSR, BP和SAE进行对比, 以说明本方法的优越性。
2.4.1 玉米数据集
表2为不同建模方法下, 玉米含水量的预测结果。 使用SSAE方法建立模型时, 可以通过有监督的形式提取光谱数据的深层信息, 两个评价指标均优于另外三种方法, RPD达到了4.313。
| 表2 玉米数据使用不同建模方法 Table 2 Prediction results of corn data sets using different modeling methods |
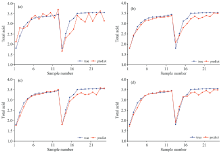
图8为采用不同建模方法, 玉米含水量的预测结果与真实理化值的对比。 横坐标为预测样品数量。 图8(a)可以看出基于PLSR模型的真实值和预测值之间还有较大的差距。 图8(b)为BP神经网络的效果, 从图中可以看出预测值与真实值也有一定的差距, 但是预测值的变化趋势与真实值基本相同。 图8(c)为SAE方法的结果, 真实值与预测值之间差距进一步减小。 图8(d)为本工作提出的SSAE的建模方法, 结合了有自动编码器和监督学习的优点, 使得预测效果最佳。
 | 图8 玉米数据不同建模方法预测效果对比 (a): PLSR模型; (b): BP模型; (c): SAE模型; (d): SSAE模型Fig.8 Prediction results of different modeling methods for corn data (a): PLSR model; (b): BP model; (c): SAE model; (d): SSAE model |
2.4.2 黄酒发酵实例
表3为黄酒发酵过程中总酸含量的预测结果。 在使用SSAE方法进行建模时, 黄酒数据集的RPD达到了4.227, 相对于PLSR方法, 黄酒数据的RPD提升了2倍。 并且两个评价指标均优于另外三种方法。
| 表3 黄酒数据使用不同建模方法 Table 3 Prediction results of yellow wine data sets using different modeling methods |
图9为黄酒总酸含量在采用不同方法下的预测结果对比。 图9(a)可知, 基于PLSR模型的预测值与真实值之间相差较大。 图9(b)为基于BP神经网络模型的效果, 模型的预测值与真实值之间的趋势大致相同, 在数值上还有一定的差距。 图9(c)和图9(d)分别是基于SAE和SSAE方法建模的效果, SAE方法能够提取数据深层的特征, 预测效果相比于BP神经网络有所提升。 SSAE在提取数据深层特征的同时加上了监督信息, 使得最终的预测效果最佳。
提出一种基于堆叠监督自动编码器的近红外光谱建模方法, 用于光谱的定量分析。 堆叠监督自动编码器(SSAE)具有多层结构, 在堆叠自动编码器(SAE)的结构基础上加入了监督层, 使得SSAE的预训练与微调均采用有监督的训练方式。 该方法不仅能够提取输入数据的深层特征信息, 而且还能建立有效预测模型。 通过对实际黄酒发酵数据集和玉米含水量数据集的应用, 证明了该方法比传统PLSR、 BP神经网络和SAE具有更好的预测性能。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|