作者简介: 韩 宇, 1996年生, 长春理工大学理学院硕士研究生 e-mail: 694164228@qq.com
大豆在生长过程中因病害影响其产量会急剧下降, 如果不及时判别出病害种类, 喷洒相关农药, 病害严重的大豆甚至会绝产。 及时判别病害种类进行合理施药, 阻止病害进一步发展是保证大豆安全生产的重要环节。 目前, 基于大豆植株细菌性病害的病原菌鉴定和聚合酶链式反应(polymerase chain reaction, PCR)的鉴定方法, 最短需要两天时间, 因此, 快速检测大豆病害种类的方法成为该作物, 也是建立智慧农业生产的关键环节之一。 应用拉曼光谱快速检测技术诊断大豆病害, 构建N-乙酰胞壁酸分子空间结构, 采用密度泛函理论通过利用B3LYP/6-31+(d, p)基组优化大豆细菌性病害标志物N-乙酰胞壁酸的分子结构计算其拉曼光谱, 并进行理论因子校正, 校正因子为0.985 7; 采用微区三级拉曼光谱技术探测该标志物N-乙酰胞壁酸的拉曼光谱, 采用平滑、 去基线、 截取波数范围等过程进行光谱预处理; 在理论和实验对比分析的基础上, 指认大豆测试和计算的拉曼光谱对应的特征峰, 峰值波数相差大多在0~10 cm-1, 实验数据与理论计算结果基本一致, 判定了振动拉曼光谱的特征峰及其对应的分子结构的关系。 结果表明: 大豆细菌性病害标志物N-乙酰胞壁酸分子在200~1 650 cm-1范围内含15个特征峰, 较强峰值和振动归属分别为229.0 cm-1的甲基摇摆振动和764.0 cm-1环内的摇摆呼吸振动等, 给出了键长、 键角和二面角等15个振动峰的空间结构参数, 指证了N-乙酰胞壁酸分子的特征结构。 结果也证明了可通过多种生物分子的大豆拉曼光谱测量, 筛选细菌性病害标志物N-乙酰胞壁酸分子的拉曼光谱, 能够有效识别细菌性病害。 智慧农业生产中利用拉曼光谱快速检测技术, 是农作物病害检测诊断的一种有效方法, 若结合应用机器学习方法与光谱分析识别, 以快速、 准确和便捷的方式为智慧农业的健康生产及保驾护航发挥效用, 是推进我国农业发展的重要环节。
The yield of soybean will drop dramatically due to disease during its growth. If the disease is not identified in time and no corresponding pesticides are sprayed, severely diseased soybeans can even be wiped out. It is very important to identify the disease species and apply the insecticide rationally to prevent the further development. Currently, it will take two days to make the pathogenic and polymerase chain reaction (PCR) identification of soybean bacterial diseases. Therefore, the method of quickly detecting the types of soybean diseases has become one of the key links in the intelligent agricultural production of this crop. Raman spectroscopy is used to rapidly diagnose soybean diseases. The molecular space structure of N-acetylmuramic acid is constructed, density functional theory (DFT) with B3LYP/6-31+(d,p) basis set was used to do the theoretical calculation. Through theoretically calculating the Raman spectra of soybean bacterial spot disease marker N-acetylmuramic acid, the characteristic peaks of the vibrational Raman spectra and their corresponding molecular structures of N-acetylmuramic acid are identified. The calculated Raman spectra should be corrected using the correction factor, and the correction factor is 0.985 7. In addition, the experimental Raman spectra of N-acetylmuramic acid are obtained using micro-zone three Grade Raman spectroscopy technology. The process of smoothing, baselines removal and wavenumber range interception was used to preprocess the spectra. The comparative analysis of theoretical and experimental results determines the characteristic peaks of vibrational Raman spectra and the corresponding molecular structures. The peak wavenumber difference is mostly 0~10 cm-1. The experimental data is consistent with the theoretical calculation results. The results show that the N-acetylmuramic acid molecule, a marker of bacterial spot in soybean, contains 15 characteristic peaks in the range of 200 to 1 650 cm-1, which can be used as a diagnostic basis. The main peak assignment at 229 and 763 cm-1 were attributed to the methyl swing vibration and ring breathing vibration. The spatial structure parameters of 15 vibration peaks such as bond length, bond angle and dihedral angle are given to identify the structure of the N-acetylmuramic acid molecule. The results also proved that the Raman spectroscopy of soybean with a variety of biomolecules could be used to screen the Raman spectroscopy of N-acetylmuramic acid, and it could effectively identify bacterial disease. Raman spectroscopy rapid detection technology is a new method for soybean disease detection and diagnosis, which plays a part in protecting healthy products in the field of intelligent agriculture. The results should be better combine with machine learning methods in spectral analysis and identification. Exploring a fast, accurate and convenient method could obtain a lot of benefits in intelligent agriculture, which plays a vital role in promoting the development of agriculture in China.
大豆是我国主要的经济农作物之一, 大部分地区在其生长的过程中都可能存在霜霉病害, 蚜虫病虫害和细菌性病害三种常见的大豆病虫害。 其中细菌性病害最为常见, 这种病害可细致地分为以下三种: 第一种是由丁香假单胞菌病原菌引起的大豆细菌性斑点病[1, 2]; 第二种是大豆细菌性斑疹病, 区别于斑点病致病变种, 该致病变种为油菜黄单胞菌病原菌; 第三种是由丁香假单胞菌烟草病原菌引发的大豆细菌性野火病[3, 4]。 传统检测病害有Biolog测定和脂肪酸测定(FAME)两种方式。 Biolog测定方法的步骤为: 在含有95个碳源的Biolog阴性微孔板中, 将每个孔板中均加入适量的所测细菌悬浮液, 在30 ℃的环境下培养24 h后, 使用Biolog读数机检测出其结果。 脂肪酸测定(FAME)把纯化的参试菌株, 在NA培养基上, 以28 ℃的生长环境培养24 h之后, 转入到含有3%胰蛋白酶地TSA固体培养基上培养24 h, 然后使用专化的微生物鉴定系统软件检测出最终的鉴定结果。 这两种检测方式存在一定的不足, 最短需要耗时2 d, 检测时间较长, 操作不便捷, 无法做到快速检测。
鉴于存在于大豆细菌性病害的细胞壁中的N-乙酰胞壁酸分子, 是区别真菌与病虫害的标志物。 N-乙酰胞壁酸分子式为C11H19NO8, 主要由C, H, N, O元素构成。 基于光谱技术检测农作物可针对其标志物进行检测判定, 该技术检测可以做到无损害, 无破坏, 并且具有鉴定速度快操作方便的优势, 因此在近几年应用广泛。 Zhao等[5]使用光谱技术检测叶片是否健康和是否缺锌的问题, 使用共聚焦拉曼光谱仪检测能够达到100%的精确度。 Weselucha Birczynska等[6]运用共振拉曼显微光谱进行试验, 最终分析出类胡萝卜素浓度与干旱胁迫之间存在一定的联系。 Martins等[7]结合拉曼光谱仪和红外光谱仪对鸵鸟油的振动带进行了光谱分析, 根据实验和理论计算证明了羧基的不同强度振动模式。 陈玉锋等[8]采用了密度泛函理论中的B3LYP杂化泛函, 理论计算出了2-巯基-5-硝基苯并咪唑分子拉曼峰值, 测得的实验数据与分析得出的吸收波长较为一致。 Yeturu等[9]采用手持式拉曼光谱仪测得花叶病毒枫叶和健康叶片的拉曼光谱, 实验结果表明能够早期鉴定枫叶黄色花叶病。 综上所述传统的大豆细菌性病害鉴定耗时较长, 新型的光谱鉴定会缩短时间, 并且检测方法会更简便, 有助于做到大豆细菌性病害的早期治疗。
理论计算使用Gaussian09量子化学软件基于密度泛函理论方法下的B3LYP/6-31+(d, p)基组, 优化N-乙酰胞壁酸分子的空间几何结构, 计算理论拉曼光谱, 其中N-乙酰胞壁酸分子构型用Gauss View5.0构造, 采用DFT方法对其空间结构进行了优化, 取修正因子(0.985 7)进行校正[10, 11], 优化构型及结果如图1, 同时在表1中给出了N-乙酰胞壁酸分子优化后的空间几何参数。
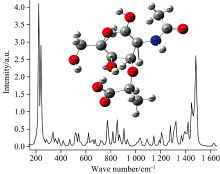 | 图1 优化后的N-乙酰胞壁酸分子构型与理论拉曼光谱Fig.1 The optimized molecular configuration and theoretical Raman spectrum of N-acetylmuramic acid |
| 表1 N-乙酰胞壁酸优化后的几何参数 Table 1 Optimized geometrical parameters of N-acetylmuramic acid |
实验设备为TriVistaTM555CRS三级显微拉曼光谱检测仪, CCD探测器使用时液氮制冷温度为-120 ℃, 设备分辨率为0.5 cm-1。 将检测的样品放置在显微镜载物台上, 置于21 ℃恒温的暗室中进行拉曼光谱检测。 实验样品为纯培养的大豆细菌性病原菌株(Pseudomonas syringae pv. glycinea, PSG), 由吉林农业大学植物保护学院提供。 N-乙酰胞壁酸化学标准品(chemical reference ubstance, CRS)(含量> 98%), 样品来自阿拉丁试剂(上海)有限公司。
拉曼光谱检测参数如下: 实验中以波长532 nm的激光作为激发光, 积分时间为5 s, 积分三次取平均。 扫描范围200~1 650 cm-1。 待测样品载于玻片上, 采集拉曼信号。 当光谱测量结束后, 重新取样品进行三次以上测量, 以避免长时间的激光照射导致分子产生光解作用, 将多次测得的拉曼光谱进行平均, 减小光谱数据误差。 采取相同的实验方式测量标准品N-乙酰胞壁酸的拉曼光谱。
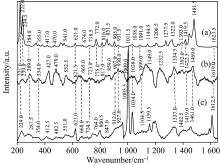
将试验测量的谱图进行预处理, 由于拉曼光谱会受到检测样品、 实验环境等的影响, 而产生干扰, 为了获取有效的光谱信息, 选择200~1 650 cm-1范围去除噪声、 荧光背景、 基线校准和平滑处理, N-乙酰胞壁酸的CRS与PSG分别见图2(b和c), 结合理论计算光谱图2(a)进行对比分析。
通过Gaussian View5.0观察理论光谱各谱峰的振动形式, 对其振动模式归属进行认证, 结果归纳到了表2。 表2中理论测得拉曼光谱在第一列, 分析纯实验拉曼光谱在第二列, 大豆细菌性病害病原菌株PSG实验测得拉曼峰值在第三列并给出了其峰值拟合后的半峰全宽, 每个谱峰所属的振动模式在第四列中给出。
| 表2 N-乙酰胞壁酸实验与理论振动频率(cm-1)与归属 Table 2 Experimental and theoretical vibration frequency (cm-1) and attribution of N-acetylmuramic acid |
从图2中可以分析出大豆细菌性病害病原菌株PSG标志物N-乙酰胞壁酸实验拉曼光谱在200~1 650 cm-1范围内有明显的拉曼振动峰分别为229.0, 287.5, 354.0, 621.0, 668.0, 724.5, 847.0, 912.0, 927.0, 1 005.5, 1 034.0, 1 104.5, 1 419.0, 1 461.0和1 612.5 cm-1。 实验测到的拉曼光谱数量多且各个峰强对比显著。 经过实验与理论拉曼光谱比较得出, 大部分的光谱线型和振动频率都基本一致。 例如229.0 cm-1振动归属为1C— H, 2C— H的摇摆振动, 跟理论拉曼光谱222.0 cm-1较为一致。
通过对大豆细菌性病害病原菌的拉曼峰值振动归属指认, 发现与理论峰值较为吻合, 但其中有个别峰值出现了频移。 从原始光谱谱线可以看出, 部分峰值可能被多个峰值叠加, 为了能更加直观看到频移和线宽变化, 对实验数据进行了福格特函数峰值拟合, 如图3所示为大豆细菌性病害病原菌拉曼光谱部分叠加峰值拟合。
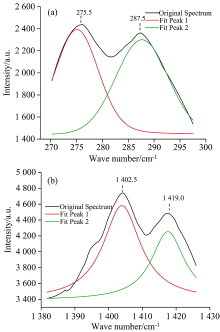 | 图3 大豆细菌性病害病原菌拉曼光谱部分叠加峰值拟合Fig.3 Fitting of partial superimposed peaks of Raman spectra of pathogens of soybean bacterial diseases |
分析其结果表明实验光谱和理论光谱个别谱峰有较小的差异, 主要存在两种原因: (1)在量子化学的计算方法基础之上, 在处理几何平衡结构的时候, 对力常数估计值存在微小的偏差; (2)处于分子内部的官能团受到了其他相邻分子影响小于分子边缘部分的官能团受到的影响, 前者拉曼频移的谱峰也就受到的影响较小, 也就更加接近于理论计算值。
拉曼光谱检测技术诊断大豆细菌性病害的方式, 区别于传统的生化检测技术, 具备简洁而快速的优势。 N-乙酰胞壁酸分子存在于大豆细菌性病害表面细菌主体中的肽聚糖的细, 构建其分子构型。 在光谱理论中, 基于密度泛函理论, 采取量子化学软件计算出该分子的拉曼光谱; 在微区三级拉曼光谱的测试中, 获得了200~1 650 cm-1范围的N-乙酰胞壁酸分子的拉曼光谱, 在校正后, 与理论结果基本一致, 波数峰值相差大多在0~10 cm-1范围内; 经过对比分析实验与理论计算的结果, 确定了主要官能团对应的振动模式的归属, 并给出了N-乙酰胞壁酸分子的二面角、 键角、 键长和空间结构参数。 该工作对研究N-乙酰胞壁酸分子结构以及大豆细菌性病害的振动光谱检测提供了基础数据, 促进了拉曼光谱技术在农作物检测诊断中的应用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|