作者简介: 郑开逸, 1983年生, 江苏大学食品与生物工程学院副研究员 e-mail: kaiyizhengjsu@126.com
在近红外光谱分析中, 将近红外光谱和浓度信息建立统计模型, 通过光谱代入模型即可预测未知样本浓度。 但是, 检测条件的变化会导致光谱的改变, 进而导致原有的模型不能准确预测光谱改变后的样本。 对此, 模型转移可以通过校正新测量的光谱(从光谱), 使得从光谱能够被原有光谱(主光谱)建立的模型准确预测。 模型转移可以使用全光谱进行校正, 但是全光谱中往往包括噪声、 背景等干扰信息, 这些干扰会增加预测误差。 故可以使用变量选择方法找出光谱中有化学意义的信息来模型转移。 但是一般的变量选择算法只选择主光谱的区间, 从光谱使用主光谱相同的波长区间模型转移。 但是在实际工作中, 主光谱和从光谱有化学意义的区间往往不一致, 主从光谱使用同一区间模型转移会增加误差; 此外, 有时二者原光谱的波长范围并不一致, 从主光谱选出的区间不能用于从光谱的校正。 对此, 提出了基于双光谱区间遗传算法(GA-IDS), 同时选择主光谱和从光谱有化学意义的区间, 进而实现模型转移。 GA-IDS算法步骤包括, ①随机产生种群; ②分析种群中每条染色体, 删去错误染色体; ③根据每条染色体, 找出其相应的主光谱和从光谱波段组合, 并计算其模型转移后的验证均方根误差(RMSEV); ④按照概率, 执行选择、 交叉、 变异操作。 在一次迭代结束之后, 返回到步骤②, 重新执行纠错、 计算RMSEV、 选择、 交叉、 变异。 达到停止迭代的要求后, 将最低的RMSEV值所对应的染色体保存下来作为最优染色体, 其所对应的主从光谱区间作为最优区间。 用玉米、 小麦两套数据测试了该算法, 结果显示, 与全光谱相比, GA-IDS选择的主从光谱区间可以显著地降低误差; 与向后迭代区间选择法(IIBS)相比, 在小样本情况下, GA-IDS的误差显著地小于IIBS方法。
As a non-destructive detection method, near-infrared (NIR) spectra have been widely used in food analysis. In NIR analysis, the model between spectra and sample concentrations should be calibrated in advance, and the concentrations of new samples can be predicted by substituting their spectra with the calibrated model. However, the variation of measurement conditions can lead to spectra changes. This problem can be solved by calibration transfer which corrects the new spectra (secondary spectra) to be accurately predicted by the old spectra (primary spectra) model. The calibration transfer always uses full primary and secondary spectra for correction. However, full primary and secondary spectra contain interference, including noise and background, which can increase prediction errors. Hence, variable select is used to selecting the informative regions of NIR for calibration transfer. The commonly used variable selection method always treats primary spectra, and both primary and secondary spectra share the same regions for calibration transfer. However, in practical work, the informative regions of primary and secondary spectra are not the same. Thus, both primary and secondary spectra using the same regions for calibration transfer can increase prediction errors. Moreover, the original spectral ranges of primary and secondary spectra may not be the same, and the secondary spectra can not use the regions selected by primary spectra for calibration transfer. In order to solve this problem, this paper proposed a Genetic algorithm for intervals of double spectra (GA-IDS), which selects informative regions for both primary and secondary spectra simultaneously for calibration transfer. The procedure of GA-IDS includes(1)Randomly generating chromosomes in the population;(2)Analyzing each one of the chromosomes and deleting the error ones; (3)Obtaining the primary and secondary spectra regions and the corresponding Root mean squared error of validation (RMSEV) based on each one of the chromosomes; (4)Executing selection, crossover and mutation operations. After finishing one loop, the GA-IDS goes to step (2) to repeat execute errors correction, RMSEV computation, selection, crossover and mutation operation. After achieving the criterion of the final termination, the spectra regions with minimal RMSEV can be retained. Two datasets, including corn and wheat datasets, were used to evaluate this algorithm. The results show that, compared with full variables, GA-IDS can select good regions for both primary and secondary spectra to reduce prediction errors. Compared with Iterative interval backward selection (IIBS), GA-IDS can achieve lower errors at the small size transfer set.
近红外光谱作为一种无损检测方法, 在食品领域中有大量的应用[1, 2]。 在近红外光谱分析过程中, 我们先要将近红外光谱和浓度信息建立统计模型, 然后将待测样品的光谱数据代入这个模型中, 即可获得其含量信息。 但是, 随着时间的推移, 近红外光谱仪器会发生老化, 进而降低模型的预测能力; 同时, 由于不同型号仪器之间存在响应差异, 一种型号仪器建立的光谱模型, 往往不能直接预测另一种型号仪器扫描的光谱; 此外, 由于基质的差异, 一组样品建立的模型, 往往不适合另一组样品的光谱。 对于此类问题, 可以通过重新建模加以解决, 但是重新建模将耗费大量的人力物力。 对此, 模型转移[3, 4]可以在避免重新建模的情况下, 对光谱进行校正, 提高原模型的预测精度。 在模型转移算法中, 直接校正法(direct standardization, DS)[5, 6]使用简单, 计算速度快, 故本工作使用DS算法作为模型转移算法。
在模型转移中, 人们通常采用全光谱模型转移。 主光谱和从光谱中往往存在背景、 噪声等干扰, 必须要将这些无意义的信息去除; 同时, 挖掘光谱中有化学意义的区间可以提高模型的精度, 故模型转移过程中必须进行变量选择。 变量选择算法广泛用于近红外光谱建模。 按照算法处理光谱的连续性, 可以分为基于离散变量的变量选择算法和基于区间的变量选择算法。 在这两种算法中, 基于区间的变量选择算法较为合理, 因为它能保证光谱的连续性, 且其筛选的光谱区段包含与待测指标有关的化学信息。 此外, 按照算法原理, 可以分为基于智能优化的变量选择算法, 即通过模拟自然的物理、 化学、 生物的过程, 实现变量选择; 基于变量重要性信息的变量选择算法, 即通过构造变量重要性向量, 然后根据该向量找出对建模有重要意义的变量; 基于贪婪算法的变量选择算法。 在这三种算法中, 贪婪算法迭代步骤较多, 且容易导致过拟合, 故应用较少。
以往的变量选择方法往往是只对主光谱进行变量选择[7, 8], 但是主光谱和从光谱的有化学意义的区间往往不一致, 用主光谱变量选择的模型直接预测从光谱会增加预测误差; 有时, 主光谱和从光谱的光谱范围都不一致, 主光谱的变量选择模型无法用于从光谱, 故有必要对主光谱和从光谱同时进行变量选择。
在变量选择算法中, GA[9, 10]可以直接处理二进制变量, 因此广泛应用于变量选择。 此外, 相比单个变量, 区间的光谱波段更能反映化合物的结构信息。 基于此, 提出双光谱区间遗传算法(genetic algorithm for intervals of double spectra, GA-IDS), 该算法同时对主光谱和从光谱进行变量选择, 进而找出有化学意义的区间组合, 进而降低转移误差。
在模型转移中, 用于建模的光谱被称为主光谱(A); 在另一个条件下扫描获得的光谱称为从光谱(B); 从光谱不参与建模, 只用主光谱模型预测。 对于主光谱, 其校正集表示为(Ac), 通过KS方法[11]从Ac中选择一定数量的样本作为主光谱转移集(At), 然后在从光谱中选择与At相同浓度的样本作为从光谱的转移集(Bt)。 接下来用从光谱的验证集(Bv)来调节模型转移参数, 同时计算变量选择中校正均方根误差(root mean squared error of validation, RMSEV), 获得最优变量集。 调节好参数之后, 用模型转移算法预测独立测试集(Bp)的样本。 从光谱的独立测试集则不参与建模和变量选择, 仅计算变量选择结果的预测均方根误差(root mean squared error of prediction, RMSEP)。
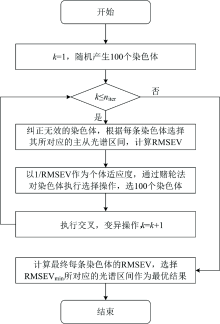
GA-IDS算法流程图如图1。
在图1中, 先对主光谱和从光谱进行区间分割, 然后将每个区间看作一个基因, 如果该基因被选中了, 则该区间所对应的的基因为1, 若未被选中则为0。 然后我们随机生成多条染色体, 获得每条染色体所对应的变量集合。 如果该染色体是错误染色体, 诸如主光谱或者从光谱没有区间被选中, 那么这条错误染色体就被删除, 其位置由重新随机生成的染色体取代。 获得主从光谱的变量选择区间后, 计算其RMSEV。 然后, 根据RMSEV, 按照遗传算法执行选择, 交叉, 变异等操作。 在一次迭代完成之后, 我们进行下一轮迭代, 再次进行纠错, 选择, 交叉, 变异操作。 其遗传算法参数值参数列于表1。
| 表1 GA-IDS的参数 Table 1 Parameters of GA-IDS |
在表1中, 迭代次数(niter)的选择, 通过实验确定。 即在多次迭代后, 当计算结果的最小RMSEV(RMSEVmin)稳定时, 其niter便选为迭代次数。
1.3.1 玉米数据
玉米数据, 玉米数据下载于: https://eigenvector.com/resources/data-sets/, 主光谱和从光谱均有80个样本, 且其波长范围为1 100~2 498 nm(700个波数)。 其中, 取mp6作为主光谱, m5数据作为从光谱, 取水分数据作为y值。 按照y值降序排列, 每4个连续的样本取出第一个样本, 这样剩下的60个样本与其所对应的mp6样本作为校正集。 对于未被选为校正集的20个y值, 按照浓度降序排列, 选择其相应的m5样本。 每两个样本中, 第一个样本和第二个样本分别为验证集和独立测试集。 故验证集和独立测试集的样本数均为10。
1.3.2 小麦数据
小麦的数据取自: http://www.wiley.com/legacy/wileychi/chemometrics/datasets.html, 这套数据有775个样本, 1050个波数点。 该数据由两种探头扫描的光谱拼接而成, 其中400~1 098 nm 的短波近红外由硅探头扫描获得, 而1 100~2 498 nm的长波近红外由硫化铅探头扫描获得。 两种由不同探头获取的光谱也可以看作两组不同光谱, 故我们将长波近红外光谱看作主光谱, 而短波近红外光谱看作从光谱。 选取蛋白质浓度数据作为预测指标。 将主光谱的775个样本随机排序, 前400个样本作为校正集。 对第401— 450号样本以及451— 775号样本, 其相应浓度的从光谱分别作为验证集和独立测试集。 对主光谱的校正集, 选择其KS较大的若干个样本作为主光谱的转移集, 其浓度相同的从光谱样本作为从光谱的转移集。
在遗传算法操作中, 主光谱和从光谱的变量数不一致, 而DS模型转移算法可以处理这种数据结构。 经过搜索, 发现在转移集样本数m=30, 区间宽度n=10的时候, 预测误差较小。 故选择m=30, n=10。 将GA每次迭代中, RMSEVmin的变化列于图2。
图2显示, 当niter< 500的时候, RMSEVmin呈现快速下降趋势, 其原理可能是, 在迭代过程中, 大量的误差较大的变量集合被删除, 而只有RMSEV较小的变量集合被保留下来。 而当niterk≥ 500时, RMSEVmin几乎保持不变。 其原因可能是, 由于被保留下的变量集几乎是相似的, 且变量集的RMSEV几乎一样, 故在选择操作中, 全部个体被保留下来。 在交叉操作中, 种群中相同的样本, 其交叉的结果还是的原来的变量集, 故交叉不能找出误差更小的个体。 虽然变异操作能够改变染色体, 但是变异后的染色体会增大误差, 进而变异后的染色体不会被选中。 故确定niter为500。
在固定了迭代次数之后, GA-IDS获得的变量集, 可用于预测独立测试集, 其结果列在表2中; 同时, 全光谱的模型转移结果, 以及之前提出的向后迭代区间选择法(iterative interval backward selection, IIBS)[12]也被用于比较。
| 表2 玉米数据的GA-IDS算法结果 Table 2 Results of GA-IDS for corn dataset |
从表2可以看出, 与全光谱相比, GA-IDS可以显著地降低RMSEVmin与RMSEP值。 这说明了, GA-IDS可以从近红外光谱中提取出有意义的信息进而降低计算误差。 同时, 与IIBS相比, GA-IDS仍旧可以获得较低的误差值。 其原因是IIBS只是单纯地通过迭代法, 向后删除变量区间, 且被删除的区间无法再被重新选中。 而GA-IDS算法, 可将前被删除的区间通过交叉、 变异操作重新被选中, 故搜索空间大于IIBS算法, 所以能够找到更好的变量集, 进而获得更小的误差。
为了进一步研究GA-IDS的计算结果, GA-IDS选择的变量见图3。
在图3(e, f), 可以看出与全光谱相比, GA-IDS显著地降低了变量数, 且选择的区间, 诸如: 1 450和1 950 nm也与水分的吸收有关[13, 14]。 与IIBS相比, GA-IDS选择了更多有意义的变量。 诸如, 1 950 nm 附近区间是水的吸收峰, 在主光谱中, IIBS没有选择该区间的变量, 而GA-IDS却选中了。 这说明了GA-IDS可以比IIBS选择出更具有物化意义的光谱区间。
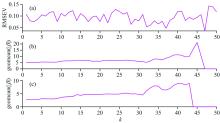
为了进一步揭示IIBS丢失1 950 nm区段水吸收峰的原因, 其主从光谱的全光谱回归系数(|β |)也显示于图3(c, d)。 图3(c)显示, 主光谱诸如1 908和1 950 nm等水吸收峰仍有较高的|β |值, 理应被选中, 但是在迭代过程中, 这些变量却被丢失了。 对此, 我们研究了IIBS迭代过程中主光谱1 884~1 910和1 940~1 966 nm两段波长的回归系数变化情况, 将其列于图4(b, c); 此外, 主光谱每次丢弃变量过程中, 其相应的最小RMSEV也列于图4(a)。
在图4的迭代过程中, RMSEV在第47次迭代中获得最小值, 而在此时1 884~1 910和1 940~1 966 nm区域却已在之前的迭代中被删除; 同时由于IIBS没有采用有放回的策略, 这些被删除的变量不能被重新选中, 故无法进一步降低预测误差。 而GA-IDS在变量选择操作中使用了有放回的策略, 即先前被删除的变量区间, 在后续遗传操作中可以重新被选中, 故GA-IDS可以选择跟多有信息的区间。
为了进一步研究算法的稳健性, 我们随机生成浓度数据不重合的校正集(主光谱)、 验证集(从光谱)和独立测试集(从光谱)。 主光谱转移集通过KS方法从主光谱中选出, 其相同浓度的从光谱作为从光谱的转移集。 按照前述方法, 用验证集进行变量选择, 变量选择最终的结果用独立测试集预测。 这样重复执行100次, 每次选择不同的转移集样本数, 其平均RMSEP数值列于图5。
 | 图5 Monte Carlo抽样下玉米不同m值的RMSEP 红线: 全光谱; 粉红线: IIBS; 蓝线: GA-IDSFig.5 Average RMSEP of Monte Carlo selection for corn dataset red line: full variables; pink line: IIBS; blue line: GA-IDS |
从图5中可见, 在每一个m值条件下, GA-IDS的RMSEP值, 均小于全光谱的RMSEP值。 说明了GA-IDS可以显著地从光谱中选择出重要的信息, 进而降低误差。 与IIBS相比, 当m≤ 35时, GA-IDS可以获得较小的误差, 当m> 35时, GA-IDS的误差和IIBS误差接近。 这说明了, 在较少转移集样本情况下, GA-IDS可以获得较低的预测误差。 其原因可能是, 在较少样本的情况下, 化学信息主要存在于变量中, 而GA-IDS其对变量区间组合的搜索范围要比IIBS大, 进而更容易找到误差较小的变量区间组合。
经过优化, 选择n=20。 随机生成校正集、 验证集、 独立测试集, 对校正集用KS方法从中选择转移集, 在每一个m值条件下, 用验证集结合IIBS和GA-IDS进行变量选择, 最后用变量选择的结果计算独立测试集的RMSEP。 将上述方法重复100次, 计算每一个m值条件下, 全光谱, IIBS和GA-IDS的RMSEP值, 结果如图6所示。
 | 图6 Monte Carlo抽样下小麦不同m值的RMSEP 红线: 全光谱; 粉红线: IIBS; 蓝线: GA-IDSFig.6 Average RMSEP of Monte Carlo selection for wheat dataset red line: full variables; pink line: IIBS; blue line: GA-IDS |
图6结果与玉米数据类似, 对每一个m值, GA-IDS的预测误差均小于全光谱的RMSEP, 说明GA-IDS算法可以从全光谱中选出有意义的区间进而降低误差。 与IIBS相比, m≤ 60, GA-IDS可以获得更小的RMSEP; 而当m> 60时, GA-IDS的RMSEP比IIBS略大。 这进一步说明了, 在小样本转移集下GA-IDS可以获得更好的计算结果。 在模型转移实际应用中, 为了减少工作量, 通常都使用较少的转移集样本, 而GA-IDS恰好适用于这种小样本情形。
GA-IDS通过对主光谱和从光谱同时进行区间选择选择, 可以获得有化学意义的区间, 同时大幅度地降低预测误差。 与全光谱相比, 对不同的转移集样本数, GA-IDS均可以获得较小预测误差; 与IIBS相比, 在转移集样本数较少的条件下, GA-IDS可以获得较小的预测误差。 故GA-IDS有较强的应用推广价值, 特别是小样本转移集的模型转移。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|