作者简介: 袁 壮, 1997年生, 广西大学硕士研究生 e-mail: 17746612550@163.com
近红外光谱是热门的食品检测方法之一, 对于这种高维光谱数据的分析常常需采用数据降维算法提取其中的特征, 然而绝大多数算法都只能针对单个数据集进行分析。 虽然已有基于对比学习的对比主成分分析成功应用于不同水果表面农残的近红外光谱检测中, 但是该方法只能以线性的方式组合原有特征, 特征提取效果存在局限性, 并且需要调节对比参数来控制背景集影响, 需要消耗更大的时间成本。 cVAE(contrastive variational autoencoder)是一种基于对比学习和变分自编码器的改进算法, 被用于图像去噪和RNA序列分析中, 它仍然具备分析多个数据集的特点, 同时因为组合了神经网络的概率生成模型而具备了提取非线性隐含特征的能力。 将cVAE算法应用于近红外光谱分析, 建立了准确的近红外光谱数据降维模型。 在实际验证中, 使用cVAE算法对购买的不同品牌和批次纯牛奶中掺假三聚氰胺进行检测。 结果表明, 使用VAE算法只能区分出不同品牌和批次的纯牛奶, 而其中是否掺假三聚氰胺这一重要信息无法表现出来; 而使用cVAE算法进行数据分析时, 由于添加了背景数据集分离了无关变量, 能够清晰的将有无掺假三聚氰胺的样本分类。 这说明了, cVAE不仅具备了cPCA(contrastive principle component analysis)在近红外光谱数据降维中的优势, 而且具备提取非线性特征的能力, 同时不需要调节可变参数, 能够更方便地建立近红外光谱降维模型。
Near-infrared spectroscopy is one of the popular food detection methods. Analyzing such high-dimensional spectral data often requires using data dimensionality reduction algorithms to extract features. However, most algorithms can only analyze a single data set.Although the contrastive principal component analysis based on contrastive learning has been successfully applied to the near-infrared spectroscopy detection of pesticide residues on the surface of different fruits, this method can only linearly combine the original features, and the feature extraction effect has limitations and needs to be adjusted. Comparing parameters to control the influence of the background set requires more time and cost.cVAE (contrastive variational autoencoder) is an improved algorithm based on contrastive learning and variational autoencoder, which is used in image denoising and RNA sequence analysis. It still has the characteristics of analyzing multiple data sets, and at the same time, it can extract nonlinear hidden features because of the combination of the probability generation model of the neural network. The cVAE algorithm is applied to near-infrared spectroscopy analysis, and an accurate dimensionality reduction model of near-infrared spectroscopy data is established. In actual verification, the cVAE algorithm was used to detect melamine adulteration in pure milk purchased by different brands and batches. The results show that when analyzing whether melamine is adulterated in pure milk purchased by different brands and batches, the VAE algorithm can only distinguish between different brands and batches of pure milk, and the important information about whether or not melamine is adulterated is not available. When using the cVAE algorithm for data analysis, because the background data set is added to separate irrelevant variables, it is possible to classify samples with or without adulterated melamine clearly.This shows that cVAE has the advantages of cPCA (contrastive principle component analysis) in dimensionality reduction of near-infrared spectroscopy data and can extract nonlinear features without the need to adjust variable parameters.The dimensionality reduction model of the near-infrared spectroscopy can be established more conveniently.
近红外光谱是发展快、 具有广阔前途的成分分析技术之一, 以其方便、 快速、 高效、 准确、 无污染和在线预测的优点, 一直处于食品分析的前沿[1, 2]。 由于近红外光谱反应的主要是分子中官能团振动的倍频和合频的吸收, 光谱特征不明显, 特征之间存在多重共线性, 对近红外光谱进行分析往往需借助化学计量学软件和机器学习算法的辅助。 数据降维和特征提取是分析高维数据的有效手段, 利用降维结果还可以进行可视化分析。 常见的数据降维算法有: 奇异值分解(SVD)[3]、 主成分分析(PCA)[4]、 线性判别分析(LDA)[5]、 因子分析(FA)[6]、 独立成分分析(ICA)[7]。 然而, 这些算法都被设计为一次只能分析一个数据集, 当数据集个数较多或者一个数据集中有多个不同的背景因素影响时, 它们只能单独作用然后需要手动分析比较数据集之间特征的相同点和不同点[3]。 cPCA是PCA的对比学习变体, 它能够将需要分析的数据集(目标集)与干扰因素(背景集)进行比较、 学习到两个集合的差异(研究人员想要的特征信息), 已经在不同水果的农药残留判别中得到优良的效果[8, 9]。 主成分只能通过线性的方式组合生成新特征, 特征提取效果存在局限性, 而在一些复杂条件下, 非线性的新特征可能在下游模型中取得更好的效果, 并且cPCA基于数据集方差最大化, 在对背景集进行约束时需要合理选择对比参数, 实际使用时存在不确定性。
普通的自编码器在理论上可以在低维度空间中完全拟合原始数据, 但是不具备任何生成能力, 对新样本无法进行适应[10, 11]。 变分自编码器通过对模型进行概率化, 添加正则化项对低维隐变量Z的编码空间进行约束, 使得模型既能一定程度上拟合原始样本, 又具备了可靠的生成能力[12]。 Z被称为隐变量, 因为它是由模型编码器部分给出的低维编码, 我们不一定知道编码中的哪些具体设置促进生成了输出。 对比变分自编码器是变分自编码器的多输入型的变体, 它主要利用了变分自编码器能够提取隐含空间特征信息的能力和神经网络多输入和多输出的特点, 同时对两个不同的数据集进行拟合。 cVAE在手写数字图片受背景干扰的情况下进行数字识别、 受不同检测批次影响的骨髓移植前后RNA信息判别分析, 以及面部图像特征提取中得到了比较好的改进效果。 我们将cVAE用于受不同品牌和批次影响的纯牛奶中是否掺假三聚氰胺的检测, 实现了非线性对比学习算法结合近红外光谱在液态食品样品中的首次应用。
三元、 伊利两种品牌的全脂纯牛奶, 国标号为GB25190, 前后两个日期购置于北京市海淀区某超市(日期新鲜、 包装完好、 国内大品牌)。 纯牛奶买回后置于常温环境中, 由于其中脂肪球和蛋白质颗粒随环境变化和时间推移发生不同程度的聚集, 造成成分不均匀。 在制备样本前使用超声振荡20 min, 使其中成分尽可能均匀分布。 掺假牛奶的制备过程为: 往试剂瓶中称取90 mg的三聚氰胺粉末(小于常温下三聚氰胺在水中溶解度0.33 g, 也符合掺假厂商实际牟利目的), 随后向其中添加30 mL纯牛奶, 将所有样本充分摇匀后静置, 等待光谱采集, 上述操作与采集过程同步且连贯。 每次制备的样本分为目标数据集与背景数据集。 考虑牛奶成分的非均匀性和光谱采集方式的限制, 每个样本采集10条光谱。 重复前后五次, 各个类型的样本及数目如表1所示。
| 表1 数据集中样本的组成 Table 1 The composition of the samples in the data set |
选用傅里叶变换型近红外光谱仪(美国赛默飞, Antaris Ⅱ )采集纯牛奶光谱, 采集方式为积分球漫反射, 分辨率为16 cm-1, 采集次数设置为64次, 每次采集扣除内置背景, 采集范围为4 000~10 000 cm-1。 光谱分析使用Unscrambler X10.4(CAMO)。 使用基于python3.6.8的Jupyter Notebook构建对比学习分类模型, 神经网络框架基于Tensorflow 2.0。
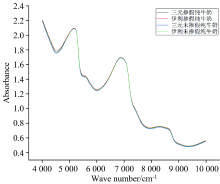
将上述数据集中的样本使用FT-NIR仪器进行扫描, 为了使观察更加清楚, 随机选取目标数据集中四种不同样本中的一条原始光谱图, 如图1所示。 从图中可以看出, 4 700~5 300, 5 400~5 600和6 200~7 200 cm-1这三个波数段的吸光度值明显大于其他波数段, 反映了纯牛奶中主要成分的光谱信息(水、 蛋白质、 脂肪等)。 四类牛奶的光谱从直观看来并无明显差异, 原始光谱曲线平滑基本无噪声, 故不进行预处理直接使用原始光谱建模。
考虑到纯牛奶成分的非均匀性, 和本实验所用均质方法的实际效果, 为了削弱不均匀性对近红外光谱重复性的影响, 我们选择接近有关文献中确定的牛奶中掺杂三聚氰胺等不同目标物的特征分类区间4 200~4 800 cm-1, 增大目标物对光谱的影响[13]。 同时为了尽可能多的保留变量个数, 经过选择后, 波段为4 000~6 000 cm-1, 共有259个波长, 随机选取四个不同类样本的一条光谱, 其间谱线如图2所示。
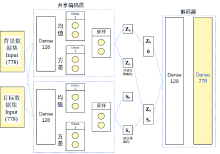
将选择后的数据代入cVAE模型进行分析, 多输入神经网络结构如图3所示。 隐藏层采用128个神经元的全连接层, 使用ReLU激活函数。 其中S为我们感兴趣的编码(也称为隐变量或特征信息), Z为不感兴趣的编码。 x为目标数据集, b为背景数据集。
Fig.3 cVAE neural network structure
神经网络的具体训练过程如下:
(1)对目标数据集和背景数据集光谱数据进行最大值归一化;
(2)设置batch大小为100个的单批次数据大小;
(3)背景数据集自动根据目标数据集大小进行倍数复制;
(4)定义神经网络损失函数, 优化器;
(5)将目标数据集、 背景数据集同时放入神经网络进行训练;
(6)根据loss下降情况和评价指标分析分类效果。
对比变分自编码器的loss函数总共包含以下几项:
(1)重构损失: 包含两项, 分别为目标数据集x和背景数据集b的重构损失;
(2)正则化项: 包含三项, 分别是目标数据集隐变量Zx和Sx, 背景数据集中隐变量Zb, 他们分别对提取的隐变量空间进行正则化约束;
(3)全局相关损失: 为了增加隐变量S和Z之间的不相关性添加的损失;
(4)判别器损失: 为了方便计算全局相关损失, 另外构建了单独的判别器, 利用分类概率估计该项。 该判别器与cVAE的编码器和解码器同时训练, 因此需要添加进总的损失函数。
实验的主要目的是为了在不同品牌和批次生产的纯牛奶中区分出掺假三聚氰胺和未掺假三聚氰胺的纯牛奶。 将200个掺假的三元牌纯牛奶、 未掺假的三元牌纯牛奶、 掺假的伊利牌纯牛奶、 未掺假的伊利牌纯牛奶混合后输入模型, 画出二维隐空间中的散点图, 如图4所示。
从图4中可以发现, VAE学习到的特征信息主要包含了纯牛奶的不同品牌和批次的信息, 这两者都会使得牛奶中成分信息不同。 对于是否掺假三聚氰胺这一重要信息, VAE无法提取。
造成VAE无法对掺假牛奶进行判别的主要原因是数据集中包含了纯牛奶品牌及批次这两个重要变量的信息, 导致不相关信息占据了数据变化的绝大部分。
我们考虑利用cVAE提取感兴趣的数据变化, 将目标数据集200个样本混合形成验证集输入经过训练的模型, 画出二维隐变量空间的数据散点图, 如图5所示。 从图5中可以发现, 蓝色和红色点聚成一类(分别代表三元掺假牛奶、 伊利掺假牛奶), 绿色和黄色点聚成另一类(分别代表三元未掺假牛奶、 伊利未掺假牛奶), 表明此时特征信息主要反应了纯牛奶中有无掺假三聚氰胺, 品牌和批次差异的无关背景信号被消除。 分类边界存在少量不明确的样本点, 这可能与神经网络的随机初始化参数有关, 需进一步优化模型。
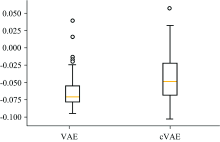
为了进一步验证cVAE同时改善了聚类的效果, 我们使用轮廓系数(sihouette score)去定量评估降维结果与真实标签更接近的聚集程度。 轮廓系数的值介于[-1, 1], 越接近1代表内聚度和分离度都相对较优, 最后将所有数据点的轮廓系数求平均, 就是该聚类效果总的轮廓系数。 重复训练100次, VAE与cVAE的相应轮廓系数的比较如图6所示, 可以发现cVAE的轮廓系数相比VAE有了明显提升, 表明cVAE提取特征后对样本的聚类效果要优于VAE。
cVAE利用神经网络的非线性特点, 同时组合概率生成模型和对比学习的优点, 是一种能够通过引入背景数据集, 来提取分析多个数据集中感兴趣信息的新型数据降维方法。 在对受品牌和批次干扰的纯牛奶中是否掺假三聚氰胺进行分析时, 使用VAE算法只能区分出不同品牌和批次的纯牛奶这一背景信息, 而使用cVAE算法能够将纯牛奶中是否掺假三聚氰胺这一关键信息提取出来, 说明cVAE能够提取非线性的对比特征。 并通过聚类评估指标对二者聚类效果进行评价, 结果表明cVAE提取到的低维空间信息对样本的聚类效果优于VAE。 神经网络的可扩展性和对海量数据的依赖性使得这一算法有更大的进步空间, 我们可以通过增加神经元数量和层数增大网络容量, 通过增加更多的新数据使得模型的泛化性能更优, 提取到的关键特征可以用于下游模型的建立, 该算法在近红外光谱分析中有着广阔的应用前景。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|